
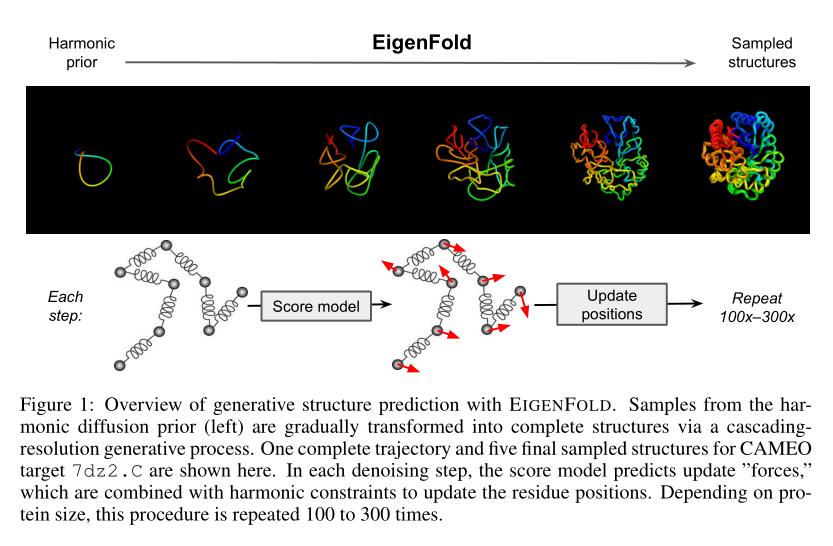
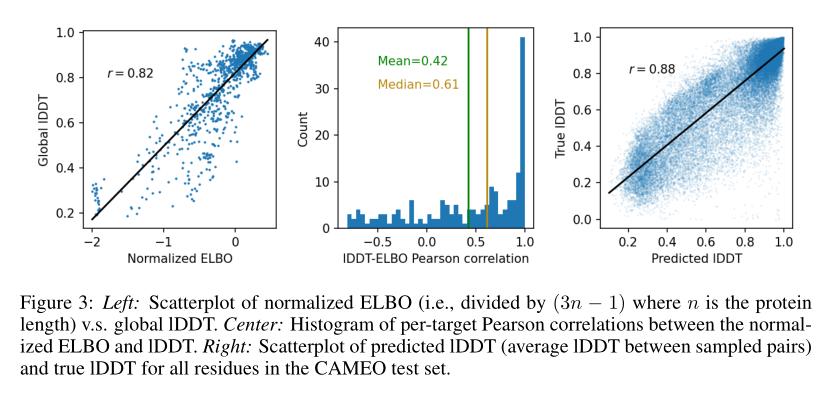
蛋白质结构预测在单个结构上已经达到了革命性的准确性,但为了捕捉生物功能所依赖的构象集合和灵活性,需要分布式建模方法。为此,本文开发了EigenFold,一种扩散生成建模框架,用于从给定的蛋白质序列中采样结构分布。本文定义了一个扩散过程,将结构建模为谐振子系统,并自然地沿系统的特征模式引出一个级联分辨率生成过程。在最近的CAMEO数据集上,EigenFold的中位TMScore为0.84,同时相对于现有方法提供了更全面的模型不确定性图像。然后本文评估了EigenFold在建模和预测折叠切换蛋白质和配体诱导构象变化方面的能力。
总结:
本文介绍了一种新的生成模型EigenFold,用于预测蛋白质的三维结构。EigenFold结合了数学中的扩散过程和图神经网络的技术,可以高效地将一维序列转化为三维结构。与传统的基于模拟和能量函数优化的方法相比,EigenFold的生成速度更快、精度更高。实验结果表明,EigenFold可以成功地预测出大部分蛋白质的结构。
标题:EigenFold: Generative Protein Structure Prediction with Diffusion Models
作者:Bowen Jing, Ezra Erives, Peter Pao-Huang, Gabriele Corso, Bonnie Berger, Tommi Jaakkola
代码:https://github.com/bjing2016/EigenFold



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


