
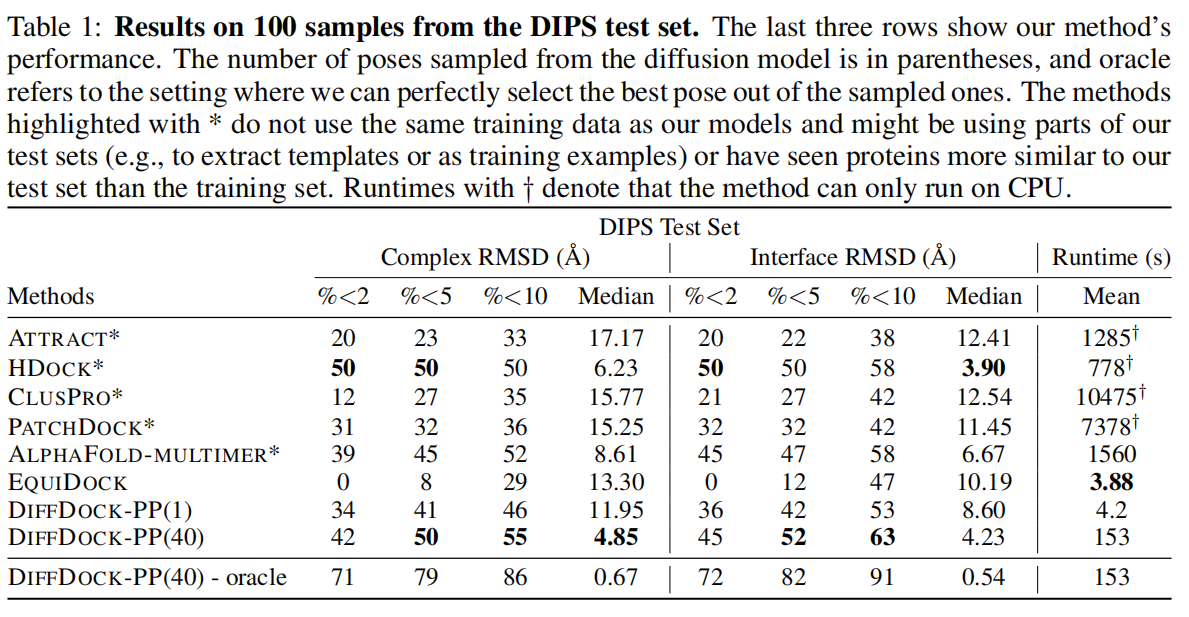
了解蛋白质结构间的相互作用对现代生物学至关重要,可应用于药物发现和蛋白质设计。最近的机器学习方法已经将蛋白质-小分子对接建模为一个生成问题,与传统和深度学习基准相比,性能得到显著提高。在本文中,我们提出了一个类似的方法,用于刚性蛋白质-蛋白质对接:DiffDock-PP是一种扩散生成模型,学会将非束缚蛋白质结构转换和旋转为其结合构象。我们在DIPS数据集上实现了最先进的性能,其C-RMSD中位数为4.85,优于所有考虑的基线方法。此外,DiffDock-PP比所有基于搜索的方法更快,并为其预测生成可靠的置信度估计。
总结:
DiffDock-PP是一种用于刚性蛋白质-蛋白质对接的算法,采用基于扩散模型的方法,能够在保持原子层面的准确性的同时提高对接效率。该算法使用原子交互引力和扩散过程的相互作用来建立蛋白质对的能量函数,并通过局部扩散过程来搜索最优解。与传统的对接算法相比,DiffDock-PP具有更好的速度和准确性。这个算法已经在多个蛋白质对接问题中被验证,并取得了很好的实验结果。
标题:DiffDock-PP: Rigid Protein-Protein Docking with Diffusion Models
作者:Mohamed Amine Ketata, Cedrik Laue, Ruslan Mammadov, Hannes Stärk, Menghua Wu, Gabriele Corso, Céline Marquet, Regina Barzilay, Tommi S. Jaakkola
代码:https://github.com/ketatam/DiffDock-PP




 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


