编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自James J. Collins和Jonathan M. Stokes团队的一篇关于抗生素发现的论文。鲍曼不动杆菌(Acinetobacter baumannii)是一种常表现出多重药物抗性的医院内革兰氏阴性病原体。通过传统的筛选方法发现针对鲍曼不动杆菌的新抗生素一直具有挑战性。幸运的是,机器学习方法可以快速探索化学空间,增加了发现新抗菌分子的可能性。在这项研究中,作者筛选了大约7500种分子,以找出能够体外抑制鲍曼不动杆菌生长的分子。

鲍曼不动杆菌(Acinetobacter baumannii)是一种医院内的革兰氏阴性病原体,通常由于其坚韧的外膜以及获取和保留经常编码抗生素耐药基因的细胞外DNA而表现出多重药物抗性。此外,它可以在表面上存活很长时间,并且对干燥有抵抗力。通过传统的筛选方法,发现针对鲍曼不动杆菌的全新抗生素一直具有挑战性。实际上,大多数达到临床应用的新抗生素都是现有类别的类似物。然而,虽然现有抗生素类别的结构类似物可能满足短期的临床需求,但由于现有耐药决定因子的高度普遍性,它们的长期疗效本质上是有限的。理想情况下,新抗生素的发现应该专注于识别具有相对于现有抗生素独特机制的新化学类型。这些化合物可能会有长时间的效用,因为预先存在的临床耐药性的概率很低。幸运的是,机器学习方法可以在计算机模拟中快速探索广泛的化学/序列空间,从而增加了发现具有抗菌活性的理想新化学类型的概率,特别是针对像鲍曼不动杆菌这样具有挑战性的病原体。
除了简单地发现结构和功能上的新抗生素,还存在一个很大的未满足需求,即应用于针对特定细菌物种的窄谱治疗。这种抗生素之所以有益,有以下两个原因:首先,相对于常规的广谱药物,针对窄谱药物产生耐药性的速度可能较低,因为窄谱药物不会施加一种普遍的选择性压力,从而有利于广泛传播耐药性基因;其次,窄谱抗生素在治疗期间不会破坏微生态的生态平衡。实际上,失调微生物群落与一系列不良健康结果相关。例如,Clostridioides difficile感染就是由于抗生素引起的微生态失调而导致的机会性感染的典型例子,仅在2017年就在美国的住院患者中导致了超过224,000例感染和13,000例死亡。
机器学习引导的abaucin的发现
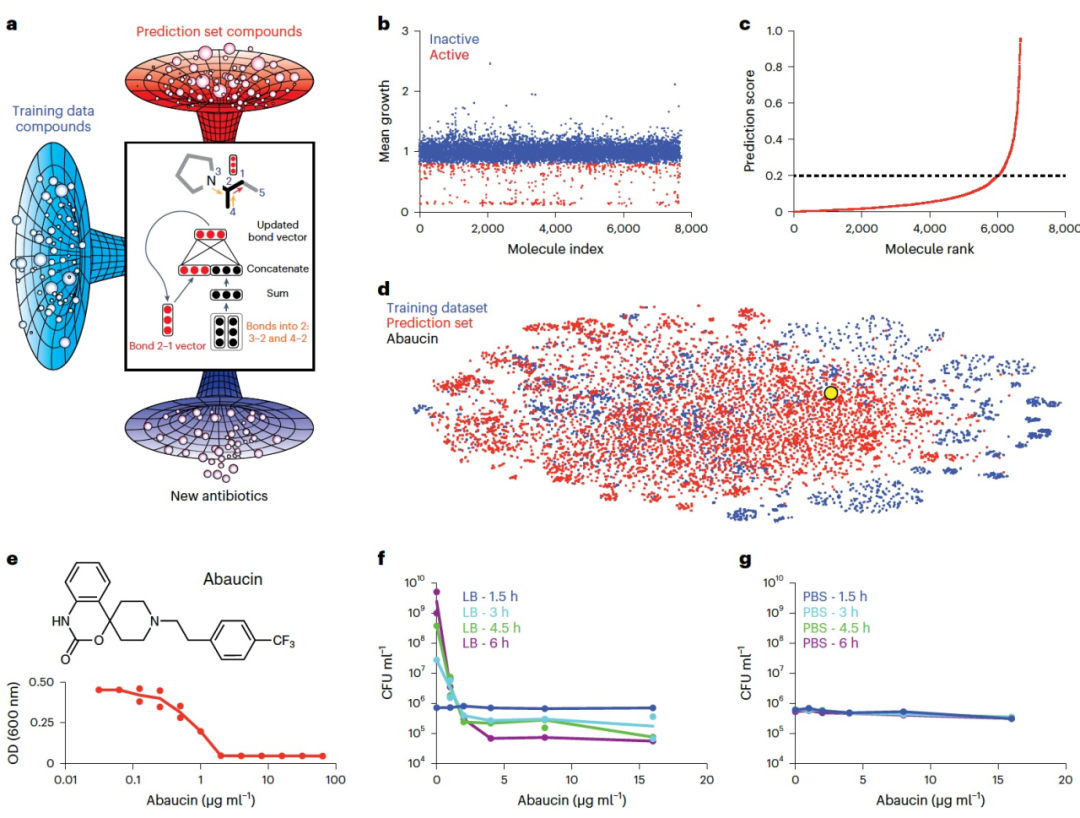
图 1
作者最近的研究突显了机器学习在使用大肠杆菌K12作为模型生物体进行新抗菌分子发现方面的实用性。在前期研究的基础上,在此将信息传递深度神经网络应用于发现针对阿克拉博芒不动杆菌的新抗生素,该致命的医院内感染的革兰阴性病原体通常表现出多药耐药性,甚至越来越多地表现出全药耐药性(图1a)。作者首先在Lysogeny Broth(LB)培养基中以50μM浓度筛选了一组7,684个小分子,以确定其中抑制阿克拉博芒不动杆菌ATCC 17978生长的分子(图1b和)。这个化合物集合包含了已过专利期的药物(2,341个分子)和从Broad研究所的各种高通量筛选子库中筛选出的合成化学品(5,343个分子)。使用传统的活性阈值将480个分子定义为“活性”,7204个分子定义为“非活性”。随后,作者使用这个数据集来训练一个二分类器,以预测结构上新的分子是否可能对阿克拉博芒不动杆菌具有活性。这种类型的模型通过一系列“信息传递”步骤,迭代地在相邻的原子和键之间交换局部化学信息。每次信息传递迭代将有关局部化学的信息传播到整个分子,从而使模型能够建立分子的更全面的表示。在进行了一定次数的信息传递步骤之后,分子的各个局部化学区域的向量表示被求和为一个捕捉整个化合物复杂性的单一连续向量。然后,使用RDKit19计算得到的固定分子特征来补充这个学习到的最终向量。这个包含了学习特征和计算特征的最终向量被用作前馈神经网络的输入向量,用于预测抗菌性能。作者使用十个分类器的集成方法对模型进行进一步优化,增强了其稳健性。最终模型在精确-召回曲线下的面积为0.337 ± 0.088,在接收者操作特征曲线下的面积为0.792 ± 0.042,为在新的化学空间中进行预测提供了信心。
在模型训练之后,作者将模型的集合应用于从更新后的Drug Repurposing Hub中筛选出对阿克拉博芒不动杆菌具有活性的抗菌分子,该化合物库包含了6,680个分子。由于该集合内部的结构多样性以及许多药物再利用中心分子具有良好的细胞毒性和药物样性能,因此选择了这个化合物库作为概念验证。十个分类器的集合为每个化合物返回了一个预测得分,表示对阿克拉博芒不动杆菌生长抑制的概率(图1c和)。那些在预测中被强烈预测为抗菌活性并且与训练集“活性”分子在结构上独特的分子被优先用于体外测试。值得注意的是,这个进行预测和优先选择分子进行验证的过程在几个小时内完成。省略RDKit特征的模型性能降低突显了这些可计算的分子特征在最大化在我们的训练数据集的情况下的预测效用方面的重要性。使用一个预测得分阈值 >0.2(图1c),然后根据与在训练数据集中的“活性”分子的Tanimoto最近邻相似度 <0.3来过滤这些预测的化合物,作者鉴定出了240个符合这些严格标准的优先级分子。这些分子以50μM浓度在LB培养基中对阿克拉博芒不动杆菌进行了测试,与获取训练数据时的条件相同。使用另一个严格的>80%生长抑制阈值,作者观察到测试的分子中有九个显示出对阿克拉博芒不动杆菌的抗菌活性。随后,对这九种优先分子进行了评估,以排除以下情况:(1)那些在已知抗生素中观察到的主要结构特征;(2)那些在科学或专利文献中报告了抗菌活性;以及(3)那些具有可能的非特异性膜活性,如存在脂环状脂肪基团。基于结构的筛选结果保留了两种分子:RS102895和serdemetan。RS102895是一种经过深入研究的CCR2选择性趋化因子受体拮抗剂,对A. baumannii ATCC 17978显示出最小抑制浓度(MIC)约为2 μg ml-1(图1d,e);serdemetan是一种转录因子HDM2的拮抗剂,显示出MIC约为32 μg ml-1。由于RS102895在抑制A. baumannii的生长方面表现出更强的效力。
在进一步的实验中,作者对RS102895进行了评估,以评估处理后的A. baumannii的存活情况。我们观察到在LB培养基中,RS102895对A. baumannii具有适度的细菌杀菌活性(图1f)。实际上,在对A. baumannii进行6小时的处理后,将RS102895从培养物中去除后,观察到A. baumannii重新生长,其中明显的滞后期随着abaucin浓度的增加而增加。作者在无养分的PBS中观察不到明显的活性(图1g)。综合而言,这些数据表明,以abaucin重命名的RS102895通过抑制在生长和分裂过程中最大活跃的生物过程,显示出其抗细菌效力,与大多数已知抗生素的作用一致。值得注意的是,这些数据还表明,abaucin并不通过破坏磷脂双层的物理方式来活跃于细胞膜,这是在预测筛选过程中有意避免的一种作用机制。实际上,具有膜活性的分子通常在无养分条件下保持细菌杀菌效力。
Abaucin具有狭窄的活性范围
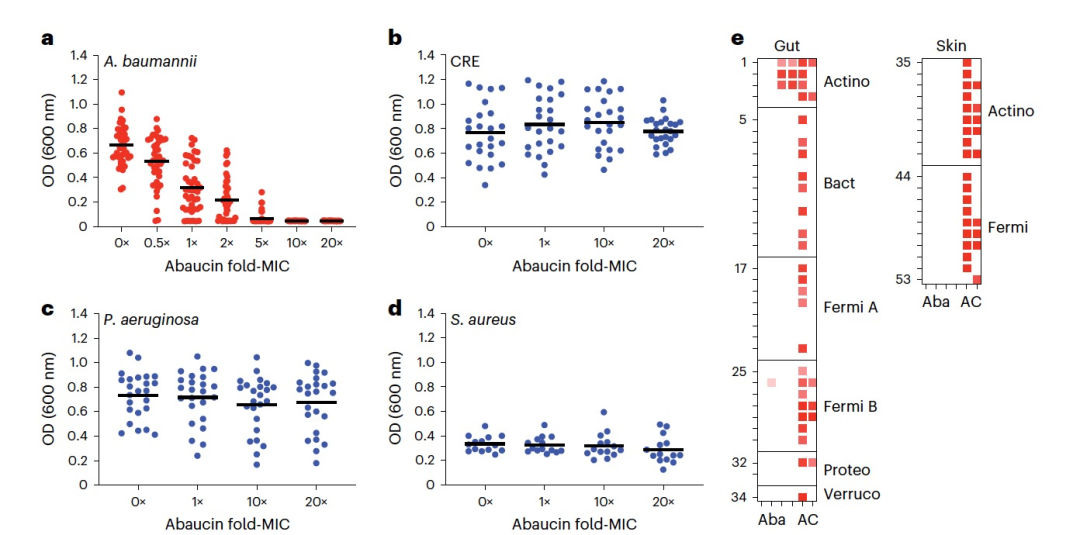
图 2
在评估abaucin对A. baumannii ATCC 17978的活性后,作者接下来测试了其对临床分离的A. baumannii的生长抑制活性。作者从疾病控制和预防中心的抗生素耐药分离物库(ARIsolate Bank)获取了41株A. baumannii,并在多种低于和高于MIC的浓度下测试了abaucin。值得注意的是,观察到abaucin能够克服这个多样临床菌株库中A. baumannii分离物内在和获得性耐药机制(图2a)。
接下来通过对24株耐碳青霉烯的肠杆菌科菌株、24株铜绿假单胞菌菌株和14株金黄色葡萄球菌菌株进行测试,来研究abaucin所显示的抗菌活性的系统进化谱。这些菌株都来自ARIsolate Bank。与对A. baumannii的观察形成鲜明对比的是,abaucin在高达A. baumannii ATCC 17978 MIC 20倍的条件下,对这些致病物种并没有显示出任何生长抑制活性(图2b–d)。实际上,目前已使用的抗生素在治疗过程中常常引发失调,导致一系列并发症,包括由机会性病原体引起的继发感染,其中C. difficile肠道感染是一个常见的例子。为了测试这一假设,作者精选了34株多样的人体肠共生分离物和19株多样的人体皮肤共生分离物,然后测试了abaucin在不同浓度下的生长抑制性能,以及ampicillin和ciprofloxacin在A. baumannii ATCC 17978的MIC浓度下的性能。正如预期的那样,ampicillin和ciprofloxacin在各种共生分离物上显示出抗菌活性,而abaucin在很大程度上避免了对共生种的生长抑制,甚至高达MIC的20倍(图2e)。实际上,在测试的53个分离物中,abaucin只在Bifidobacterium breve和Bifidobacterium longum这两种肠道分离物中显示出真正的生长抑制,而且这种情况发生在高于A. baumannii ATCC 17978中观察到的MIC水平。
结论
A. baumannii急需全新的结构和功能的抗生素,由于其能够吸收和保留抗生素耐药性决定因子,根除它变得非常困难。此外,选择性抗生素有望限制耐药性决定因子的水平传播,降低治疗期间失调的可能性。作者通过机器学习引导发现abaucin突显了算法方法在发现针对A. baumannii的新抗菌分子方面的效用,为该领域提供了一个有望的新的狭谱分子框架,以解决世界上最具挑战性的革兰氏阴性病原体之一。
参考资料
Liu, G., Catacutan, D.B., Rathod, K. et al. Deep learning-guided discovery of an antibiotic targeting Acinetobacter baumannii. Nat Chem Biol (2023).
https://doi.org/10.1038/s41589-023-01349-8



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


