本文介绍一篇由浙江大学侯廷军教授、谢昌谕教授和之江实验室陈广勇研究员团队联合发表的关于分子生成方法的论文。受凝聚态物质和统计物理学多尺度建模研究的启发,作者提出了一种以蛋白质口袋为条件的三维分子生成模型ResGen。该模型采用并行多尺度建模策略,可以捕捉到蛋白靶点与配体间更高层次的相互作用,并实现更好的计算效率。与目前最优(SOTA)方法相比,ResGen生成的分子具有更合理的化学结构,并拥有更好的靶点亲和能力。

研究背景
分子生成是合理药物设计领域的热点研究方向,高质量的分子生成模型可以有效提升先导化合物发现的效率。然而迄今为止,大部分已报道的分子生成工作都采用了基于配体的分子生成(LBMG)策略,即学习活性分子所在的”化学空间”,然后对其进行插值,从而产生新的分子结构。这些基于配体的分子生成模型能够生成大量结构新颖的化合物,但仍有方法学上的固有局限性,例如, LBMG无法有效考虑生成分子与靶标之间的相互作用模式。为了克服LBMG策略的缺陷,研究者们越来越关注基于结构的分子生成(SBMG)方法,即基于靶标结构进行相应的分子生成。
在早期的研究中,基于靶标结构的分子生成方法并不明确模拟生成的分子与目标口袋之间的相互作用。而最近提出的算法,例如 LIGAN、GraphBP和 SBDD,则可以直接根据配体与靶标之间的相互作用产生新的小分子结构。这些方法虽然能够生成结构新颖的化合物,但这些化合物在化学合理性以及靶点亲和力方面仍然不能令人满意。在这些研究的基础上,以Pocket2Mol为代表的更新一代SBMG策略则可以有效生成与靶标结合更加紧密的分子。但本文的研究表明,这些已报道的SBMG 策略仍然倾向于生成随机分子,而不是具有活性的苗头化合物。这说明尽管目前的SBMG方法研究已经取得了相当多令人鼓舞的进步,仍有许多技术障碍有待研究者们克服。
模型框架
ResGen将以蛋白质口袋为条件的三维分子生成问题表述为两个尺度的自回归问题,即全局尺度和原子组件尺度。具体来说,全局尺度的自回归是指:
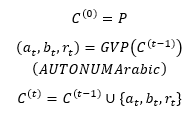
其中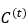 表示在生成过程的第t步时的蛋白-小分子相互作用信息,当t=0时只包含蛋白质信息。原子组件尺度则包括
表示在生成过程的第t步时的蛋白-小分子相互作用信息,当t=0时只包含蛋白质信息。原子组件尺度则包括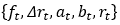 ,其中
,其中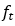 是焦点原子,
是焦点原子,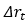 是下一个原子和焦点原子的相对坐标矢量差,
是下一个原子和焦点原子的相对坐标矢量差,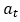 是原子类型,
是原子类型,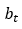 是成键关系。全局尺度自回归明确地表示了分子生成是如何以特定蛋白质口袋为条件的。与此同时,原子组件尺度的自动回归使 ResGen 能够直接生成三维分子。此外,全局尺度的回归意味着ResGen所生成的每个原子都基于之前步骤中生成的分子片段和蛋白质口袋结构;而原子组件尺度自动回归是指每次生成的下一个原子都会根据其组分进行分解。于是,生成分子的每一步都等同于对如下分布进行采样:
是成键关系。全局尺度自回归明确地表示了分子生成是如何以特定蛋白质口袋为条件的。与此同时,原子组件尺度的自动回归使 ResGen 能够直接生成三维分子。此外,全局尺度的回归意味着ResGen所生成的每个原子都基于之前步骤中生成的分子片段和蛋白质口袋结构;而原子组件尺度自动回归是指每次生成的下一个原子都会根据其组分进行分解。于是,生成分子的每一步都等同于对如下分布进行采样:
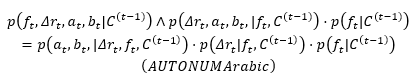
因此ResGen可以将完整的分子生成过程分解为分步采样,从而以自回归式方式实现整个分子的生成。值得注意的是,ResGen利用粗粒化将蛋白表征为残基图,这使得蛋白与小分子图区分度更高,从而有利于模型分别学习蛋白内部、小分子内部以及蛋白-小分子间的相互作用信息。该策略不仅降低了模型对于显存的需求,同时还有利于增强模型对给定口袋的感知能力。更多细节请参照原文。
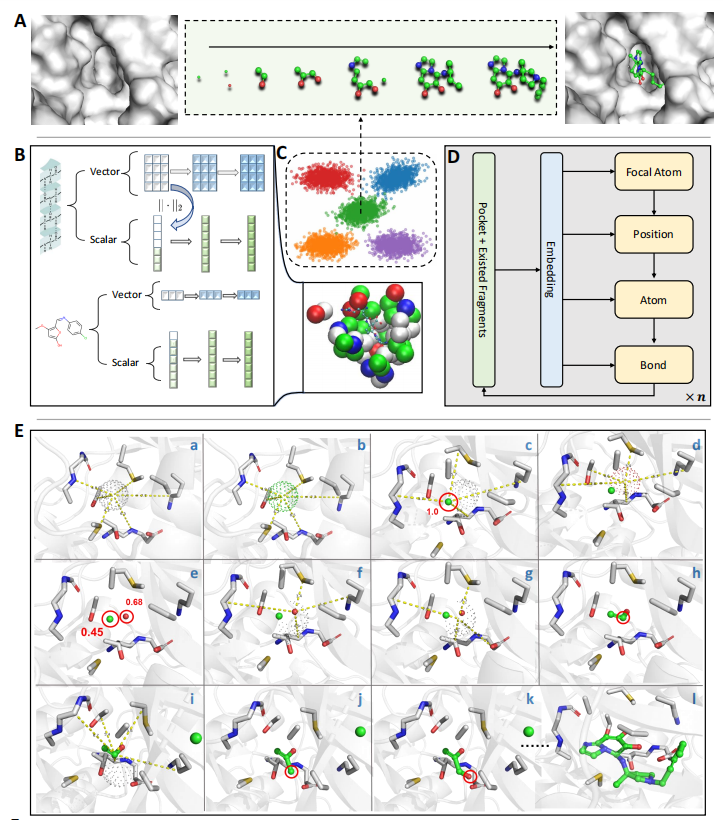
图1:ResGen算法框架示意图。
结果与讨论
口袋感知三维分子生成模型有两个被广为接受的检验指标:(1)模型是否学习了配体在不同蛋白质口袋中的特征拓扑分布,即分子图的目标依赖性;(2)模型是否学习了口袋内配体的几何分布,即原子位置和构象的合理性。针对这两个检验指标,作者对ResGen和现有的 SOTA 模型进行了一系列评估。对于第一条检验指标,作者评估了针对测试集中的靶点和现实世界中的治疗靶点设计生成的分子的结合能和类药性。此外,作者还计算了生成的分子与训练/测试集/治疗靶点上的活性分子之间的分子相似性,从相似性的指标来评估生成分子的质量。对于第二条检验指标,作者设计了构象合理性实验,并分析了蛋白与小分子之间的相互作用模式。在构象合理性实验中,作者计算了直接生成的分子构象和传统构像生成软件产生的分子构象之间的均方根偏差(RMSDs),并且比较了生成样本与训练分子之间的键长分布。此外,为了分析相互作用模式,作者展示了几个蛋白-配体的相互作用谱,以验证ResGen是否成功学习了这些依赖靶点几何结构的相互作用模式。
在测试集上的分子生成
表1:CrossDock测试集上的Top5分子性质
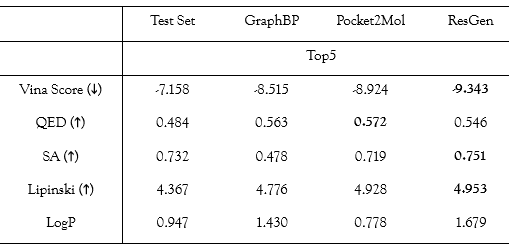
首先,作者使用CrossDock数据集的测试集部分评估了模型的泛化能力,部分结果如表1所示。总体而言,ResGen生成的分子在包括结合能和药物相似性在内的大部分指标上都优于GraphBP和Pocket2Mol生成的分子。其中Vina Score代表了生成分子和对应蛋白靶标的结合能,这一指标能够在一定程度上反映模型是否感知到了口袋内的化学环境。如表 1 所示,ResGen 在Vina Score上的表现意味着 ResGen 比其他两个 SOTA 模型更有机会生成和靶标结合更紧密的分子,作者认为这可以归功于ResGen的多尺度建模表征结构,因为这种结构更有利于捕捉蛋白质口袋和配体间更高层次的相互作用(如片段-残基相互作用)。在实践中,能否将一个有机化合物推进为候选药物不仅取决于其与蛋白质相互作用的强度,还取决于它的类药性和可合成性。因此,作者还评估了生成分子的一系列类药性指标,如 QED、SA、Lipinski 5规则以及LogP,这些指标一定程度上反映了生成分子的类药性。如表 1 所示,ResGen 在 SA 和 Lipinski 指标上得分最高,这表明 ResGen 更有可能为这些未见过的蛋白质口袋生成易于合成的类药配体。
针对真实靶标的分子生成
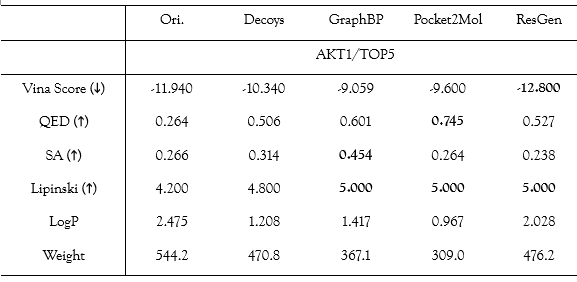
现有的大多数三维口袋条件分子生成研究都侧重于从算法设计的角度展示其模型的能力,却极少对这些模型在真实药物设计场景中的表现进行评估。因此本文中作者尝试了填补这一空白:作者精心挑选了几个具有代表性的治疗靶点,整理了其靶标结构以及具有实验活性的配体化合物,并随机选择了一批无活性小分子作为阴性对照。图 2A 显示了各组分子的结合亲和力分布,分布越偏左,结合能绝对值越大,亲和力越高。从中可以观察到ResGen 生成的分子不仅比阴性对照(Random)和其他 SOTA 模型生成的分子得分更高,而且整体分布甚至略好于实验活性分子。同时,尽管Pocket2Mol在之前的未见目标质量测试中表现出色,但由它生成的分子分布与随机集的分子分布较为接近。对于GraphBP而言,其生成的分子的对接得分分布实际上劣于随机集的得分分布,这表明其生成的分子与靶标间的结合并不紧密。图2C中显示的其直接得到的分子构象也表明了这一点。值得注意的是,从表2中可以得知,GraphBP 生成分子的SA 指标得分最高,可能是因为这些分子比其他方法生成的分子分子量更小,因此自然更容易合成。最后,考虑到表 3 中的所有指标,尤其是 logP 和分子量,作者得出结论:Pocket2Mol 生成的分子与随机集最为相似,而 ResGen 生成的分子则与活性分子最为相似。图2A 提供了更多佐证,随机分子和 Pocket2Mol 分子的结合亲和力分布几乎重叠,而ResGen分子的分布则位于最左侧。
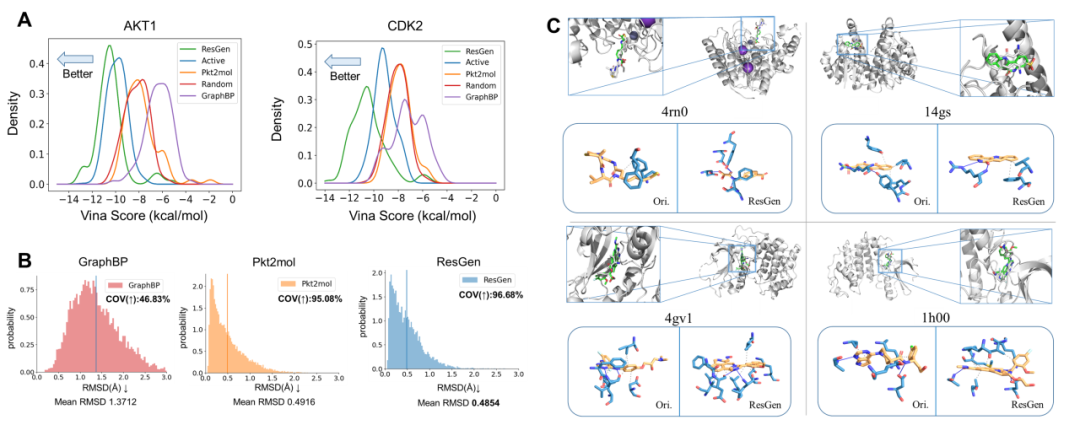
图2:三种方法在代表靶标数据集上结果。A)对接得分分布;B)构象质量实验分布,以RMSD为指标;C)相互作用分析案例。
表2:生成分子在代表靶标数据集上的性质(以AKT1为例)。
构象合理性实验
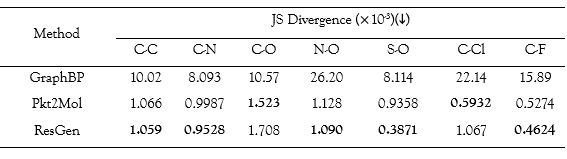
评估三维生成模型学习能力的另一个重要指标是生成构象的合理性。作者在本研究中进行了子结构和全局构象对比实验。为了评估子结构的几何合理性,作者采用定量指标 Jensen-Shannon Divergence (JSD)衡量了生成分子与训练分子之间常见类型的键长差异。如表3所示,ResGen 在六种键长中的四种上取得了最佳表现。在全局构象评估实验中,作者将直接从三维生成模型中提取的构象与通过经典构象生成算法ETKDG和UFF力场弛豫获得的构象进行了比较。具体来说,作者对模型生成的每个分子用所述的计算方法生成20 个构象,随后计算了这些构像与模型生成的原始构象间的RMSD。图 2B 提供了RMSD分布、RMSD 平均值和覆盖率(COV,构象生成任务的常用指标)。RMSD 平均值最低的结果表明,与其他两个SOTA方法相比,ResGen 能生成更平滑的构象,这突出表明了它在捕捉蛋白质口袋内部复杂几何分布方面的强大能力。另一个有趣的现象是,ResGen 获得的 COV 指标(96.68%)几乎与 SOTA 构象生成模型的 COV 指标(约 90%)相当,这暗示ResGen 在未来或许可以应用于另一项与分子生成密切相关的任务,即分子构象生成。
表3:不同方法的键长分布与训练集的键长分布比较。
针对AlphaFold预测结构的分子生成分析
在基于结构的分子生成方法中,科学家往往希望了解模型对蛋白质结构的敏感性。在分子生成当中,这体现为不同的蛋白质结构应当对应于不同的分子分布。要达到这种灵敏度,就需要模型对蛋白-小分子相互作用足够敏感。为了讨论这一问题,作者以X 射线晶体结构和AlphaFold 预测结构为条件分别生成了两组分子,并对了比这两组分子的结构特征。作者指出,这一实验当中主要存在两种情况:在第一种情况下,AlphaFold 预测的构象 “封闭 “了晶体构象中存在的口袋,导致模型无法在原口袋位置生成完整的分子,而是在新形成的空腔中生成小片段,如图3第二行所示。这一现象表明了ResGen 的分子生成过程灵敏地依赖于给定的蛋白质口袋。在第二种情况下,AlphaFold 预测构象中形成的口袋与晶体口袋相比差异较小,但是模型仍然可以捕捉到这种变化。如图3第四行所示,ResGen生成的分子更多地占据了AlphaFold 预测构象中的空腔结构(如图中红圈所示)。这一实验同样证明了ResGen对靶点结构的敏感性,也暗示了正确的蛋白结构对于SBMG策略的重要性。
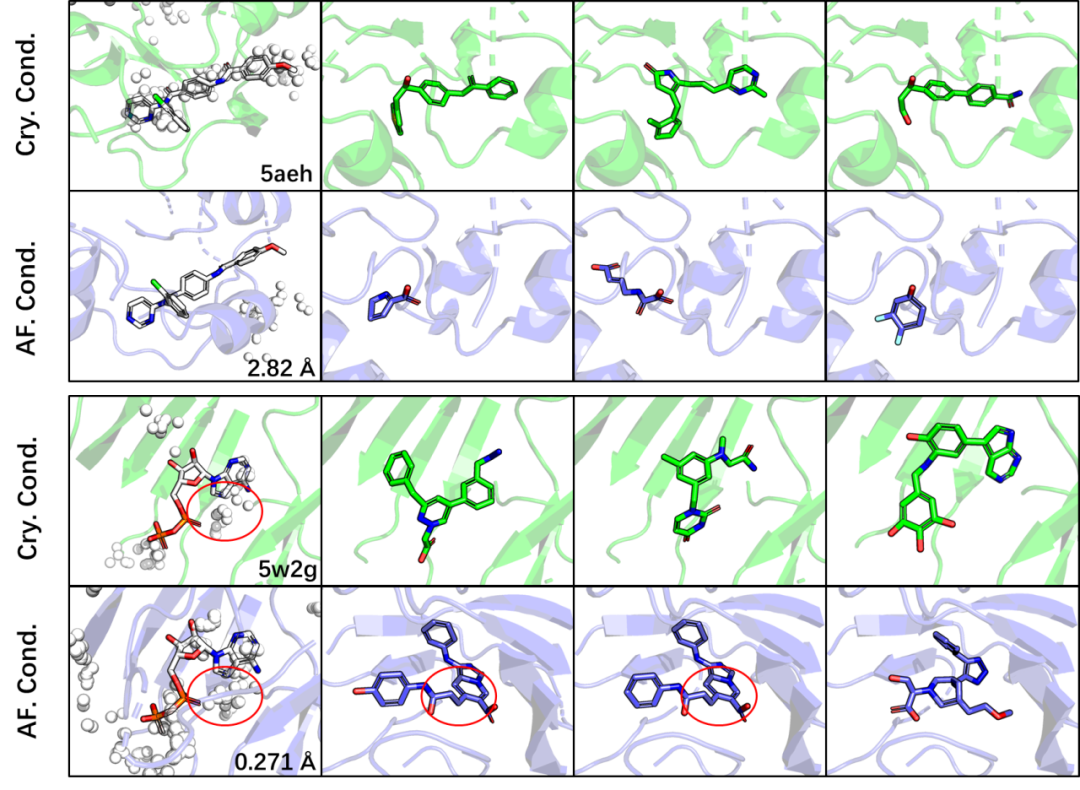
图3:基于晶体结构和AlphaFold预测结构生成的分子,其中白色配体为共晶配体,X Å 为经过对齐后预测结构与真实结构间的RMSD。第一列中的白色圆球代表可能的结合位点。
结论
在蛋白质口袋中生成三维分子是一个非常有趣的问题,但这一问题仍然具有很强的挑战性。受凝聚态物质和统计物理学中多尺度建模技术的启发,作者提出了一种新型的等变条件生成模型 ResGen,用于为任何给定的蛋白质口袋生成三维分子。文章中设计的实验从生成分子的性质,几何结构,在真实世界靶标上的表现,与靶点间的相互作用以及针对AlphaFold预测蛋白结构的生成等多个角度讨论了ResGen的生成能力,也为今后基于结构的分子生成方法的开发提供了诸多启发。
参考资料
Zhang, O., et al. ResGen is a pocket-aware 3D molecular generation model based on parallel multiscale modelling. Nature Mach Intell (2023)
https://www.nature.com/articles/s42256-023-00712-7



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


