编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Peter K. Koo的一篇关于基因组表示的论文。深度卷积神经网络(CNN)在对调控基因组序列进行训练时,往往以分布式方式构建表示,这使得提取具有生物学意义的学习特征(如序列模体)成为一项挑战。在这里,作者对合成序列进行了全面分析,以研究CNN激活对模型可解释性的影响。作者表明,在第一层过滤器中使用指数激活与其他常用激活相比,始终导致可解释且鲁棒的模体表示。令人惊讶的是,作者证明了具有更好测试性能的CNN并不一定意味着用属性方法提取出更可解释的表示。具有指数激活的CNN显着提高了用属性方法恢复具有生物学意义的表示的效果。

近年来,卷积神经网络(CNN)在基因组序列分析中变得越来越流行,在各种调控基因组预测任务中展示了最先进的准确性。然而,理解CNN为什么做出给定的预测仍然是一个挑战,这使得它们被称为黑盒模型。最近在解释模型预测方面取得了进展,主要是通过归因方法,如显著性图、集成梯度、DeepLIFT、DeepSHAP以及基因组学中的体外突变等方法,以及其他解释性方法。归因方法在基因组学中特别受关注,因为它们提供了每个输入核苷酸对模型预测的独立贡献,这一技术自然地扩展到评分单核苷酸变异的功能影响。实际上,解释性图可能难以解释,需要下游分析来获得更可解释的特征,例如通过平均聚类归因得分来获取序列模体。
在基因组学中,从训练好的CNN获取洞察力的另一种方法是通过可视化第一层过滤器,以获得显著特征,如序列模体;然而,最近的研究表明训练过程和设计选择会影响过滤器学习模体表示的程度。例如,在第一层后使用大的最大池化窗口大小会使部分特征的空间顺序变得模糊,从而阻止深层级将其层次化组装成完整特征表示。因此,CNN的第一层过滤器必须学习整个特征,因为它只有这一个机会。这些设计原则的一个缺点是它们局限于较浅的网络。网络的深度增加了其表现力,使其能够构建更广泛的特征。在基因组学中,深层网络在分类性能方面取得了更大的成功。显然,性能和可解释性之间存在一种权衡,这与网络的深度密切相关。
指数激活导致可解释的模式
指数激活函数在基因组学中是最常用的卷积神经网络(CNN)激活函数之一。其他的激活函数包括sigmoid、tanh、softplus和ELU。这些激活函数中,我们对指数激活函数很感兴趣,因为它在负值时被限制在零,而在正值时迅速偏离零。与ReLU或softplus激活函数不同,后者也将负值限制为零,但将正值线性缩放,高度偏离的指数函数原则上可以增强正信号的灵敏度同时保持较低的背景水平。指数函数的输入应该缩放到函数的敏感区域(最佳缩放因子根据信号和背景水平而变)。通过将激活函数设置为标准指数函数,网络可以通过第一层滤波器将预激活值缩放到这个阈值。此外,ReLU和softplus激活函数的线性行为可能更宽松,因为如果背景通过第一层传播,那么更深层次的网络仍然可以构建纠正这种噪声的表示。另一方面,对于具有指数激活的CNN,第一层和更深层使用ReLU激活函数,如果背景噪声通过第一层传播,那么其余被线性缩放的网络将无法处理这种指数放大的噪声。为了成功,网络必须选择一种在激活之前抑制背景噪声并仅传播具有辨别性的信号的策略,作者预计这将导致更可解释的第一层滤波器。因此,作者建议将指数激活仅应用于深层CNN的单一层(即所需具有可解释参数的层),而对其他层使用传统的激活函数,如ReLU。对于基因组学,第一层滤波器中高度理想的模式表示是指数激活函数的理想层。
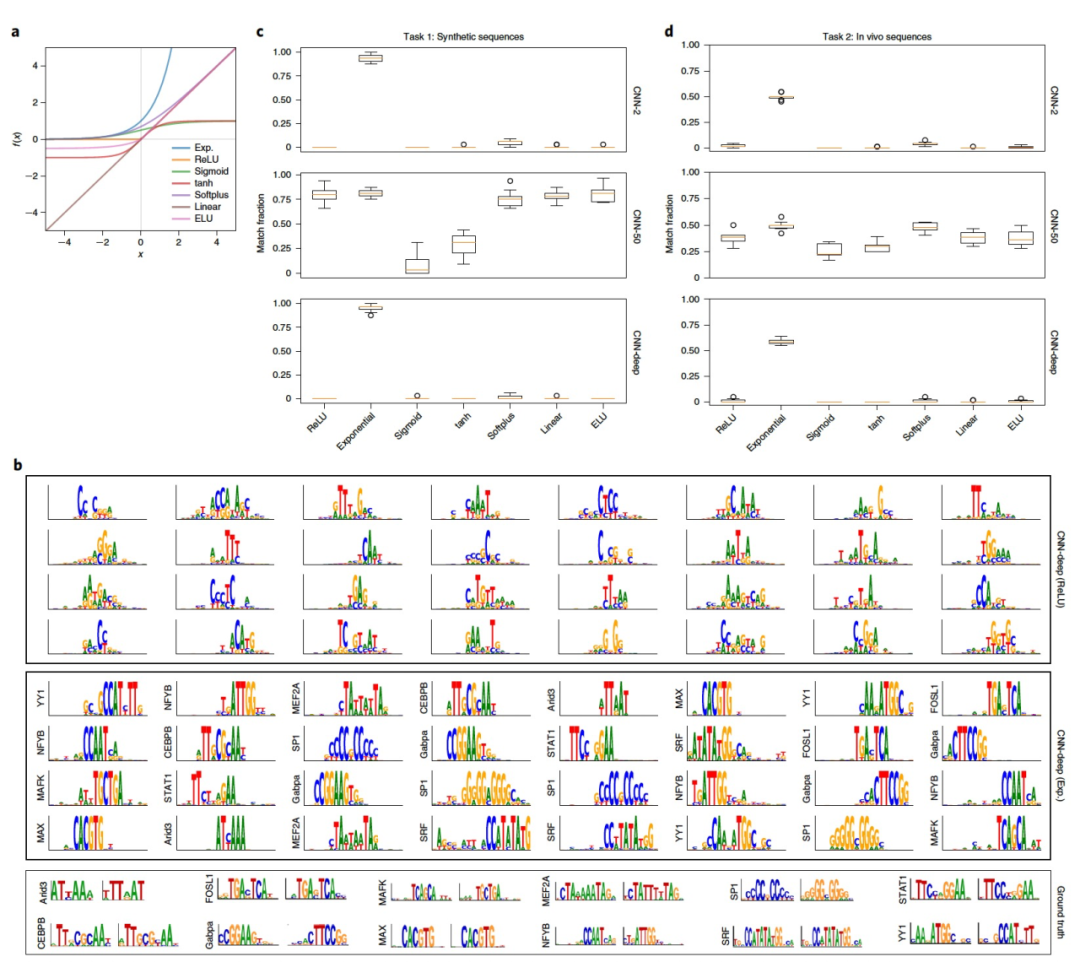
图 1
为了测试CNN激活对表示学习的影响程度,作者在一个多任务分类数据集(称为任务1)上均匀地训练和测试了使用不同第一层激活函数的各种CNN。任务1的目标是根据随机DNA序列中嵌入的转录因子模式的存在来确定类别成员资格,其中每个转录因子模式表示一个唯一的类别。作者探索了三种CNN,它们使用为具有ReLU激活的CNN设计的表示学习概念,即CNN-2、CNN-50和CNN-deep。CNN-50被设计为在两个卷积层后的第一个层之后有大的最大池化,从而为学习第一层的局部表示提供归纳偏差,而CNN-2使用小的最大池化,允许它通过将第一层中学习到的部分模式组合成更深层次的整体表示,以分层方式构建分布式表示。CNN-deep由四个卷积层组成,使用小的最大池化,旨在构建分布式模式表示。
在任务1的保留测试集上,通过AUPR给出的分类性能在所有网络和激活函数之间大体上是可比较的。CNN-deep表现出轻微的整体优势,而CNN-50则具有稍低的性能,tanh和sigmoid激活导致最差的分类结果。调整初始化策略可能会改善性能,但在这里没有进行探索以保持系统性的方法。为了量化学习到的第一层滤波器与真实模式的匹配程度,使用基于激活的对齐方法可视化第一层滤波器,并使用TomTom量化第一层滤波器中与真实模式存在显著匹配的比例(图1b)。在CNN-50中设计用于学习带有ReLU激活的整个模式表示的滤波器,也能够捕获其他激活函数下的真实模式,但sigmoid和tanh激活的性能较差,这是符合预期的(图1c)。另一方面,设计用于学习分布式表示的CNN(CNN-2和CNN-deep)对大多数激活函数来说无法做到这一点,唯一的例外是指数激活,在定量(图1c)和定性上都产生了改进的模式匹配结果。尽管指数函数的无界行为可能导致CNN激活发散,但在实践中,训练过程中没有明显的问题,其收敛时间与具有ReLU激活的CNN相当,并且梯度保持稳定。
由于指数激活抑制背景噪声并传播信号,第一层滤波器扫描可以用来标记序列上的模式实例。具有指数激活的CNN-deep的第一层滤波器扫描在序列上的位置上产生清晰的峰值,这些位置正是模式被植入的地方。用于模式扫描的黄金标准是位置权重矩阵(PWMs),它们是位置概率矩阵与背景核苷酸水平之间的对数比值。由于作者对任务1有着真实的结果,我们可以使用AUROC来量化模式定位性能。具有指数激活的CNN-deep产生了0.889 ± 0.201的定位AUROC,明显高于ReLU激活(0.391 ± 0.331,误差表示所有测试序列上平均值的标准偏差)。对于真实的PWMs,定位AUROC为0.884 ± 0.252。这证实了在理想情况下,PWM扫描是在序列上标记模式的一种强大方法。在实践中,模式的活性可能取决于周围核苷酸的上下文,并且在细胞类型之间可能有微妙的差异。此外,PWM的性能对于背景频率的选择也很敏感。CNN的优点在于它们能够以端到端的方式推断模式模式并进行预测。
学习稳健模式表示的CNN在解释方法方面更具强
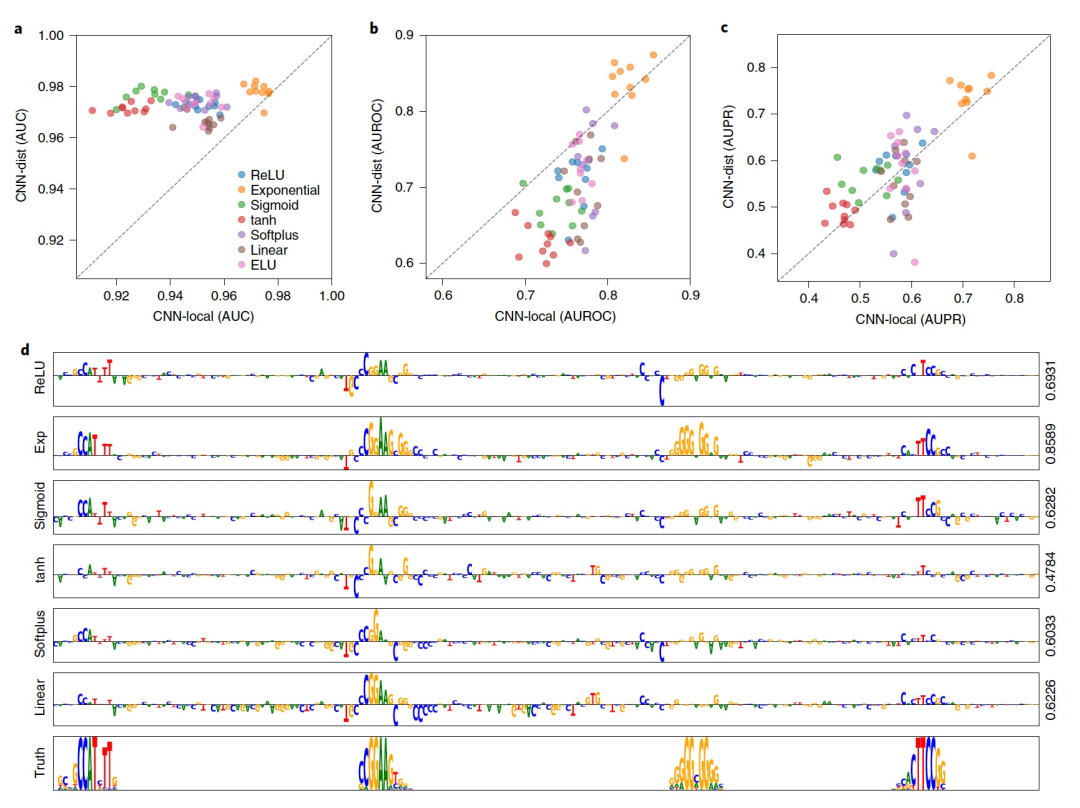
图 2
虽然滤波器可视化是评估CNN学习表示的强大方法,但它们并未指明决策是如何做出的。解释性方法的目标是通过识别对模型预测重要的输入特征来解决这个问题。为了理解激活函数在使用解释性方法恢复具有生物学意义的表示方面的作用,我们在一个合成的调控分类任务上训练了两个CNN(CNN-local和CNN-dist),该任务用于模拟染色质调控的billboard模型。这个二分类任务称之为任务3,旨在预测DNA序列是否包含至少三个从核心模式集合中采样得到的模式,而不是从背景集合中采样得到的模式。CNN-local是一个浅层网络,有两个隐藏层,旨在学习使用ReLU激活的可解释滤波器表示,而CNN-dist是一个深层网络,有五个隐藏层,学习特征的分布式表示。更好的准确性并不意味着更好的可解释性。根据AUC来衡量的分类性能在CNN-dist和CNN-local之间是可比较的,CNN-dist略优于CNN-local(图2a)。使用显著图(即预测相对于输入的梯度)给出的解释性得分,发现在大多数激活函数下,CNN-local的解释性AUROC略高于CNN-dist,唯一的例外是指数激活(图2b);另一方面,CNN-dist在AUPR指标下表现稍好(图2c)。每个指标描述了解释性得分的稍有不同方面。AUROC捕捉网络正确预测嵌入模式的能力,同时惩罚虚假的噪声,因此CNN-local不太容易将与真实模式无关的位置归因给模式(假阳性较低)。AUPR考虑了假阴性率,因此在这里改进的性能表明CNN-dist更擅长捕捉更多的真实模式,而CNN-local则可能会漏掉一些。这项研究的一个限制是作者只考虑了嵌入在随机序列中的真实模式,而没有考虑由于偶然而产生的虚假模式,这会导致标签噪声。
有趣的是,使用softplus和线性激活的CNN-dist相对于CNN-local表现出更高的分类性能,但在解释性指标下明显较低。这反驳了一般的直觉,即改进的预测模型应该更好地捕捉特征表示以解释改进的性能,并且提示预测性能不一定意味着在使用解释性方法时具有可靠的解释性。这种准确预测与模型可解释性之间的差异在计算机视觉领域也被观察到。值得注意的是,作者发现指数激活在所有测试的CNN上始终导致更优异的解释性能,无论是定量指标(图2b,c)还是定性指标(图2d)。
结论
在基因组学中,深度学习的一个重要优势是其能够自动从数据中学习特征,从而实现准确的预测。模型的可解释性对于理解这些特征至关重要。然而,深度CNN往往学习分布式的序列模式,这些模式对于人类来说过于复杂难以理解。虽然归因方法可以识别影响决策的输入特征,但在实践中,它们的分数往往是嘈杂的且难以解释。在这里,作者展示了在第一层应用指数激活的强大方法,它可以鼓励第一层滤波器学习序列模式,并且还可以提高归因得分的效力,揭示更可解释的表示。这项研究带来的一个主要后果是引起了警示,即一个具有较高分类性能的CNN不一定会在使用归因方法时提供有意义的表示。以前的研究主要集中在使用单个模型比较不同归因方法得到的表示。在这里,我们展示了不同模型(每个模型的分类性能相当)使用相同的归因方法得到了相当不同的表示。我们相信学习分布式表示的CNN可能学习了一个构建更多噪声模式的函数,这可能不会直接影响分类性能,但会导致解释性差。研究底层函数的特性是解决这个问题的关键。
参考资料
Koo, P.K., Ploenzke, M. Improving representations of genomic sequence motifs in convolutional networks with exponential activations. Nat Mach Intell 3, 258–266.
https://doi.org/10.1038/s42256-020-00291-x



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


