编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Fabian J. Theis和Mohammad Lotfollahi的一篇关于细胞形态学的论文。高通量筛选技术的进步使得我们能够探索富含表型信息的方法,例如高内容显微镜技术,从而加速药物靶点鉴定和作用机制研究。然而,将这些实验扩展到庞大的药物或基因干扰空间面临挑战,因为只有少数化合物在筛选中显示活性。尽管机器学习方法在各种应用中被广泛使用,但在预测涉及未知现象的场景时,特别是将未见过的控制细胞图像转换为所需的干扰现象,机器学习方法并未表现出可靠的能力。作者提出了一种生成模型,即图像干扰自编码器(IMPA),它利用未经处理的细胞图像作为输入,预测化学和基因干扰的细胞形态学效应。

高通量基于图像的特征分析技术的出现使得我们可以对数千种化学和基因干扰进行筛选,以鉴定潜在的药物靶点、作用机制和基因功能。这些特征分析测量细胞亚细胞结构的形态学变化,通过使用多种荧光染料对其染色,以突出不同的细胞器和组分。例如,Cell Painting 是最常用的无偏见的基于图像的特征分析方法,可以使用荧光染料测量不同的亚结构。虽然当前技术具有可扩展性,但探索可能合成的大量药物分子或基因干扰的组合空间是不可行的。因此,进行更大规模的筛选实验可能面临实验上的挑战和高昂的成本。为了改进干扰空间的导航,以实现最优实验设计,我们需要计算方法来预测实验中未测量的干扰引起的形态学响应。生成的细胞响应可以用于缩小假设空间,促进基于表型的药物发现。
在高通量图像数据中预测表型响应的计算方法已经在监督和无监督的设置中进行了探索。监督任务包括化合物作用机制(MoA)和药物毒性的预测,以及实验活性注释。相反,无监督方法可用于在特定干预和条件下生成细胞图像特征的虚拟表示,或预测(多药效)药物组合的响应。然而,目前缺乏一种生成模型,能够对干扰图像进行反事实预测。在生物数据的生成建模方面,MorphNet是一个生成对抗网络(GAN)模型,基于基因表达合成细胞形态学,但没有解决干扰特异性图像生成。其他最近的研究专注于表示学习。例如,CLOOME通过对比学习学习图像和药物分子结构的联合表示,并利用嵌入空间进行药物或图像条件的实例检索。然而,该模型并没有设计用于预测对控制细胞的干扰响应。同时,与作者的工作同时进行的研究中,域适应的生成建模已被用于在高内容显微镜筛选中创建批次不变的细胞表示,但其在细胞形态学干扰预测方面的应用尚未探索。同时,Mol2Image采用条件流式生成模型,根据化学化合物的结构生成细胞图像。然而,它对未知药物合成图像的详细探索不足,并且没有考虑干扰引起的控制细胞的形态学变化。
总结起来,这一空白促使作者采用一种新的方法来回答问题:“如果未处理的细胞受到特定干扰,它的图像会是什么样子?”为了解决这个需求,作者引入了图像干扰自编码器(IMPA),这是一个深度生成模型,旨在预测高通量图像特征分析筛选中小分子和基因干扰对细胞的响应。IMPA采用了图像转换任务的风格转移方法。该模型学习将细胞图像分解为其风格(干扰表示)和内容(细胞表示)。通过训练,IMPA可以将细胞转换为所需的风格,即干扰,同时保持其与干扰无关的内容。IMPA的独特之处在于它利用了非配对数据,消除了在处理前后筛选相同细胞图像的要求。考虑到在大规模实验中收集配对图像的不切实际性,这一方面至关重要。
使用风格和内容表达式学习干扰引起的形态学响应
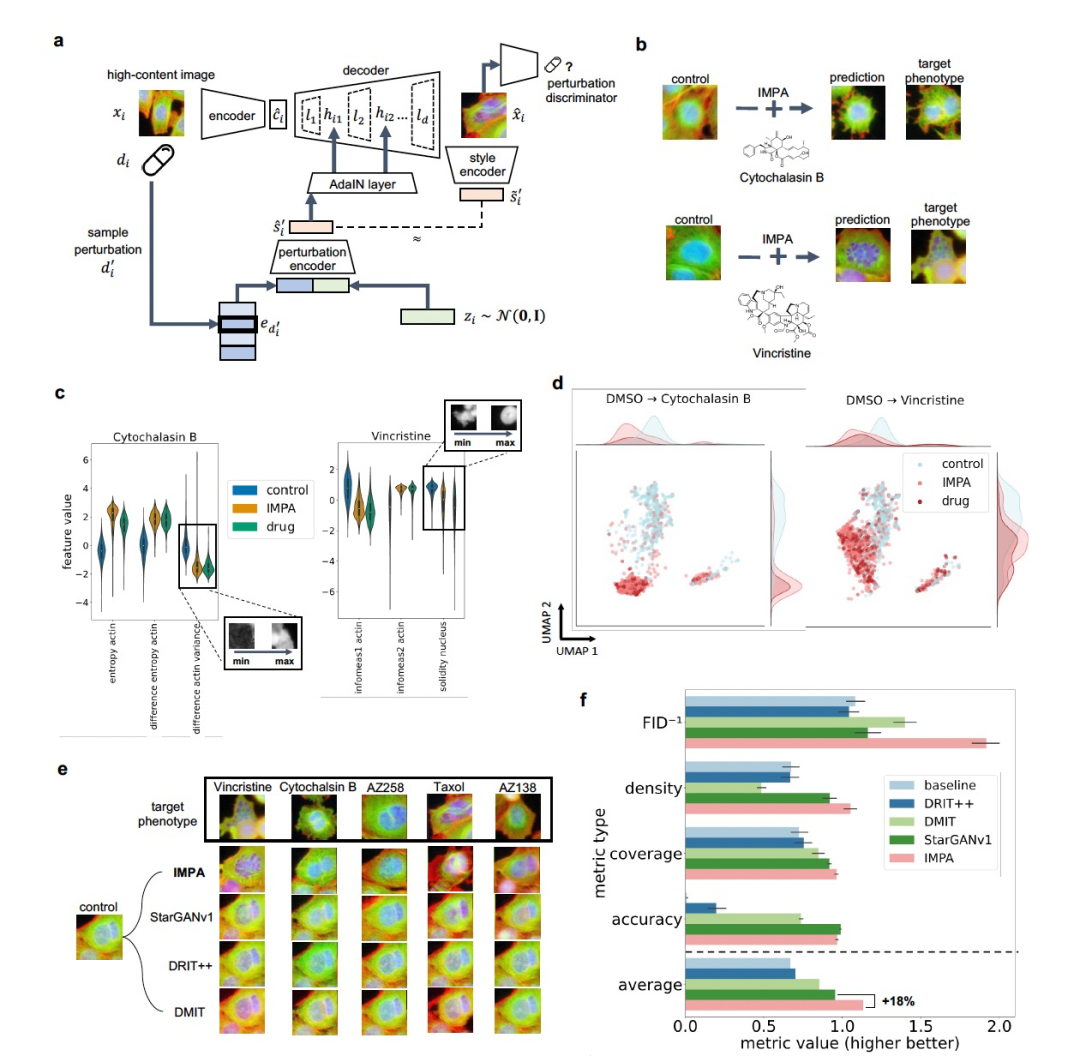
图 1
我们通过将每个图像的表示分解为其所受的干扰(即风格)和细胞的表示(即内容),来建模高内容成像筛选中对干扰的表型响应。这种方法基于“风格迁移”,是深度学习和计算机视觉中的一个活跃研究领域。风格迁移涉及将图像的特征(例如艺术风格)修改为另一个图像的特征,同时保持原始图像的内容。按照这种方法,作者开发了IMPA,一种条件生成对抗网络,给定控制细胞的输入图像,可以生成对所需干扰的反事实图像响应(见图1a)。IMPA基于StarGANv2中提出的架构,但作者对条件机制进行了修改,以支持使用先前干扰嵌入。这种建模方面为IMPA的干扰选择和表示提供了灵活性,允许使用多样的示例,例如将药物的分子结构作为干扰建模的风格,将基因敲除研究中的共表达基因表示作为干扰建模的风格。为了确保预测正确地匹配所需的干扰风格,我们针对每个干扰训练一个鉴别器分类器,用于区分经过此类干扰处理的真实细胞图像与生成的图像。解码器网络必须合成出关于目标效应的准确图像,以欺骗多任务鉴别器将其分类为目标风格(干扰)的真实图像。相反,鉴别器在真实数据上进行训练,以识别真实和生成的处理图像之间的区别。多任务鉴别器并不尝试对不同干扰之间的图像进行分类,而是根据干扰类别预测图像是真实图像还是生成图像。这种方法使得在不同干扰下的表型响应相似且难以分类时更加适用。最后,采用循环一致性损失来鼓励模型学习可逆的映射,从而将受干扰的图像转换回输入条件的风格。所有单元之间的相互作用在StarGANv2中被证明能显著提高生成性能。
IMPA准确地预测了药物干扰后的形态学变化
BBBC021数据集包含了受到113种化合物干扰的p53野生型乳腺癌细胞模型的图像,并在三个通道上进行了成像:细胞核、β-微管蛋白和肌动蛋白。作者数据预处理为围绕单个细胞裁剪的图像,共得到了约97,000个图像块。为了更直观地评估生成的图像,我们首先在五种药物(AZ138,AZ258,Cytochalasin B,Taxol,Vincristine)的子集上对模型进行了训练,这些药物与对照组相比,对形态学有明显的影响。对于这种情况,作者使用RDKit软件包提取了每种药物的干扰嵌入。每个嵌入表示定义了从分子图中原子邻域导出的拓扑和结构药物属性的描述符。对预测的定性分析显示,IMPA将控制细胞转换为目标干扰风格,同时保留了细胞方向和平移等内容信息(见图1b)。例如,已知Cytochalasin B可以阻断肌动蛋白的聚合。IMPA对Cytochalasin B的预测结果显示在肌动蛋白通道中相对于源控制图像,其产生了均匀性和延伸的丧失。
为了评估IMPA对表征干扰的重要特征的影响,作者使用CellProfiler软件计算了一系列356个特征,包括细胞图像在IMPA转换前后的形状、纹理、面积和强度分布。作者的目标是调查IMPA如何影响这些特征,并与实际干扰图像相比较控制细胞的特征对齐(见图1c)。为了对每种干扰进行相关特征的子集化,作者通过使用CellProfiler特征训练了一个随机森林分类器,在单独的二元分类设置中区分控制细胞和受干扰细胞,来估计特征的重要性。在转换后的图像中重要形态特征的分布与实际图像相比较控制图像时,显示出类似的偏移(在图1c中显示为基线)。作者进一步将IMPA与三个现有的GAN模型进行了比较,这些模型执行图像风格转换任务。值得注意的是,这些先前的模型并非特别设计或在先前干扰嵌入的条件生成情况下使用,见图1e。为了定量比较这些模型,作者使用了三个常用的GAN评估指标:Fréchet Inception Distance (FID),density和coverage,这些指标评估生成图像的分布与目标真实图像分布之间的相似性。此外,作者还测量了一个准确度指标,评估预训练分类器在正确标记生成图像的目标药物的作用模式方面的准确性。为了建立下限性能,我们包含了对比控制细胞和真实干扰图像之间的基线比较,模拟无法进行风格转换的模型。作者的研究结果显示,IMPA在四个指标中的三个方面优于对比的风格转换模型(见图1f)。具体来说,IMPA在FID方面的结果减少了27%,在覆盖率方面增加了5%,在密度方面增加了14%,在所有指标中与第二好的方法相比,整体性能提高了18%。在作用模式分类准确度方面,IMPA的表现与StarGANv1相当,仅出现2%的准确度下降,而明显优于其他所有模型。
IMPA能够对未见过的药物进行外推预测
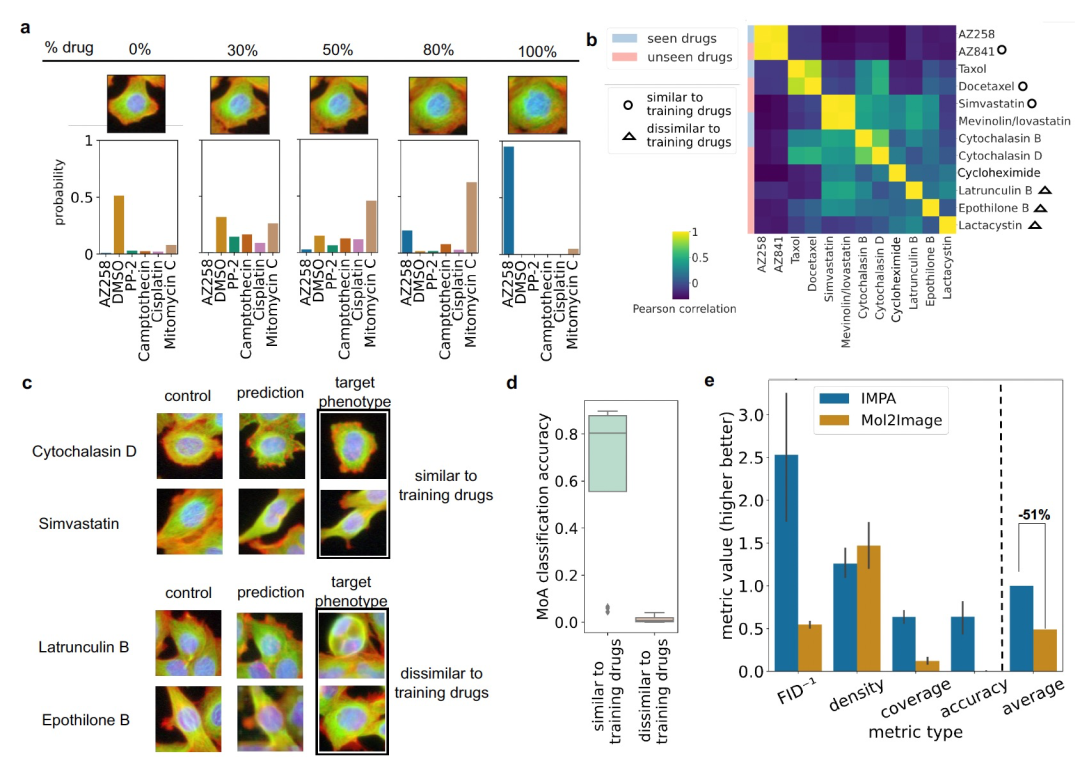
图 2
对于药物筛选来说,一个长期存在的挑战是选择化学库,以覆盖化学空间的不同区域,对应不同的作用方式。然而,由于实验成本和物流问题,完全探索所有可能的化合物是不现实的。有意义的风格编码可以用于导航化学空间,以了解具有类似效应或介于未处理和处理细胞之间的中间表型的药物(见图2a)。为了展示风格空间可进行插值的特性,作者研究了在控制组药物编码和目标药物编码之间的线性路径上生成细胞图像的效果。风格空间的插值可能为我们提供关于各种方面的见解,比如对多个干扰的响应相似性或类似作用方式药物在表型谱上的效应大小差异等。然而,需要进一步研究来全面探索和理解药物空间遍历的含义以及它们与形态学变化的关系。作者通过使用分类器将中间图像分类到训练药物中,对结果进行了检查。对于药物AZ258(见图2a),分类器正确将未经转换的图像指派给对照组,将完全转换的图像指派给目标药物类别。实验表明,在产生最终的目标表型之前,分类器对于将转换后的图像标记为AZ258或Mitomycin C之间不确定,而添加完整的药物风格会生成可靠的AZ258图像。在大规模筛选中进行训练时,IMPA可以逐渐用不同水平的药物风格干扰控制图像,并探索未见过治疗周围的化学空间,从而定义与已知对细胞形态有影响的分子的相似性。
IMPA预测基因干扰的表型响应
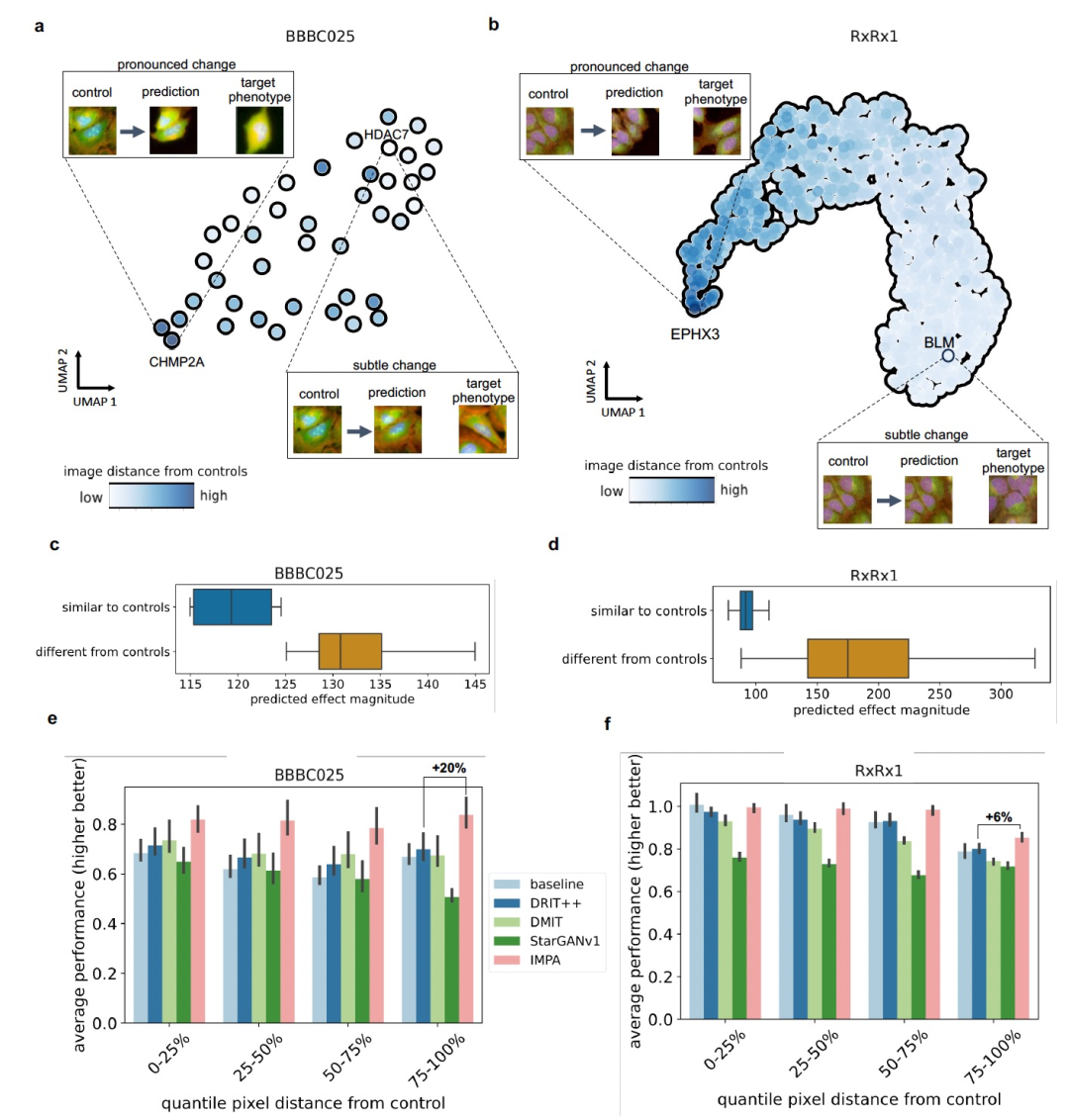
图 3
为了评估IMPA的泛化能力,并测试其在更微弱形态学效应的复杂情况下处理的能力,作者对使用U2OS细胞的两个基因敲除数据集进行了实验。为了评估模型的表示能力,作者通过使用风格编码器提取图像特征并计算它们的平均值来为每个干扰生成一个嵌入,从而获得每个干扰一个单一的点。然后,在二维空间中可视化这些表示(见图3a-b)。为了评估这种干扰表示的分布是否与效应大小相关,作者在二维UMAP坐标上注释了每个干扰与其相应控制之间的平均距离。由于干扰和控制的图像是不配对的,作者考虑控制和干扰之间的FID来定义响应大小。这个分数用于计算两组图像的感知差异,方法是根据预训练的卷积网络计算它们的特征表示的Wasserstein距离。作者的结果显示UMAP图上有一个连贯的渐变,特别是在RxRx1数据集中。更具体地说,有影响的干扰的平均风格编码逐渐与类似控制的干扰分开,而类似控制的干扰聚集在一起(见图3a-b)。此外,结果表明IMPA有效地预测了两个数据集中的积极敲除的表型效应当选择与控制之间距离最大的干扰时。相反,其他图像转换模型,如StarGANv1和DRIT++,在这个任务上失败,要么产生次优的结果,要么无法捕捉到积极干扰引起的形态学差异。
在大规模、无偏的表型屏幕中,预期只有一小部分干扰会产生显著效应。因此,对于生成模型来说,捕捉其输出中的效应和缺失效应都是很重要的。为了调查这一点,作者用FID来量化IMPA对控制图像在转换前后的距离(见图3c-d)。作者考虑了与控制之间最近和最远的10%的干扰,期望在后者类中观察到明显更强的形态学变化。如预期的那样,在其图像与相应控制图像接近的干扰中,IMPA在BBBC025和RxRx1数据集中分别导致10%和50%的平均形态学变化较弱,相比于其未处理对照的最远10%的基因敲除。这一观察结果表明,作者模型引起的表型转换与数据集中观察到的实际干扰效应大小相关。此外,作者评估了模型的性能与RxRx1和BBBC025数据集中干扰和控制图像之间的距离有关(见图3e-f)。
结论
在高内容筛选中合成预测干扰引起的形态学响应对于指导实验设计朝向有希望的治疗方案至关重要。为此,作者实现了IMPA,一种条件生成对抗网络,将未处理细胞的图像变形为在查询干扰作用下的样子。IMPA获取特定处理的潜在编码,并将其作为风格叠加到控制细胞图像上,以展示由于干扰引起的表型效应。
参考资料
Palma, A., Theis, F. J., & Lotfollahi, M. (2023). Predicting cell morphological responses to perturbations using generative modeling. bioRxiv, 2023-07.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


