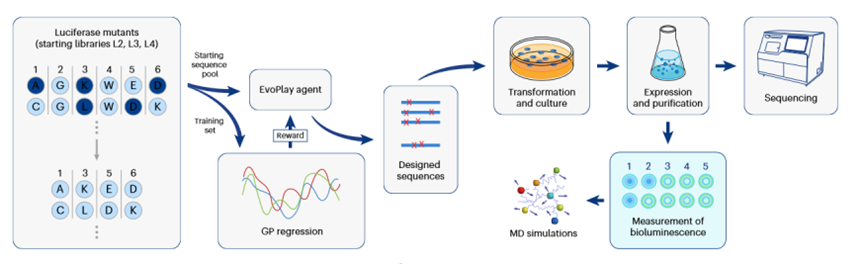
 蛋白质工程能够通过定向进化等手段设计具有特定生理功能的蛋白质序列,这在药物发现和酶工程领域都有重要的应用。机器学习指导的蛋白质定向进化(Machine-learning-guided directed evolution, MLDE)能够进行序列空间的高效采样、加快优化周期、减少实验的投入。
深圳华大智造的杨梦团队将蛋白质序列空间采样和序列优化任务巧妙地转化为在棋盘上落子与对弈行为,提出了一个基于AlphaZero的自博弈强化学习算法Evo Play。Evo Play能够指导蛋白质序列的定向进化,作者将其应用于结合多肽的序列设计和荧光酶的结构改造,并通过生物实验验证活性。该项研究工作近日已在Nature Machine Intelligence期刊上发表(Nat. Comput.
蛋白质工程能够通过定向进化等手段设计具有特定生理功能的蛋白质序列,这在药物发现和酶工程领域都有重要的应用。机器学习指导的蛋白质定向进化(Machine-learning-guided directed evolution, MLDE)能够进行序列空间的高效采样、加快优化周期、减少实验的投入。
深圳华大智造的杨梦团队将蛋白质序列空间采样和序列优化任务巧妙地转化为在棋盘上落子与对弈行为,提出了一个基于AlphaZero的自博弈强化学习算法Evo Play。Evo Play能够指导蛋白质序列的定向进化,作者将其应用于结合多肽的序列设计和荧光酶的结构改造,并通过生物实验验证活性。该项研究工作近日已在Nature Machine Intelligence期刊上发表(Nat. Comput.Sci. 2023, 5, 845–860.)【1】。 作者首先将经典的强化学习算法应用于蛋白质序列设计中。EvoPlay接收全长和指定区域的蛋白质序列作为输入,将序列上残基的突变事件定义为一个动作空间,通过神经网络指导的蒙特卡罗树搜索(Monte Carlo tree
search, MCTS)算法在蛋白质序列空间中不断突变、搜索和优化序列,对每个突变状态进行评分,并在序列满足预定要求时停止并输出优势序列。 EvoPlay能够避免其他强化学习模型仅在终态序列进行评分和奖励的弊端,在增强传统MLDE采样效率的同时还能够外接序列功能或结构预测模型(如Alphafold2),实现端到端地输出序列对应的功能性质和三维结构。 作者使用以GP回归评分值为奖励,选取了4个具有代表性的多肽体系用以验证EvoPlay能否高效地生成高质量的结合肽序列。对于其中的核糖核酸酶体系,EvoPlay生成的序列展示出显著的新颖性和多样性(图1),且表现出与RNase1良好的结合构象(图2)。实验测定结果也验证了EvoPlay设计肽与RNase1靶标具有比野生型更强结合亲合力。
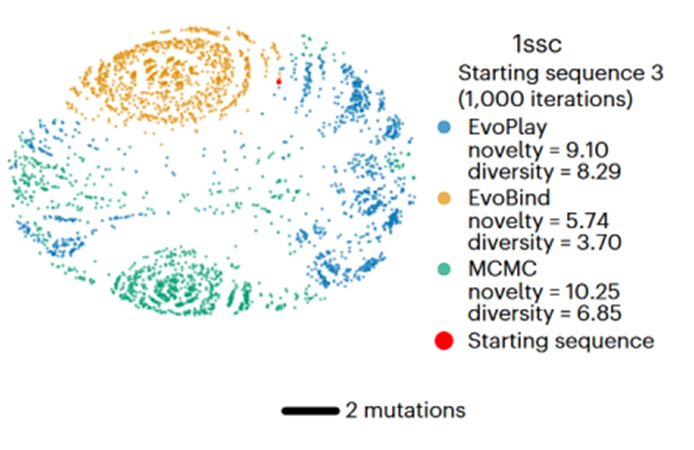 图1:EvoPlay设计的多肽序列新颖性和多样性
图1:EvoPlay设计的多肽序列新颖性和多样性
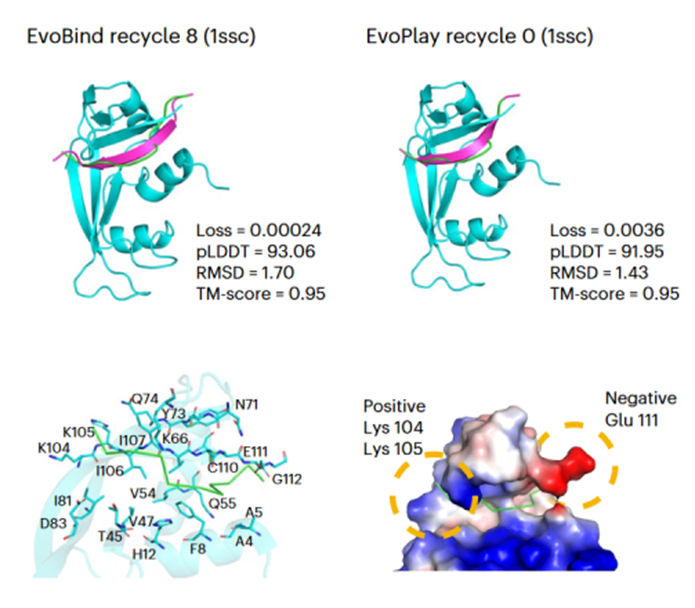 图2:多肽–蛋白复合物叠合结构、界面残基以及表面静电势
作者还使用EvoPlay模型对荧光素酶进行了序列探索,设计了29个荧光素酶突变体,并通过实验评估这些突变体的荧光强度,其中有11个超过起始突变库的最大值,有4个超过野生型荧光强度的6倍(图4左),并且分子动力学模拟(MD)显示突变体具有与野生型相似的肽–复合物结合模式(图4右)。
图2:多肽–蛋白复合物叠合结构、界面残基以及表面静电势
作者还使用EvoPlay模型对荧光素酶进行了序列探索,设计了29个荧光素酶突变体,并通过实验评估这些突变体的荧光强度,其中有11个超过起始突变库的最大值,有4个超过野生型荧光强度的6倍(图4左),并且分子动力学模拟(MD)显示突变体具有与野生型相似的肽–复合物结合模式(图4右)。
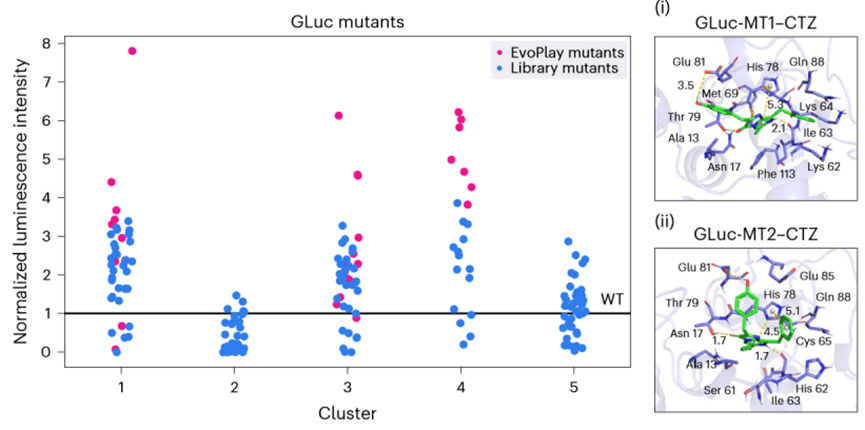 图4:29个Evo Play设计和起始突变体库的发光强度以及MD模拟复合物界面结构
小结:本研究将强化学习博奕类算法应用于蛋白质分子的定向进化中,开发了一个指导蛋白质设计的模型框架EvoPlay。作者运用EvoPlay在多种蛋白–多肽体系上进行了实践,显著提高复合物结合亲合力和酶活性,验证了其在高效设计高质量多肽/蛋白质上的性能。相对于其他强化学习方法而言,EvoPlay能够对每个突变状态进行采样,避免了仅从终态获得稀疏奖励、陷入局部最优的弊端,在计算速度和泛化能力上也具有较强的优势。同时,作者认为EvoPlay能够进行添加蛋白骨架的残基采样概率作为输入、考虑酶的构象并将残基级突变拓展至片段级等改进,应用于更多的蛋白质工程场景中。
参考文献【1】 Wang,
图4:29个Evo Play设计和起始突变体库的发光强度以及MD模拟复合物界面结构
小结:本研究将强化学习博奕类算法应用于蛋白质分子的定向进化中,开发了一个指导蛋白质设计的模型框架EvoPlay。作者运用EvoPlay在多种蛋白–多肽体系上进行了实践,显著提高复合物结合亲合力和酶活性,验证了其在高效设计高质量多肽/蛋白质上的性能。相对于其他强化学习方法而言,EvoPlay能够对每个突变状态进行采样,避免了仅从终态获得稀疏奖励、陷入局部最优的弊端,在计算速度和泛化能力上也具有较强的优势。同时,作者认为EvoPlay能够进行添加蛋白骨架的残基采样概率作为输入、考虑酶的构象并将残基级突变拓展至片段级等改进,应用于更多的蛋白质工程场景中。
参考文献【1】 Wang,Y., Tang, H., Huang, L. et al. Self-play reinforcement learning guides
protein engineering. Nat. Mach. Intell. 2023, 5, 845–860.
正文完
可以使用微信扫码关注公众号(ID:xzluomor)



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


