编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Martin A. Walsh, David I. Stuart和Payel Das的一篇关于药物发现的论文。对于新出现的药物靶点蛋白,药物抑制剂的发现是具有挑战性的,尤其是当目标结构或活性分子不为人所知时。在这里,作者通过实验证实了一个大规模训练的深度生成式框架在蛋白质序列、小分子和它们之间的相互作用上的广泛适用性。

新型分子设计是一个具有挑战性的问题,它涉及提出具有期望性质的以前未被识别的化合物,在药物研发和材料工程领域有广泛应用。例如,在药物研发工作流程中的一个关键目标是识别候选分子,这些分子可以与已知的药物靶蛋白及/或相关通路发生相互作用,并具有可测量的活性抑制。寻找这些抑制剂化合物通常涉及对包含标准化学化合物或较小化学片段的库进行高通量筛选。该方法的成功率在0.5%至1%之间,这取决于所筛选库的大小和目标特性。这低的成功率部分是由于巨大的搜索空间,现在估计在10^33至10^80可行分子之间,而其中只有极小一部分通常具有所需的特征。对这个广阔的化学空间进行穷举列举是不可行的,因此对要进行筛选的化合物进行优先排序是具有挑战性的,并且难以有把握地进行。除了需要进行成千上万次的筛选实验外,所选库的初步选择通常需要对感兴趣的目标蛋白与已知配体的结构信息有详细了解。
基于深度学习的生成模型具有潜力以“无规则”的方式实现对具有所需功能的先前未知分子的发现,因为它们首先旨在学习已知化合物的密集连续表示(以下简称潜在向量),然后修改这些潜在向量以解码为未见过的分子。因此,这些模型为先前未被有意识人类偏见限制的未开发化学空间提供了访问权限。然而,对于特定靶点药物样式的抑制剂设计任务,必须采用“反向分子设计”方法,其中通过分子属性属性来引导对学习化学表示的导航。在设计针对先前未知靶点的抑制剂时,需要足够数量的示范分子,这很可能是不可得的,需要昂贵且耗时的筛选实验才能获得。由于大多数现有的深度生成框架仍依赖于从特定靶点库的结合物化合物中进行学习,它们限制了超越已知和单一分子的固定库的探索,同时阻止了机器学习框架向先前未知的靶点的泛化。因此,尽管一些使用深度生成模型进行特定靶点抑制剂设计的研究已得到实验验证,目前尚未报道过这些模型在没有详细的靶点特异性先前结合数据的情况下,用于处理跨不同蛋白靶点的验证抑制剂发现的演示。
基于深度生成模型的属性条件分子生成
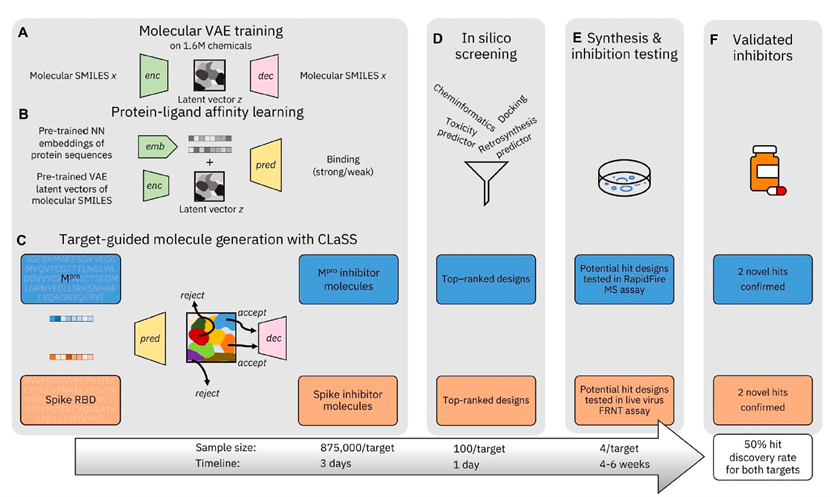
图 1
作者提出的抑制剂发现流程如图1所示,包括三个主要步骤:(A至C)使用深度生成框架在目标条件下进行候选物设计,(D)进行体外筛选以进行候选物优先级排序,以及(E)对优先选择的分子进行湿实验验证。对于全新分子设计,作者使用深度生成框架CogMol作为基础,该框架使得可以为不同的靶点设计抑制剂分子,而无需在靶点特定数据上进行训练或微调模型。
CogMol的工作原理如下:首先,它使用变分自动编码器(VAE),这是一类基于深度学习的流行生成模型,作为生成基础(图1A)。AVAE由一对神经网络组成,即编码器-解码器对。编码器神经网络将分子的简化分子输入行记录系统(SMILES)字符串映射为低维表示。解码器,也是神经网络,然后将潜在向量z转换回重建的SMILES 。解码器是随机的——它从潜在分布中采样以产生输出。编码器-解码器对进行端到端训练,同时优化两个目标。第一个目标包括最小化损失项,以确保从相应的潜在嵌入准确重构输入SMILES。第二个目标包括一个正则化项,将潜在编码限制为标准正态分布。由此产生的潜在空间是连续的,使得可以平滑地插值以及从潜在空间随机采样各种不同的分子。为了学习具有关于多种化合物的通用知识的有意义的潜在分子表示,在CogMol中,VAE在公共数据库中的超过160万个小分子上进行训练。一旦学习了化学潜在表示,CogMol在该表示上执行属性条件抽样,以生成具有向设计规范倾斜属性的未见过的分子实体。
从机器设计的配体库中进行候选物优先级排序
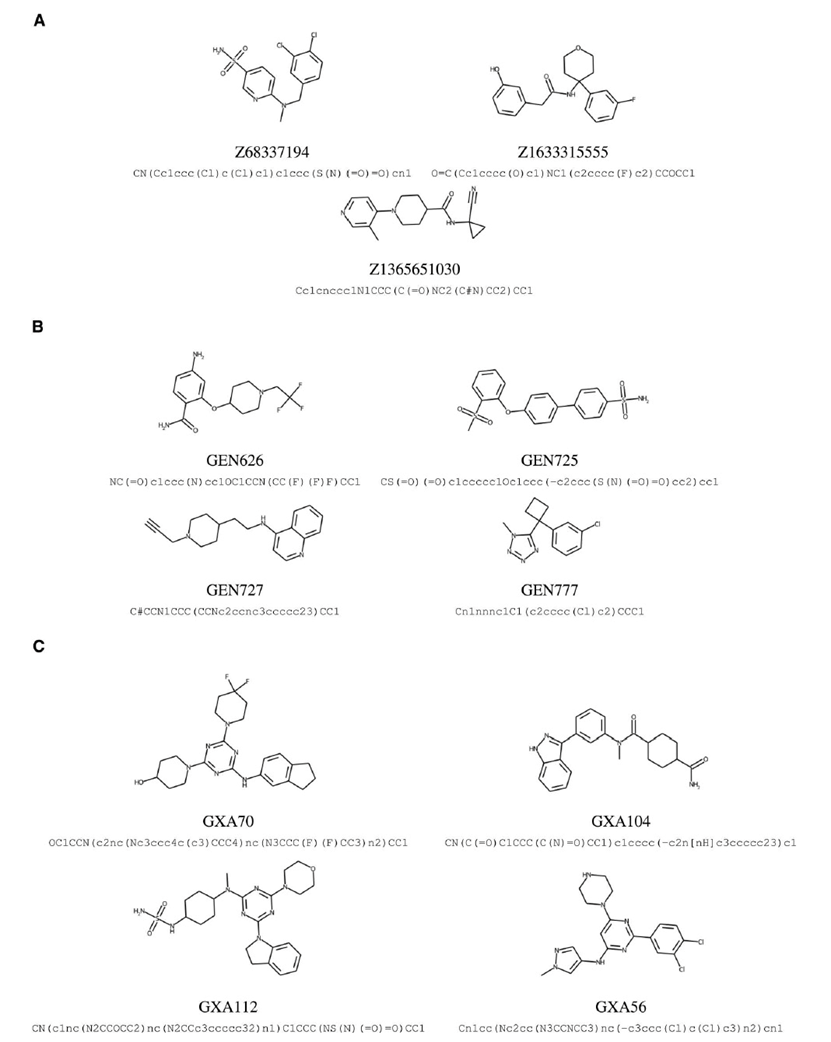
图 2
接下来的阶段包括对生成的候选物进行体外筛选(图1D),以便为其合成和湿实验评估进行优先级排序。出于实际考虑,作者试图将要合成和测试的机器设计的全新化合物数量保持非常少,每个靶点大约10个。通过进行仔细的分析,包括基于机器学习的反向合成预测,来定义这个集合。作者使用了一组物理化学属性、通过对接模拟预测的靶分子结合自由能以及使用机器学习进行的反向合成和毒性预测。对于反向合成预测,作者使用了基于变换器神经网络的IBM RXN平台,该网络在化学反应数据上进行了训练。对于毒性预测,使用了基于神经网络的内部模型,该模型在公开可用的体外和临床毒性数据上进行了训练。在体外筛选结束时,每个靶点的候选物数量约为100个,由化学制造商Enamine有限公司的谨慎判断进一步缩减至每个靶点约10个。通过有机合成化学家专家的评估预测反应模式的可行性,以及预测反应物的商业可用性和成本,确定了候选物合成清单。根据Enamine提供的合成成本和交付时间,选择了每个靶点的最终四个候选物。图2列出了由生成式机器学习框架设计并合成的八种全新化合物。其中五种化合物使用了IBM RXN的预测的最佳路径进行合成。对于两种化合物,GEN626和GEN777,预测结果不成功。对于GXA104,RXN预测中包含的反应物不可用。总的来说,这些结果显示了基于机器学习的反向合成预测的实用性,能够可靠地识别出可行的候选物并推荐可行的合成途径。
全新设计的针对RBD的化合物表现出对基于尖峰蛋白的伪型病毒和活病毒的抑制效果
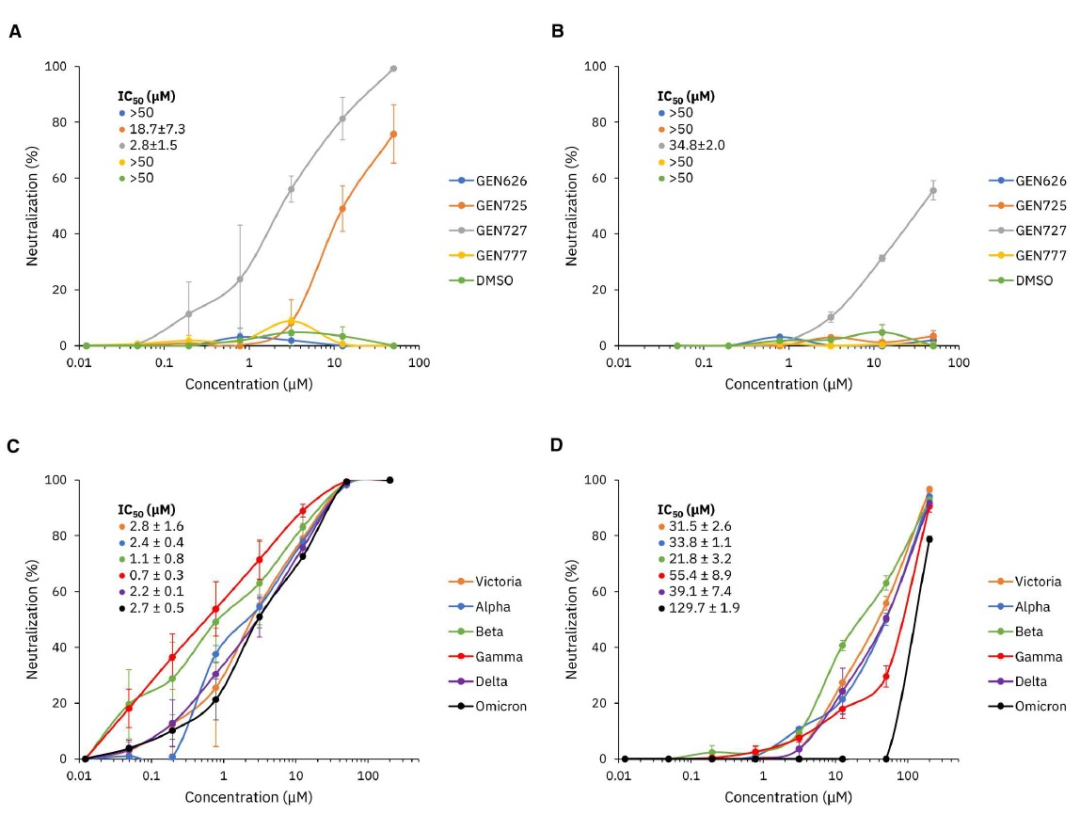
图 3
针对蛋白RBD设计的CogMol化合物,作者使用含有尖峰蛋白的分离物来测量其中和能力。这些结果总结如图3所示。在四种候选物中,GEN725和GEN727显示出IC50值小于50μM(分别为18.7μM和2.8μM),表明发现了具有合理抑制伪病毒50%成功率的先前未知的化合物(图3A)。GEN727还表现出对活病毒的中和能力(图3B)。作者进一步检查了GEN727在不同SARS-CoV-2变体中的有效性,图3C显示,GEN727在所有VOCs中对含尖峰蛋白的伪型態病毒进行中和,IC50值介于0.7μM到2.8μM之间。活病毒数据也显示,IC50都小于50μM(图3D)。
全新设计的尖峰蛋白抑制剂针对RBD中的保守脂质结合口袋
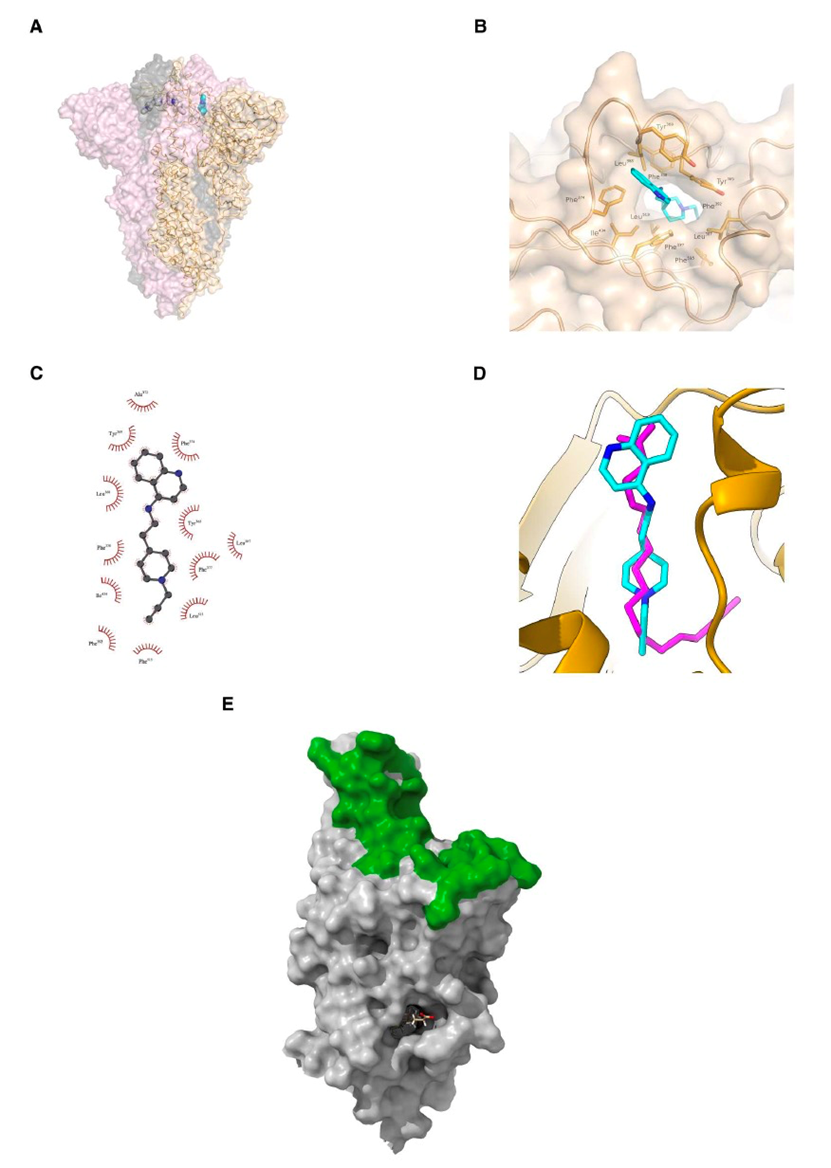
图 4
值得注意的是,当前无法获得使用全新设计的抑制剂与RBD的详细结合位姿数据。因此,作者使用体外模拟来提供对可能的结合模式的见解。首先在尖峰蛋白RBD存在的情况下对生成的分子进行了对接模拟((PDB)ID:7Z3Z)。图4显示GEN727与RBD中的几个酪氨酸和疏水性残基接触,如Tyr365、Tyr369和Phe374。对接模拟还揭示了GEN725与GEN727类似的相互作用模式。这些酪氨酸和苯丙氨酸构成了尖峰蛋白RBD的脂质结合口袋。该口袋与绝大多数中和抗体的结合位点相距较远且不同,这些抗体在ACE2结合位点聚集(请参见图4E)。
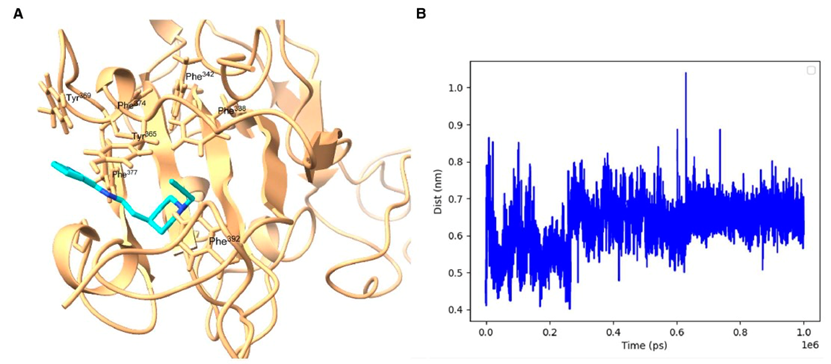
图 5
GEN727的对接模拟显著重现了天然脂质的结合(请参见图4D),表明脂质结合功能维持了GEN727靶向的保守位点。自分子动力学(MD)模拟进一步了解GEN727与脂质结合位点的结合。图5显示了在1微秒的模拟时间内,GEN727与脂质结合口袋中的保守残基之间的稳定相互作用。一致地,从MD模拟估计得到的GEN727-RBD总相互作用能为-46.68 ± 0.64 kcal/mol。
结论
此项研究展示了一个基于深度生成基础模型的单一统一抑制剂设计框架在不同靶蛋白上的实际应用性。生成框架仅需要更易获取的目标序列信息来指导设计。基于大规模的化学分子、蛋白质序列和蛋白质-配体结合数据构建的深度生成框架,作为目标感知抑制剂分子设计的生成基础模型,无需对目标特异性数据进行进一步的微调,并且可以外推到原始训练数据中不存在的目标序列。因此,CogMol框架的这种广泛适用性将其置于新兴的“基础模型”类别中,这些模型在广泛的无标签数据上进行了预训练,并可以在最小的微调下用于不同的下游任务。
参考资料
Vijil Chenthamarakshan et al. ,Accelerating drug target inhibitor discovery with a deep generative foundation model.Sci. Adv.9,eadg7865(2023).
DOI:10.1126/sciadv.adg7865



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


