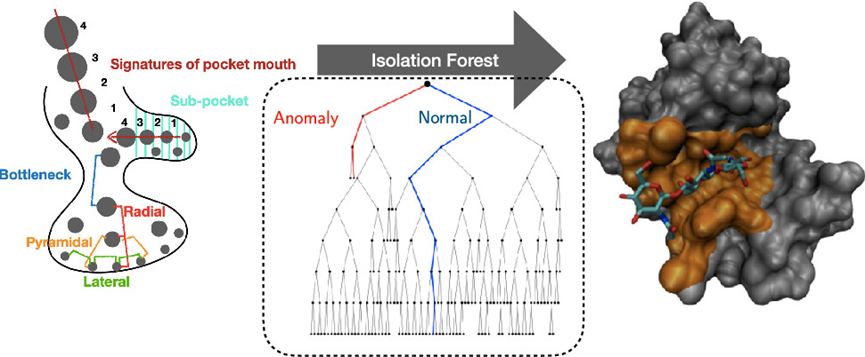
 与生理过程相关的蛋白质活性位点是药物开发的主要目标。为此,已经发展了多种蛋白口袋的预测方法,包括基于进化和模板的方法、基于能量的方法和基于几何的方法。然而,如何准确地预测结合位点仍然是一项科学挑战。最近,意大利CONCEPT实验室的Walter Rocchia教授团队推出了一种名为SiteFerret的创新算法 。该算法将基于几何的预测方法与基于机器学习的独特排序策略相融合,在口袋预测方面取得了出色的通用性,能够更好的预测其他方法难以识别的位点,例如多肽结合口袋与浅口袋。相关研究工作发表于美国化学会出版的理论化学与计算化学核心期刊Journal of
与生理过程相关的蛋白质活性位点是药物开发的主要目标。为此,已经发展了多种蛋白口袋的预测方法,包括基于进化和模板的方法、基于能量的方法和基于几何的方法。然而,如何准确地预测结合位点仍然是一项科学挑战。最近,意大利CONCEPT实验室的Walter Rocchia教授团队推出了一种名为SiteFerret的创新算法 。该算法将基于几何的预测方法与基于机器学习的独特排序策略相融合,在口袋预测方面取得了出色的通用性,能够更好的预测其他方法难以识别的位点,例如多肽结合口袋与浅口袋。相关研究工作发表于美国化学会出版的理论化学与计算化学核心期刊Journal ofChemical Theory and Computation (J. Chem. Theory Comput. 2023, 19, 15, 5242–5259)【1】。 SiteFerret算法的工作流程如图-1所示,首先运用NanoShaper软件为蛋白计算溶剂排斥表面(SES),过程中会生成不同半径的探针球体,通过对SES凹陷区域的探针球体进行特定的聚类就可以获得候选口袋。同时,SiteFerret为每个候选口袋计算几何、聚类与化学方面的特征,包括口袋体积、入口数、聚类打分、氨基酸组成、疏水性分数等。这些特征信息将输入一个Isolation Forest(IF)算法来为口袋进行打分与排序。IF是一种经典的无监督学习方法,与传统的二分类方法相比,IF可以输出更为准确的口袋评估结果。
 图-1:SiteFerret算法的蛋白口袋预测流程
在算法性能测试阶段,研究者使用了多个专业数据集来深入评估SiteFerret在蛋白口袋预测方面的表现。首先在Binding MOAD数据集上,虽然SiteFerret预测的Top1与Top3对真实口袋的命中率不如Fpocket与NS-Volume算法,但在Top10中的表现超过了这两种知名算法。随后,研究者进一步使用业内广泛认可的LIGSITE-PocketPicker数据集进行算法评估。该数据集包含的结构更为多样,涵盖了复合体(holo)蛋白结构与无配体结合的Apo蛋白结构。在这一数据集上,SiteFerret的预测表现较为出色,特别是与Fpocket进行对比时,它在多个指标上都显示出更高的准确性。
图-1:SiteFerret算法的蛋白口袋预测流程
在算法性能测试阶段,研究者使用了多个专业数据集来深入评估SiteFerret在蛋白口袋预测方面的表现。首先在Binding MOAD数据集上,虽然SiteFerret预测的Top1与Top3对真实口袋的命中率不如Fpocket与NS-Volume算法,但在Top10中的表现超过了这两种知名算法。随后,研究者进一步使用业内广泛认可的LIGSITE-PocketPicker数据集进行算法评估。该数据集包含的结构更为多样,涵盖了复合体(holo)蛋白结构与无配体结合的Apo蛋白结构。在这一数据集上,SiteFerret的预测表现较为出色,特别是与Fpocket进行对比时,它在多个指标上都显示出更高的准确性。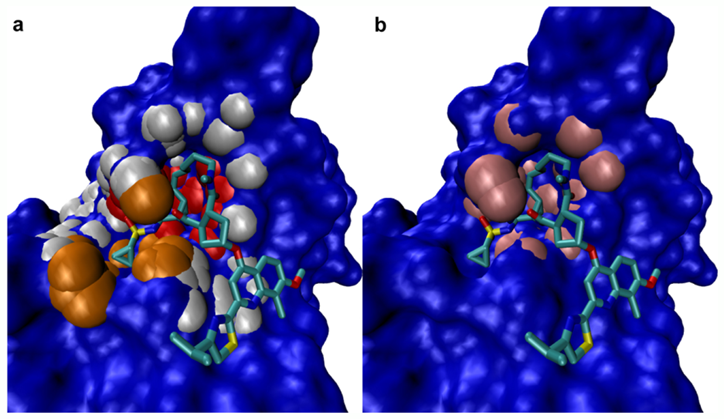 图-2:SiteFerret(a)与Fpocket(b)为HCV NS3蛋白酶的口袋预测结果。
研究者还使用了包含115个蛋白–多肽结合位点的数据集来评估算法性能。这类结合位点区别于蛋白–小分子位点,在几何与化学性质方面贴近蛋白–蛋白相互作用位点,因此在预测方面更具有挑战性。而测试结果显示,SiteFerret的预测最为精准, Top1/3/10的预测命中率均超越了Fpocket和NS-Volume两种算法。为进一步展示SiteFerret的实际应用价值,研究者提供了一些具体案例。例如,在HCV NS3蛋白酶上,SiteFerret预测的结合口袋与实际的结合位点十分吻合(图-2),而Fpocket的预测结果则不够精确。在HIV整合酶、胰岛素等蛋白上,SiteFerret可以成功预测出一些位于蛋白浅表面的结合口袋(图-3),这是其他两种算法无法预测的位点。这些发现凸显了SiteFerret在应对一些具有挑战性的蛋白口袋预测任务时的优越性。
图-2:SiteFerret(a)与Fpocket(b)为HCV NS3蛋白酶的口袋预测结果。
研究者还使用了包含115个蛋白–多肽结合位点的数据集来评估算法性能。这类结合位点区别于蛋白–小分子位点,在几何与化学性质方面贴近蛋白–蛋白相互作用位点,因此在预测方面更具有挑战性。而测试结果显示,SiteFerret的预测最为精准, Top1/3/10的预测命中率均超越了Fpocket和NS-Volume两种算法。为进一步展示SiteFerret的实际应用价值,研究者提供了一些具体案例。例如,在HCV NS3蛋白酶上,SiteFerret预测的结合口袋与实际的结合位点十分吻合(图-2),而Fpocket的预测结果则不够精确。在HIV整合酶、胰岛素等蛋白上,SiteFerret可以成功预测出一些位于蛋白浅表面的结合口袋(图-3),这是其他两种算法无法预测的位点。这些发现凸显了SiteFerret在应对一些具有挑战性的蛋白口袋预测任务时的优越性。
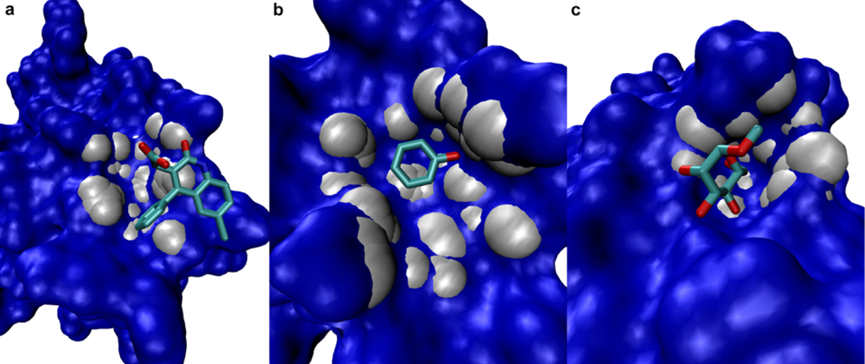 图-3:SiteFerret对一些蛋白浅口袋的成功预测案例。(a)HIV整合酶,(b)胰岛素蛋白,(c) 刀豆蛋白A
最后,为深入探究哪些特征对模型的预测过程产生了显著影响,研究者采用SHAP框架进行了特征分析。SHAP是一种基于博弈论的方法,能够为每个特征分配一个重要性值,以反映该特征在模型预测中的影响程度。结果如图-4所示,左侧部分分析了几何特征的重要性,这些特征与口袋的形状、大小和复杂性有关。其中,入口的有效半径、瓶颈数量、入口数量等特征被认为是高度重要的。图片右侧部分分析了化学特征的重要性,如CYS、MET、TRP这样的氨基酸残基在重要性上位于顶部,而其他一些如PHE则在重要性上位列底部,同时口袋疏水性的重要性又高于亲水性。
图-3:SiteFerret对一些蛋白浅口袋的成功预测案例。(a)HIV整合酶,(b)胰岛素蛋白,(c) 刀豆蛋白A
最后,为深入探究哪些特征对模型的预测过程产生了显著影响,研究者采用SHAP框架进行了特征分析。SHAP是一种基于博弈论的方法,能够为每个特征分配一个重要性值,以反映该特征在模型预测中的影响程度。结果如图-4所示,左侧部分分析了几何特征的重要性,这些特征与口袋的形状、大小和复杂性有关。其中,入口的有效半径、瓶颈数量、入口数量等特征被认为是高度重要的。图片右侧部分分析了化学特征的重要性,如CYS、MET、TRP这样的氨基酸残基在重要性上位于顶部,而其他一些如PHE则在重要性上位列底部,同时口袋疏水性的重要性又高于亲水性。
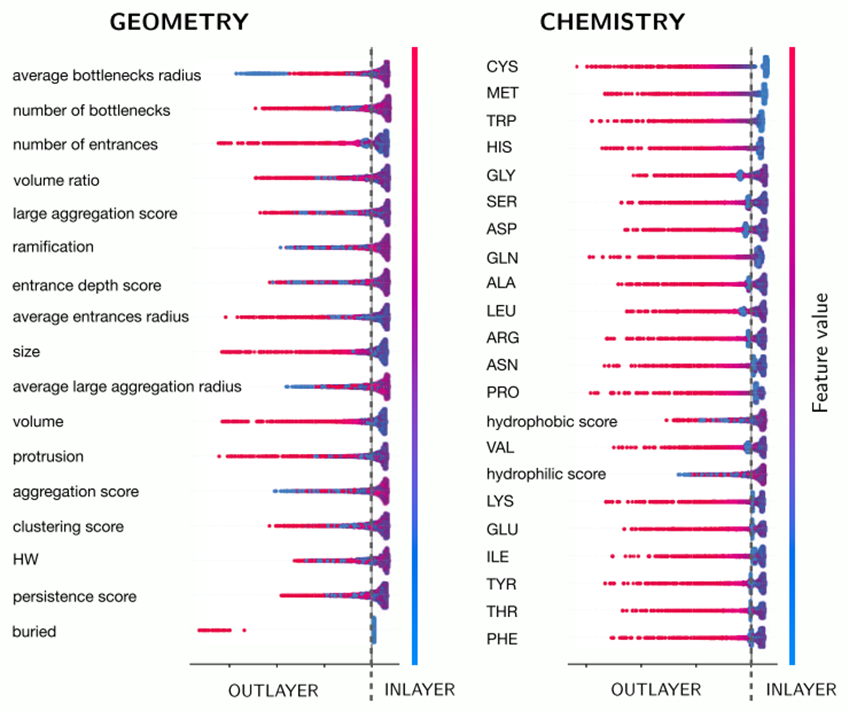 图-4: 基于SHAP的特征重要性分析结果。重要性由上至下排序,每项特征的数值用红(大)/蓝(小)颜色表示,虚线右侧表示可能的结合口袋,虚线左侧表示可能性较低的候选口袋。
【小编评论】研究者在这项工作中推出了一种名为SiteFerret的新型蛋白口袋预测算法。通过综合考虑蛋白口袋的几何、聚类和化学信息,该算法在多个专业数据集上都展现出了较好的预测能力,具有出色的通用性。然而,从实际应用角度来看,小编认为SiteFerret更适用于预测多肽结合位点和蛋白浅位点这样的特定任务。因为它预测小分子结合口袋的能力与顶级算法之间仍存在一定的差距。并且,SiteFerret的预测效率也有待提升,它需要输出多个预测口袋才能较好地找到真实位点,实际应用的时候会导致后续模拟和分析工作的复杂性增加。期待该算法未来的进一步优化和迭代。
参考文献
Gagliardi,
图-4: 基于SHAP的特征重要性分析结果。重要性由上至下排序,每项特征的数值用红(大)/蓝(小)颜色表示,虚线右侧表示可能的结合口袋,虚线左侧表示可能性较低的候选口袋。
【小编评论】研究者在这项工作中推出了一种名为SiteFerret的新型蛋白口袋预测算法。通过综合考虑蛋白口袋的几何、聚类和化学信息,该算法在多个专业数据集上都展现出了较好的预测能力,具有出色的通用性。然而,从实际应用角度来看,小编认为SiteFerret更适用于预测多肽结合位点和蛋白浅位点这样的特定任务。因为它预测小分子结合口袋的能力与顶级算法之间仍存在一定的差距。并且,SiteFerret的预测效率也有待提升,它需要输出多个预测口袋才能较好地找到真实位点,实际应用的时候会导致后续模拟和分析工作的复杂性增加。期待该算法未来的进一步优化和迭代。
参考文献
Gagliardi,L.; Rocchia, W. SiteFerret: Beyond Simple Pocket Identification in Proteins. Journal
of Chemical Theory and Computation 2023, 19 (15), 5242-5259.
正文完
可以使用微信扫码关注公众号(ID:xzluomor)



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


