原文作者:Ewen Callaway
“好,现在开始。”美国华盛顿大学计算化学家David Juergens正打算设计一种过去30多亿年的演化进程中从未出现过的蛋白质。
在今早的一次视频通话中,Juergens打开了他协助开发的人工智能(AI)工具RFdiffusion的云版本。这个神经网络和其他同类工具正在将定制蛋白质带入主流科研界——就在不久前,定制蛋白质还有很高的技术门槛而且常以失败告终。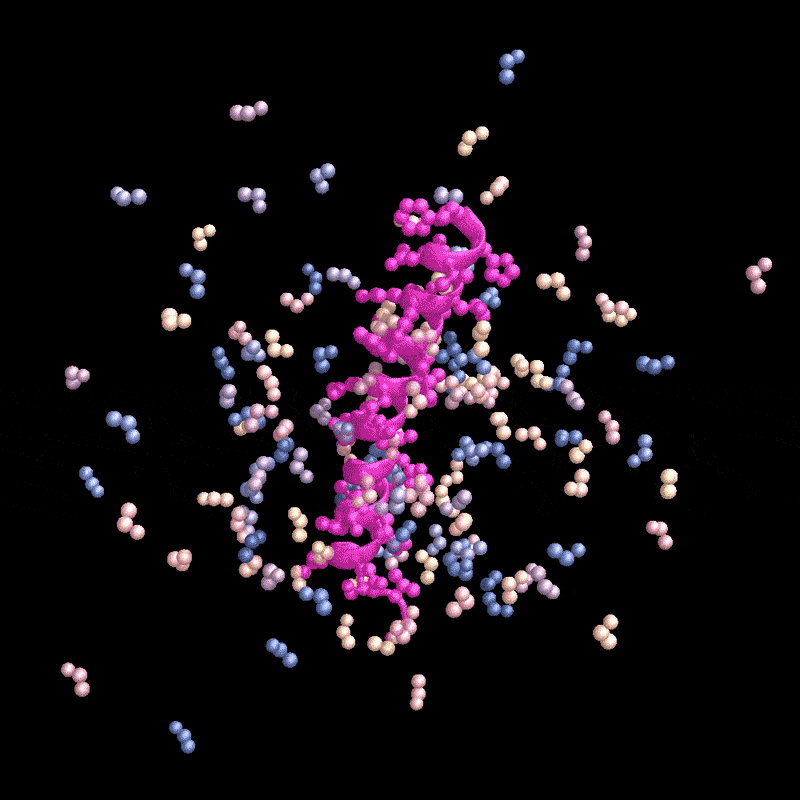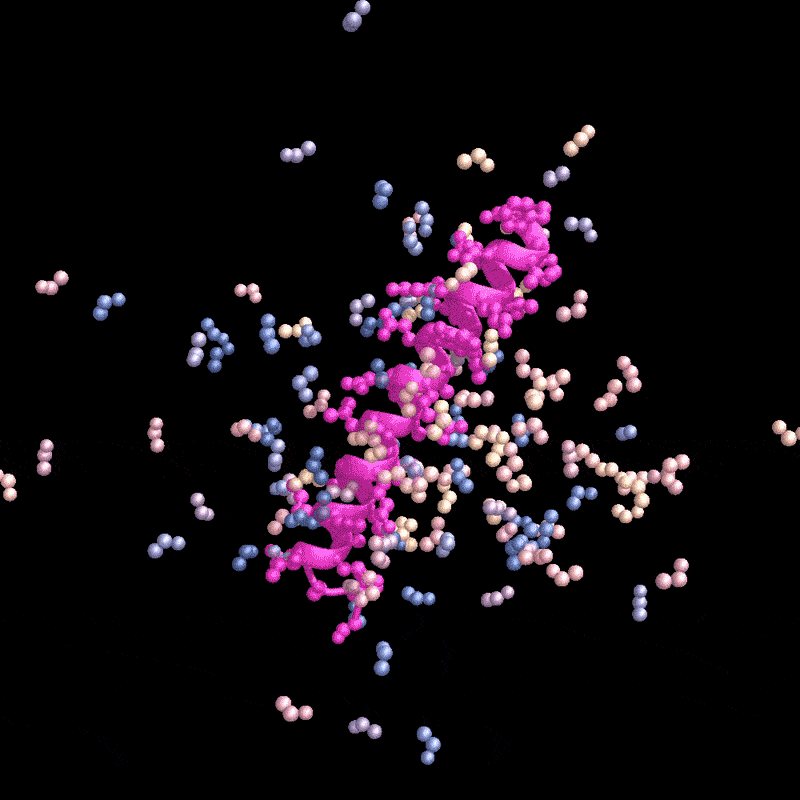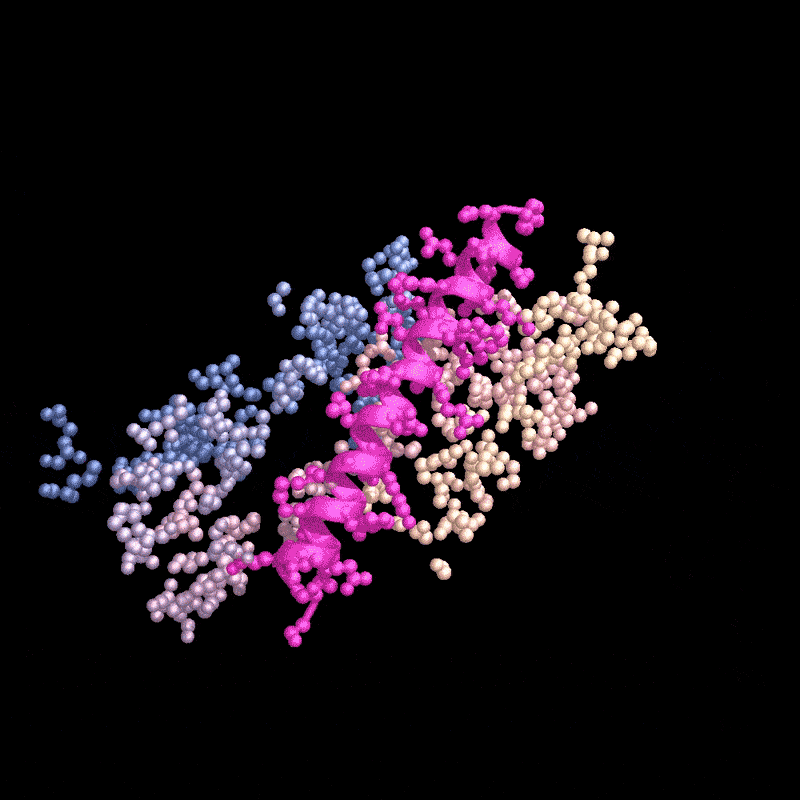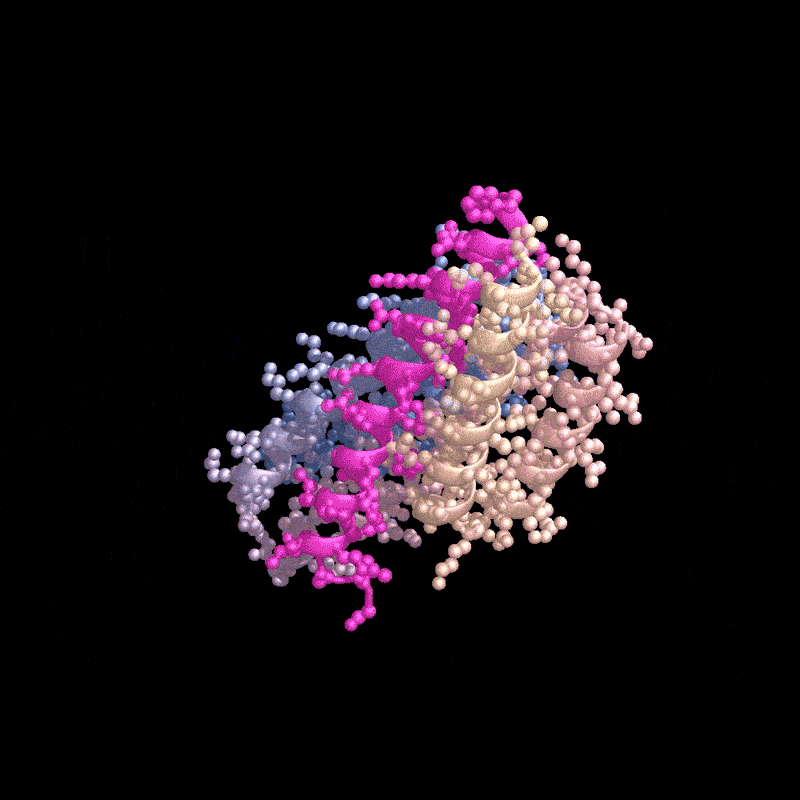
名为RFdiffusion的AI工具设计了一种能与甲状旁腺激素(粉色)结合的蛋白质。来源:Ian C. Haydon/UW Institute for Protein Design
这些蛋白质或能成为疫苗、疗法和生物材料的基础。“这绝对是颠覆性时刻。”美国麻萨诸塞州生物科技公司Generate Biomedicines的首席技术官Gevorg Grigoryan说。这家生物科技公司试图将蛋白质设计运用到药物研发中。
这些工具的灵感来自能合成逼真图像的AI软件,如今年因为合成了教皇穿着设计师款白色羽绒服的照片而走红的Midjourney。研究人员发现,类似概念能用来生成符合设计者要求的逼真的蛋白质形状,也就是说,现在可以快速设计出与另一个生物分子紧密结合的新蛋白质。而早期实验显示,当研究人员合成这些蛋白时,其中一小部分有用的蛋白质和软件预测的表现一样。
研究人员说,过去一年里,这些工具颠覆了蛋白质的设计过程。“能力一下子突飞猛进”,纽约哥伦比亚大学计算生物学家Mohammed AlQuraishi说道。他的团队开发了一款蛋白质设计工具。“你现在可以设计出具有你想要功能的蛋白质了。”
华盛顿大学计算生物物理学家David Baker说:“你在为某个问题定制一种蛋白质结构。”Baker的团队开发了RFdiffusion,Juergens也是该团队成员。该团队在2023年3月发布了这款软件,并在《自然》发表了描述该神经网络的论文[1]。(该论文的预印本于2022年末发布,当时AlQuraishi[2]和Grigoryan团队在内的其他多个团队都报道了类似的神经网络)。
这是蛋白质设计者首次拥有了这些可重复和可靠的工具,并能用它们打造一个全新的产业,Grigoryan说,“接下来的挑战是,你可以用它做什么?”
宏大设计
Juergens在一个类似在线税务计算器的web表单中输入了他想要的一些蛋白质的特性。它必须有100个氨基酸长,能形成名为同二聚体的有两个蛋白组成的对称复合物结构。许多细胞受体都是这种构型,而一个新的同二聚体或能作为合成的细胞信号传导分子,华盛顿大学计算生物化学家Joe Watson说道。Watson协助开发了RFdiffusion,也参与了今天的视频通话。但今早的设计只是为了模拟一个逼真的蛋白质,并无其他目的。
几十年来,研究人员在设计新蛋白质的道路上艰难前行着。最初,他们尝试拼凑现有蛋白的有用部分,像是一个酶中能催化化学反应的某个部分。这种方法需要理解蛋白质的折叠和工作原理,还依赖直觉和许多试错。科学家有时候需要筛选几千种设计,才能找到一个符合预期的结构。
Baker说,能根据氨基酸序列准确预测蛋白质结构的AlphaFold(由伦敦的AI公司DeepMind开发,现为Google DeepMind)和其他AI模型的出现带来了曙光。设计者发现,这些用真实蛋白质序列和结构训练的神经网络还能用来从头创造蛋白质。
过去几年里,Baker的团队还有其他团队发布了多款基于AI的蛋白质设计工具。这些工具使用的一种方法名为“幻觉”(hallucination),这需要创造一串随机的氨基酸链,再由AlphaFold或RoseTTAFold这种类似工具进行优化,直到它形成这种神经网络认为能折叠成特定结构的东西。另一种方法名为“修复”(inpainting),这种方法利用一段特定的蛋白质序列或结构,并利用RoseTTAFold在它周围构建分子的剩余部分。
不过,这些工具都还没达到完美。实验显示,“幻觉”方法设计出的结构在实验室中合成时,并不总是能形成折叠到位的结构,最后变成试管底部的一堆物质。“幻觉”方法除了小蛋白以外也很难生成其他产物(虽然其他研究人员在2月的一篇预印本论文里指出,这种方法或能用来设计更长的分子[4])。“修复”方法在利用短片段形成蛋白质方面也差强人意。虽然这种方法确实能得到理论上的蛋白质结构,但它无法就某个问题给出不同的解决办法,从而提高成功率。
这时就要用到近几个月里发布的RFdiffusion和类似的蛋白质设计AI工具了。它们和合成逼真图像的神经系统如Stable Diffusion、DALL-E、Midjourney遵循相同的原理。这些“扩散”(diffusion)网络用数据进行训练,无论数据是图像还是蛋白质结构,训练过程都会“加噪”,最后的结果与开始的图像或结构完全没有相似性。随后这个网络会学习给数据“去噪”,反向执行任务。
RFdiffusion这类网络使用蛋白质数据库(PDB)里储存的上万种真实蛋白质结构进行训练。当该网络设计了一种新蛋白时,它会从总的噪声开始:氨基酸的随机组合。Watson说:“你想知道产生这些噪声的蛋白是什么。”经过几轮去噪,它就会产生很真实但其实是全新的蛋白质。
Baker团队在测试RFdiffusion时除了蛋白质长度以外没给任何指示,这个网络给出了看起来很逼真的各种蛋白质,与用PDB训练出来的全不一样。
但是,该团队还能指导程序在去噪过程中根据特定设计要求来设计蛋白质,这个过程名为“条件反射”(conditioning)。
比如,Baker的团队让 RFdiffusion设计有特殊折叠结构或是能附在另一个分子表面(结合背后的互作机制)的蛋白质。Grigoryan的团队还开发了一个名为 Chroma的扩散网络,通过条件反射让它设计形似26个英语大写字母和阿拉伯数字的蛋白质[3]。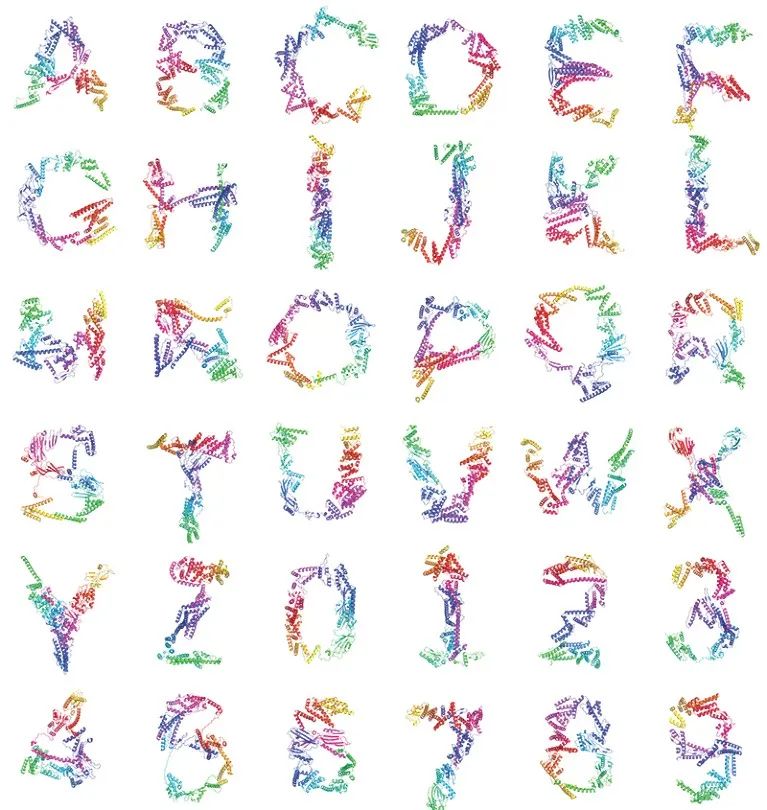
AI设计出了形似英文字母表的蛋白质。来源:John Ingraham, Wujie Wang, Max Baranov, Gevorg Grigoryan
噪声信号
Juergens的计算机屏幕一开始显示有噪声,也就是AI系统会从各种随机的氨基酸序列开始。它们是一些红色杂乱的线条,很像小孩的手指画。它们还会逐帧变换成更复杂的结构,拥有类似蛋白质的特征,比如名为α螺旋的紧密螺旋结构,以及名为β-折叠的反向排列的丝状结构。“这是α-β拓扑学的完美结合,”Juergens一边看着只用了几分钟就设计好的蛋白一边说,“看起来很好。”
Baker的实验室里都在使用这个工具。他说:“整个设计过程与一年之前大相径庭。”这个神经网络可以出色完成其他方法做起来很低效、很困难或不成功的任务。
该研究报道的一项分析中[1],研究团队一开始用了另一个蛋白的片段,如免疫细胞能识别的一个病毒蛋白的一部分,并让AI工具产生100个不同的新蛋白,来看看有多少能包含他们想要的基序。团队用25个不同的初始形状挑战了这个任务。最后的结果并不总是包含一开始的片段,但RFdiffusion能产生至少一个有23个目标基序的蛋白,相比之下,“幻觉”方法只有15个,“修复”方法只有12个。
RFdiffusion还被证明能设计出自组装成复杂纳米粒子的蛋白,这些纳米粒子能递送药物或疫苗成分。之前的AI方法[5]也能设计出这种蛋白质,但Watson认为RFdiffusion的设计更加精妙。
RFdiffusion一类的神经网络似乎很善于设计能与另一种特定蛋白质结合的蛋白质。Baker的团队用这个网络设计出了能与癌症和自身免疫疾病等疾病中的蛋白紧密结合的蛋白质。他说,在一项尚未发表的成功实验中,他们为一个很难靶向的免疫信号传导分子设计了能紧密结合的蛋白——针对该靶点的抗体药物每年能产生数十亿美元的收益。Watson说:“它扩大了我们能找到结合蛋白并开发有用疗法的蛋白质的范围。”
真实世界测试
由于Baker团队设计了许多蛋白质,测试它们是否具有预期功能便成了一项艰巨任务。“一个机器学习人设计出的蛋白质能让100名生物学家忙上好几个月。”马萨诸塞州微软研究院(Microsoft Research) 的生物医学机器学习研究院Kevin Yang说[6]。
但早期迹象显示,RFdiffusion的设计是“货真价实”的。在他们描述的另一项任务中,Baker团队用这个工具设计了含有p53关键片段的蛋白质,p53是一个信号传导分子,在许多癌症中过度活跃(同时也是一个极具吸引力的药物靶点)。当该团队合成该软件设计的95个蛋白质时(通过使工程改造细菌表达这些蛋白),超过一半都具备p53与其天然靶点MDM2结合的能力。最好的设计比天然的p53的结合力度强了1000多倍。Watson说,当研究人员使用“幻觉”方法时,设计结果虽然在预测中能成功,但实际在试管中却没有。
总体上,Baker说他的团队发现RFdiffusion有10-20%的设计能与预期靶标紧密结合,产生有用的效果,而使用AI之前的方法只有不到1%的成功率。(Watson说,之前的机器学习方法无法可靠地设计结合蛋白)。华盛顿大学的同事、生物化学家Matthias Gloegl说他最近的成功率都接近50%,意味着可能只要一两周而不是几个月的时间就能得到有用的设计。他说:“这有点疯狂。”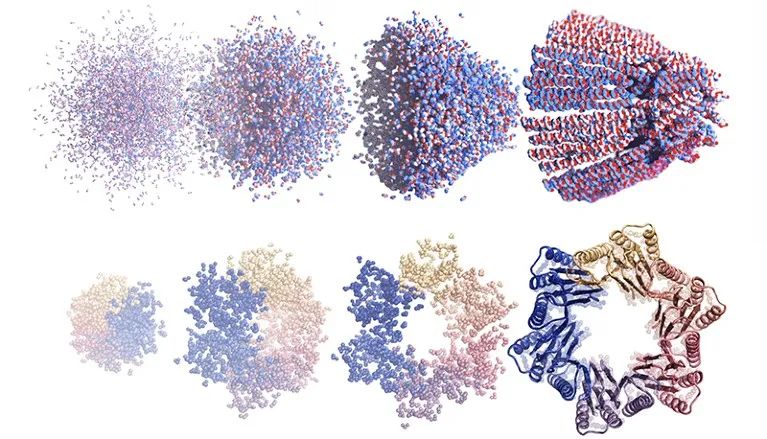
基于扩散的AI绘图生成器利用噪声设计的一种漏斗形蛋白质组装(上排)和一种有6个蛋白质链(下排)的环形结构。来源:Ian C. Haydon/UW Institute for Protein Design
哈佛大学演化生物学家Sergey Ovchinnikov说,到6月底,RFdiffusion的云版本每天约有100名用户。澳大利亚悉尼大学的生物化学家Joel Mackay尝试让RFdiffusion设计能与他实验室研究的其他蛋白质结合的蛋白,包括细胞内控制基因活性的转录因子。他发现整个设计过程很简洁,并用计算机建模证实了这些蛋白理论上可以和这些转录因子结合。
Mackay正在测试这些蛋白质在细胞内产生时是否能按预期改变基因表达。他希望一切顺利,因为这类发现能以简单的方式开关细胞内的特异性转录因子,这样就不需要借助于研发周期动辄几年的药物——前提是这些药物真的能被开发出来。他说:“如果这种方法对我们的蛋白质有效,那它简直就是‘变局者’。”
未来优化
英国牛津大学免疫信息学家Charlotte Deane说,RFdiffusion这类最新模型是“一次质变”。但关键问题仍未解决。她说:“它能做的是让人们看到这些扩散模型的潜力。”
她和其他科学家还有生物科技公司都很感兴趣的一个应用是设计更复杂的结合蛋白,比如抗体,或是T细胞(一类免疫细胞)使用的蛋白受体。这些蛋白质有很灵活的卷曲结构,能与它们的目标互锁,而RFdiffusion目前最擅长类似三明治的扁平界面。Baker表示他们在抗体方面已经取得了进展。
Ovchinnikov等人认为,总体上,对于功能取决于松散区域折叠成不同形状的生物分子来说,这类设计难度比较大。这些特征已被证明很难用AI来模拟。“如果你的问题是是否能与其他东西结合并抑制它,”Ovchinnikov说,“那么问题是可以用这些方法解决的。但如果要完成更复杂的任务,比如模拟自然现象,你就要加入一些灵活性。”
加州大学旧金山分校的计算生物学家Tanja Kortemme正在用 RFdiffusion设计能作为传感器或细胞控制开关的蛋白质。她说,如果一个蛋白质的活性位点取决于少数氨基酸的排列,这个AI网络就能表现得很好,但它很难设计出活性位点更复杂的蛋白质,这需要更多的关键氨基酸,这也是她和同事正在努力解决的问题。
最新扩散模型的另一个局限在于它们无法设计出与天然蛋白质截然不同的蛋白质,Yang说。因为这种AI系统都是用科研人员表征过的现有蛋白质训练的,他说,所以它们设计的蛋白质都是“照葫芦画瓢”。设计外观异常的蛋白质可能要求我们进一步理解赋予蛋白质其功能的物理机制。
Yang说:“这样更容易设计出能执行天然蛋白质不能完成任务的蛋白质。今后还有很多发展空间。”
最新的蛋白质设计工具已被证明在针对特定任务设计蛋白质时非常强大,只要功能能以结构的形式表述,比如蛋白质结合的表面,AlQuraishi说。但他强调,RFdiffusion这类工具还无法处理其他特征,比如设计一种无论什么形状都能完成特定反应的蛋白质——虽然你知道你想要什么但你不知道它的几何构造应该是怎样的。
Grigoryan说,未来的蛋白质设计工具还需要具备能根据各种需求设计蛋白质的能力。潜在的治疗性蛋白质不仅需要和其靶标结合,还要不与其他对象结合,而且能轻松量产。
研究人员正在探索的一个方向是是否能用直白的语言来描述这些蛋白质的设计,就和给Midjourney这类图像生成工具的提示差不多。Watson说:“你可以想象我们把某种蛋白质的描述写出来,然后让它们合成出来再测试。”
Grigoryan和同事已经朝这个目标迈进了一步。在他们2022年12月发布的一篇预印本论文[3]中,他们训练Chroma 把描述融入设计中,并根据文本描述给出设计,包括‘有CHAD结构域的蛋白’(结合了多种螺旋的蛋白结构)或是‘氨基转移酶的晶体结构’(参与合成和分解蛋白的酶)。
Juergens今天早上花了几分钟设计的蛋白质只是一个蛋白质3D结构的模型。Juergens随后又用另一种AI工具得到了能折叠成该结构的氨基酸序列。在最后检查中,他将序列接入AlphaFold,看看该软件是否能预测出与设计相符的折叠结构。结果完全相符,AlphaFold的预测与设计结构平均只有1埃(一个氢原子的宽度)的差距。
Watson说:“这样的准确率我们可称之为设计成功了。”接下来要做的唯一事情是看看这个蛋白在现实中的表现。
参考文献:
1. Watson, J. L. et al. Nature https://doi.org/10.1038/s41586-023-06415-8 (2023).
2. Lin, Y. & AlQuraishi, M. Preprint at https://arxiv.org/abs/2301.12485 (2023).
3. Ingraham, J. et al. Preprint at bioRxiv https://doi.org/10.1101/2022.12.01.518682 (2022).
4. Frank, C. et al. Preprint at bioRxiv https://doi.org/10.1101/2023.02.24.529906 (2023).
5. Wicky, B. I. M. et al. Science378, 56–61 (2022).
6. Wu, K. E. Preprint at https://arxiv.org/abs/2209.15611 (2022).
原文以AI tools are designing entirely new proteins that could transform medicine标题发表在2023年7月11日《自然》的新闻特写版块上
© nature
doi: 10.1038/d41586-023-02227-y
版权声明:
本文由施普林格·自然上海办公室负责翻译。中文内容仅供参考,一切内容以英文原版为准。欢迎转发至朋友圈,如需转载,请邮件China@nature.com。未经授权的翻译是侵权行为,版权方将保留追究法律责任的权利。
© 2023 Springer Nature Limited. All Rights Reserved
|点击关注我 👇 记得标星|



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


