编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Michel F. Sanner团队的一篇论文。深度学习(DL)方法在预测蛋白质结构方面取得的准确性进展,以及它对结构生物学产生了深远影响。AlphaFold2是一个DL方法,已经在预测蛋白质-肽相互作用方面进行了评估,结果显示其性能显著优于RoseTTAfold和传统的对接方法PIPER-FlexPepDock。随后,新的AlphaFold2模型专门用于预测多聚体组装,此外新的从头开始折叠模型OmegaFold也已经发布。作者评估了这些新的DL折叠模型在对接蛋白质-肽相互作用时的成功率,并将其与他们的最新专注对接软件AutoDock CrankPep (ADCP) 进行了比较。评估使用相同的数据集和性能度量标准来进行。

蛋白质与肽的相互作用对许多生物功能至关重要,据估计,15-40%的细胞内蛋白质相互作用是通过这种相互作用介导的。许多蛋白质-蛋白质相互作用,尤其是涉及内在无序蛋白质(IDPs)的相互作用,是通过肽片段介导的。治疗性肽(TPs)已经使用了一个世纪,最近对肽的新合成策略正在减轻与TPs相关的缺陷。环化、包括非标准氨基酸,特别是D-对映体,以及结合基团可以减少蛋白酶敏感性,延长半衰期。这些进展使得TPs在近年来经历了复兴,目前已经有80种基于肽的药物在主要市场上获得批准,还有400至600种肽处于临床开发中。此外,TPs还允许作用于历史上占主导地位的细胞外激素受体以外的受体。已经进入临床的例子包括针对受体酪氨酸激酶的肽、抑制细胞内靶标的肽以及干扰其他蛋白质-蛋白质相互作用的肽。氨基酸数在2到约20之间的寡肽位于小分子药物和大型治疗性分子(如抗体和蛋白质)之间。从生物化学和治疗学上看,TPs与两者都有所不同,它们构成了一类具有有趣特性的药物,包括高度特异性和亲和性、几乎没有毒性,以及模拟或干扰蛋白质-蛋白质相互作用的能力。了解肽与其他生物大分子相关的结构细节,有助于确定结构活性关系,并设计和优化新的治疗性肽。然而,由于蛋白质-肽相互作用通常具有高度瞬时的性质,因此实验解析这些结构具有挑战性。因此,在近年来研究人员开发了几种计算方法来预测蛋白质-肽复合物。这些方法根据是否已知结合位点分为有针对性和无针对性对接类别,作者最近开发了AutoDock CrankPep,这是一种有针对性的肽对接方法,被认为是有针对性肽对接的最新方法。尽管它们的评分函数存在缺陷,而且与小分子对接相比,抽样问题更为复杂,但目前的肽对接方法通常可以在前10到100个解中识别出正确的构象,但仅在考虑靠前排名的对接构象时,成功率才达到约20%。
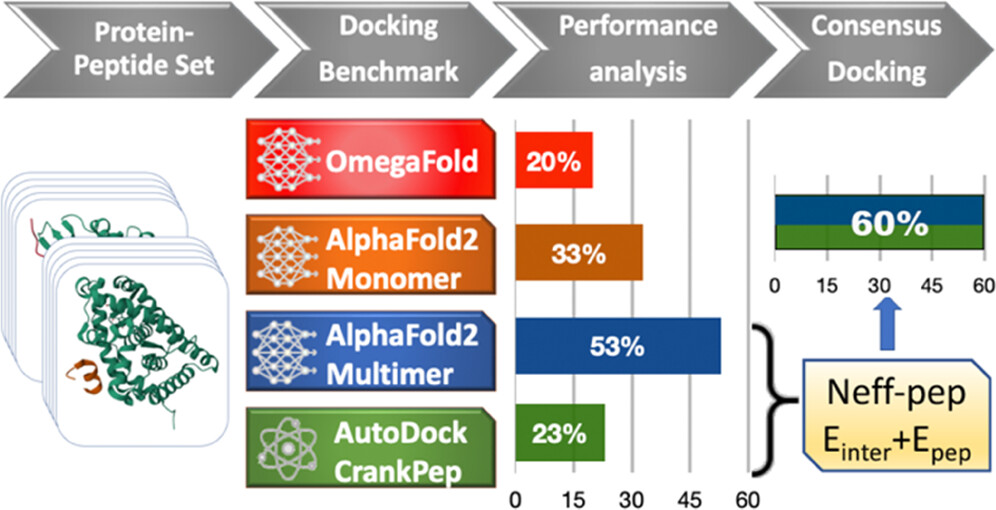
AlphaFold2是一种旨在预测单体蛋白质结构的技术,因此被称为AlphaFold2 monomer。最近研究表明,AlphaFold2 monomer在预测蛋白质-肽复合物方面表现出令人惊讶的优异性能。在这项工作中,研究人员使用AlphaFold2 monomer模型来折叠一个由蛋白质和肽序列组成的单链,它们之间由一个30个氨基酸长的聚甘氨酸链连接。下文中称此方法为AF2mono。对于一个非冗余的96个复合物集合,这种方法在2.5 Å的肽骨架RMSD内预测了一半的复合物,超过了肽对接软件程序PIPER FlexPepDock的性能。最近,一种基于分子动力学的方法显示出在线性肽与大分子相结合的九个复合物和循环肽与大分子相结合的三个复合物方面可以获得更高的准确性和成功率。尽管这种方法在盲目对接和有针对性对接方法方面超越了最先进的方法,即AF2mono和ADCP,但小数据集和有意选择相对较小的大分子提示了这些计算可能存在较高的计算成本,这可能限制了其实际应用。最近,新的AlphaFold2模型,称为AlphaFold2 multimer,已经发布。这些模型依赖于与早期AF2mono模型相同的深度神经网络架构,但专门用于预测已知化学计量学的多聚体组装。此外,一种称为OmegaFold的新型深度学习方法已经可用。OmegaFold执行真正的从头开始折叠,而无需使用多序列比对(MSAs)。
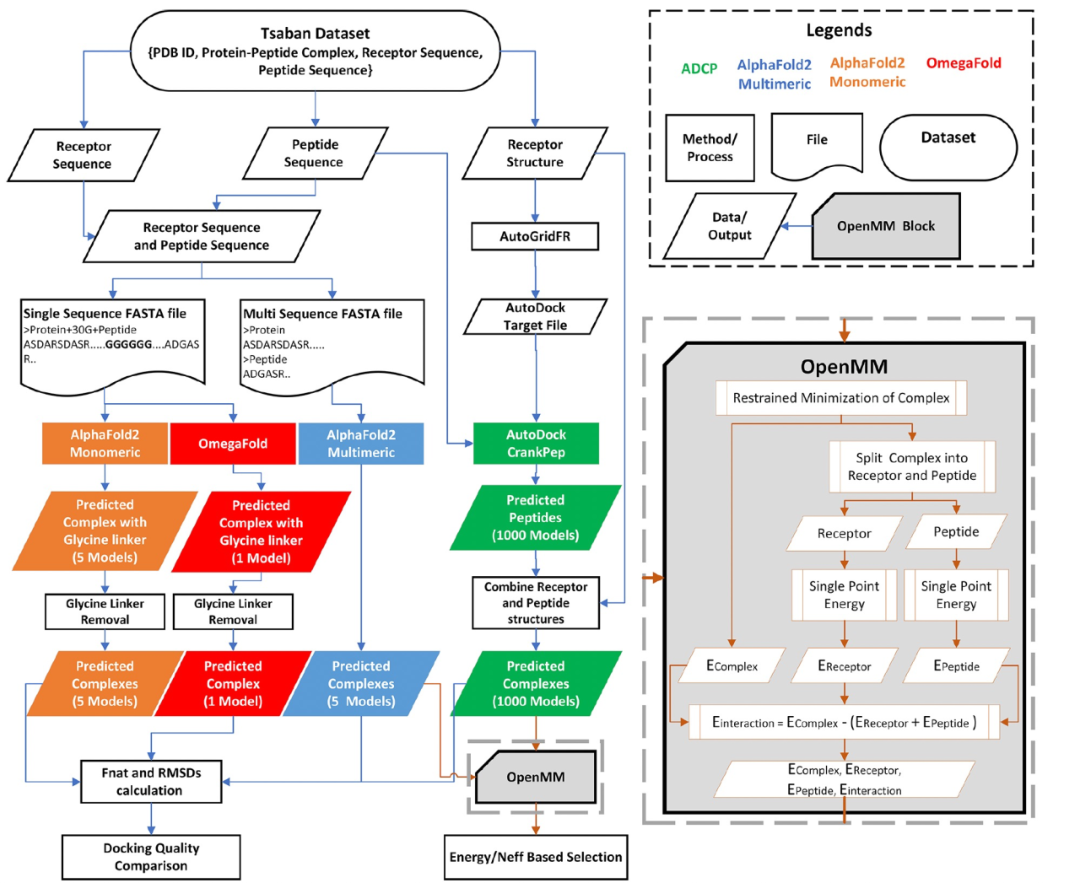
图1
Tsaban等研究人员报告了AF2mono优于DL方法RoseTTAFold以及他们的传统盲肽对接方法Piper FlexPepDock(PFPD),使其成为一种最先进的肽对接方法。在这里,作者将AF2mono与较新的AF2multi和OmegaFold(OF)DL方法以及传统有针对性的肽对接方法ADCP进行比较,以提供对DL和卷积肽对接方法在已知结合位点的情况下的比较。作者提供了这些方法准确预测蛋白质-肽相互作用能力的比较,基于一组99个蛋白质-肽复合物,并使用相同的度量标准评估了所有方法的对接成功情况。图1描述了执行此比较的工作流程。作者的结果表明,尽管OF在肽对接方面表现不佳,但AF2multi改进了AF2mono和ADCP的性能。作者发现,为每个AF2multi模型生成多个姿势只会略微改善性能,因为AF2模型往往会收敛到一组相似的解决方案。与此相反,ADCP实现了卓越的采样方法,通常在其前100个解决方案中报告了正确的解决方案;然而有个问题它的当前评分功能未将这些解决方案报告为最佳解决方案。此外,虽然AF2multi可以对接长肽,但它仅限于包含标准氨基酸的线性肽,严重限制了其在治疗性肽中的适用性。另一方面,ADCP只能有效用于具有少于30个氨基酸的肽,但可以对接常见于治疗性肽中的具有化学修饰的肽,包括环化肽、非标准氨基酸和D-氨基酸。作者展示了对于既可以使用AF2multi又可以使用ADCP对接的肽,可以通过共识对接方法将对接结果组合起来,从而通过共识对接方法实现了较高的成功率。作者描述了一种方法,该方法可以有效地在这两种方法产生的最高排名的姿势之间进行选择,为作者的数据集实现了令人瞩目的60%的共识对接成功率。总之,这些结果说明了AF2multi和ADCP是高度互补的方法,可以一起使用,以实现更高的对接成功率,接近小分子对接方法所享受的成功率。
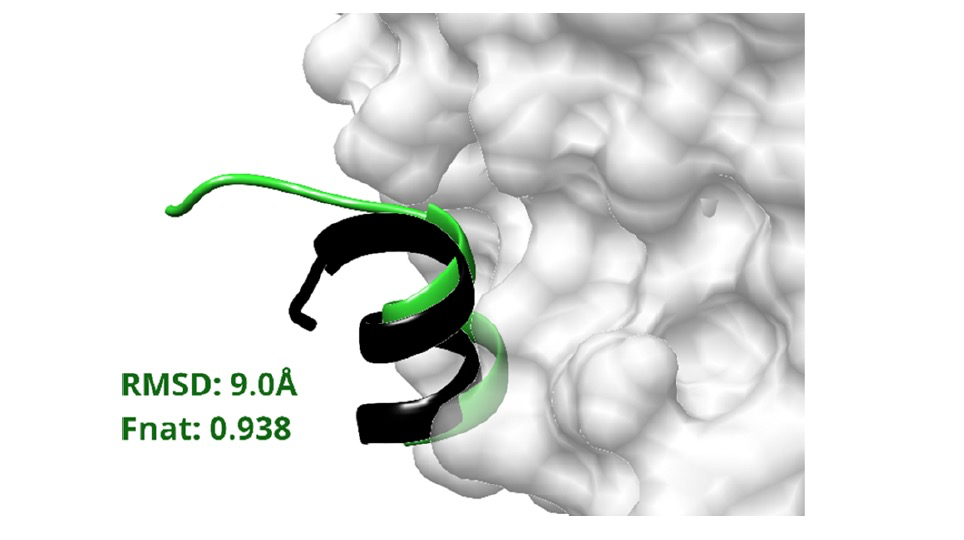
图 2
通常情况下,对于小分子的结合,我们希望它与大分子的结合模式在Root Mean Square Deviation (RMSD) 指标下要小于等于2 Å,因为小分子中的大多数原子与大分子发生相互作用。然而,肽(一种较长的生物分子)通常会结合在蛋白质表面的浅凹槽中,并且常常有许多原子并不直接与大分子发生作用。这可能导致肽的结合姿势的RMSD值较高,尽管实际上它们准确地捕捉了肽与大分子之间的相互作用(图2)。为了评估结合成功的程度,作者使用“原生接触的比例”(Fnat)来评估,这是与其他肽结合研究类似的方法。原生接触是指在晶体学复合物中,蛋白质和肽之间的重原子对,它们之间的距离在5 Å以内。通过对比结合位点和晶体学复合物中的原生接触,我们可以计算出“原生接触的比例”。对于蛋白质-蛋白质相互作用,Fnat值大于或等于0.5的复合物被认为是高质量的结合解。尽管作者报告Fnat值从0.0到1.0的成功率,但在比较各种结合方法的性能时,采用了一个更为严格的标准,即Fnat值大于或等于0.8,用于定义高质量的结合解。
实验部分
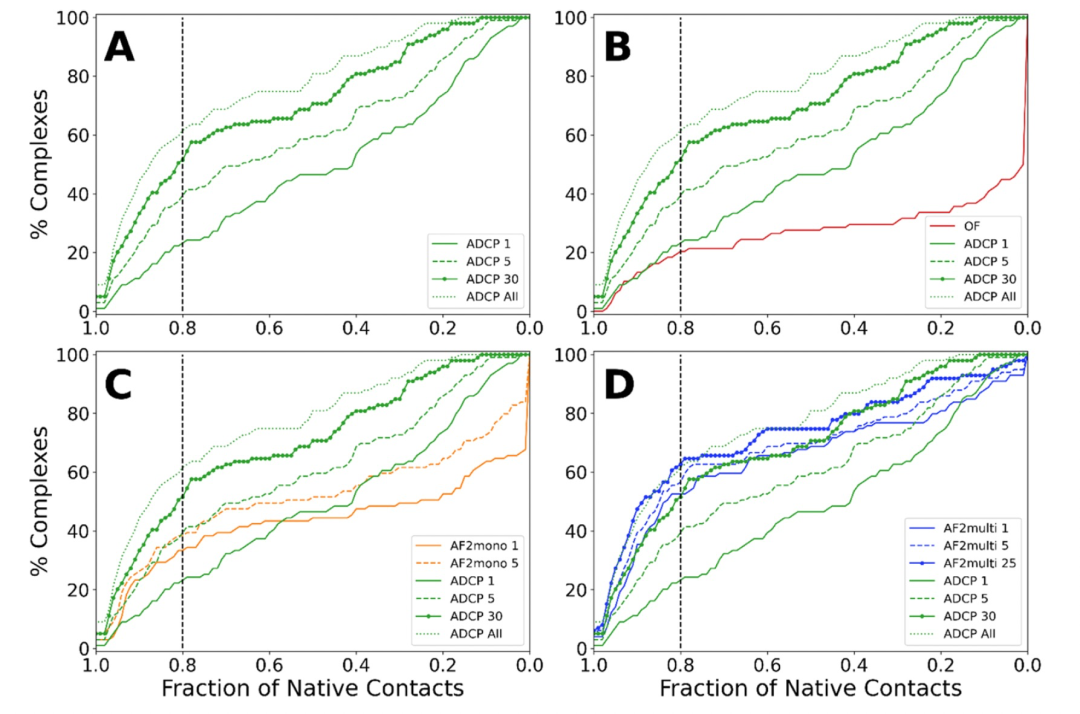
图 3
图3展示了复合物成功率的百分比,作为所有结合方法在考虑不同数量的排名靠前解时所实现的原生接触比例的函数。线的颜色表示不同的结合方法:红色代表OF,绿色代表ADCP,橙色代表AF2mono,蓝色代表AF2multi。线的样式表示在计算百分比时考虑的解的数量。实线表示仅考虑排名最高解时(所有方法都包括在内)所实现的给定Fnat的复合物的百分比。虚线表示考虑排名前5的解时获得的百分比(除了OF方法)。带有珠子的实线分别表示AF2multi和ADCP的前25或30个解。最后,绿色虚线表示ADCP报告的所有解。
ADCP取得了排名靠前解的成功率为23%(图3A)。当考虑排名前5和前30个解时,这一数字显著增加至39%和52%。当考虑所有报告的解时,成功率达到了62%,表明ADCP具有出色的采样能力,但也突显了其用于构象排名的评分函数的不足之处。与ADCP相比,OF的性能不如(图3B),其结合成功率为20%。由于OF每个复合物仅提供一个解,因此无法分析更多解对其性能的影响。虽然AF2mono在排名最高解方面表现比ADCP好(33%),但对于前5个解的性能与ADCP相似(图3C)。最后,AF2multi在排名最高解方面表现出色,其结合成功率为53%(图3D)。AF2mono和AF2multi的曲线紧凑,表明这些方法倾向于识别和报告相对较少的解,当解正确时,这可能是一个优势,否则可能是一个劣势。此外,在寻找备选的结合模式时,这也可能是一个不足之处。需要注意的是,当考虑30个或更多排名靠前解时,ADCP与AF2multi具有竞争力(图3D)。用于此基准测试的数据集专门为深度学习方法而编制。因此,它仅限于由标准氨基酸组成的线性肽。类似于环化或氨基酸修饰等改姓肽在治疗性肽中经常遇到,但目前无法通过深度学习方法处理。虽然AF2multi成为处理线性未改性肽的结合方法首选,但ADCP对于这些改性以及其识别备选结合模式的支持,使这两种方法成为互补的方法,支持更广泛范围的肽和生物靶标的结合。
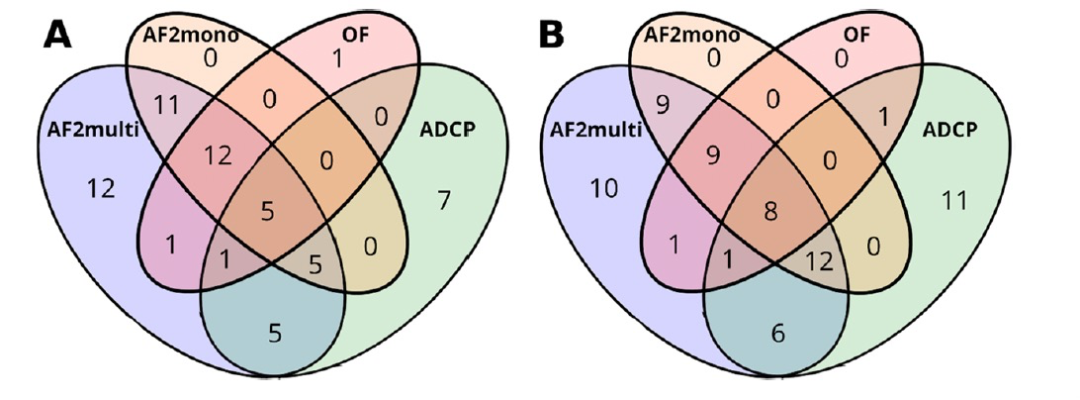
图 4
不同的方法在不同的复合物上表现出成功和失败(图4)。在考虑排名靠前、前5名、前30名和所有解时,对于61%、69%、76%和83%的复合物,至少有一种结合方法产生了高质量解。从AF2multi中获得高质量预测的复合物集合大于AF2mono,并包含后者。因此,组合这两种方法没有优势。同样,将OF与AF2multi组合在一起没有明显的优势,因为OF只在已由AF2multi正确预测的解集中添加了一个复合物。将ADCP与AF2multi组合在一起是有利的,因为ADCP在结合肽方面表现成功,而AF2multi则未能结合。当考虑选择这些解之间的合适方法时,具有正确解的复合物数量可能潜在地增加到59、68、75或82,分别对应于排名靠前、前5名、前30名或所有解。当将所有四种方法组合用于排名靠前解时(图4A),所有四种方法的组合显示出可能的61%一致性结合成功率,仅略优于仅组合AF2multi和ADCP(60%)。当使用每个复合物的多个解时,ADCP比其他方法更快地占据解空间(图4A、B),表明AF2multi和ADCP的组合最有益。此外,将排名前5的AF2multi解与排名前30的ADCP解组合在一起,理论上可以达到76%的结合成功率,前提是能够在这些解之间选择接近原生构象的可靠方法。这表明深度学习与基于物理方法的组合具有潜力,可以实现与小分子结合方法一样的结合成功率,并因此对于治疗性肽的基于结构的合理药物设计产生重大影响,就像小分子结合方法一样。
参考资料
Shanker S, Sanner MF. Predicting Protein-Peptide Interactions: Benchmarking Deep Learning Techniques and a Comparison with Focused Docking. J Chem Inf Model. 2023 May 22;63(10):3158-3170. doi: 10.1021/acs.jcim.3c00602. Epub 2023 May 11. PMID: 37167566.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


