编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Sagar D. Khare团队的一篇论文。酶具有精确和选择性地读取、编写和编辑DNA的能力,已经彻底改变了生物化学科学和技术。如果能够获得类似的酶,用于有选择性地“编辑”蛋白质,将产生广泛的影响。

蛋白酶的多特异性通过对多个底物的特异性识别和非识别而实现的,对于许多生物过程和疾病至关重要。例如,病毒和宿主蛋白酶通过对宿主和病毒靶位点的选择性识别和切割对许多RNA病毒的生命周期至关重要,包括严重急性呼吸综合症冠状病毒2(SARS-CoV-2)。因此,识别蛋白酶的蛋白质切割靶标将更深入地揭示蛋白酶的机制和生物功能。由于蛋白酶抑制剂通常被设计成模仿底物,因此预测底物的能力也可能有助于设计针对新型病毒的抑制剂。此外,能够推断蛋白酶特异性的全局景观,即由给定酶识别(和不识别)的所有底物序列基序组成的集合,还可以实现选择或设计定制蛋白酶,以降解选择的生物技术相关或与疾病相关的靶标。
当前的蛋白酶底物切割位点识别的实验方法涉及对潜在底物库进行切割的实验,一次处理一个蛋白酶变体。除了工作量大和耗时外,还只能对蛋白酶-底物序列多样性进行有限的抽样。因此,开发快速、具有成本效益且可推广的计算方法以精确预测特异性是有价值的。作者认为,一个更具语义丰富的特异性模型将包括底物序列和蛋白酶-底物复合物能量学的两个方面。特异性依赖于酶和底物之间的残基级别相互作用,因此假设高分辨率的蛋白酶-底物复合物能量学表示将具有很高的预测价值。由于蛋白质的能量是序列的结果,作者预计足够粒度和准确的能量学表示可能会消除对序列特征的需求。使用能量而不是基于序列的模型来预测蛋白酶特异性,自然地可以通过针对旨在改变蛋白酶特异性的定向进化轨迹进行训练来设计蛋白酶。为了建模特异性景观编码蛋白酶-底物复合物的拓扑和能量特征,作者开发了蛋白质图卷积网络(PGCN)。PGCN使用实验获得的数据和一个物理直观的基于结构的分子相互作用能量图,将特异性预测作为分类问题。
模型概要
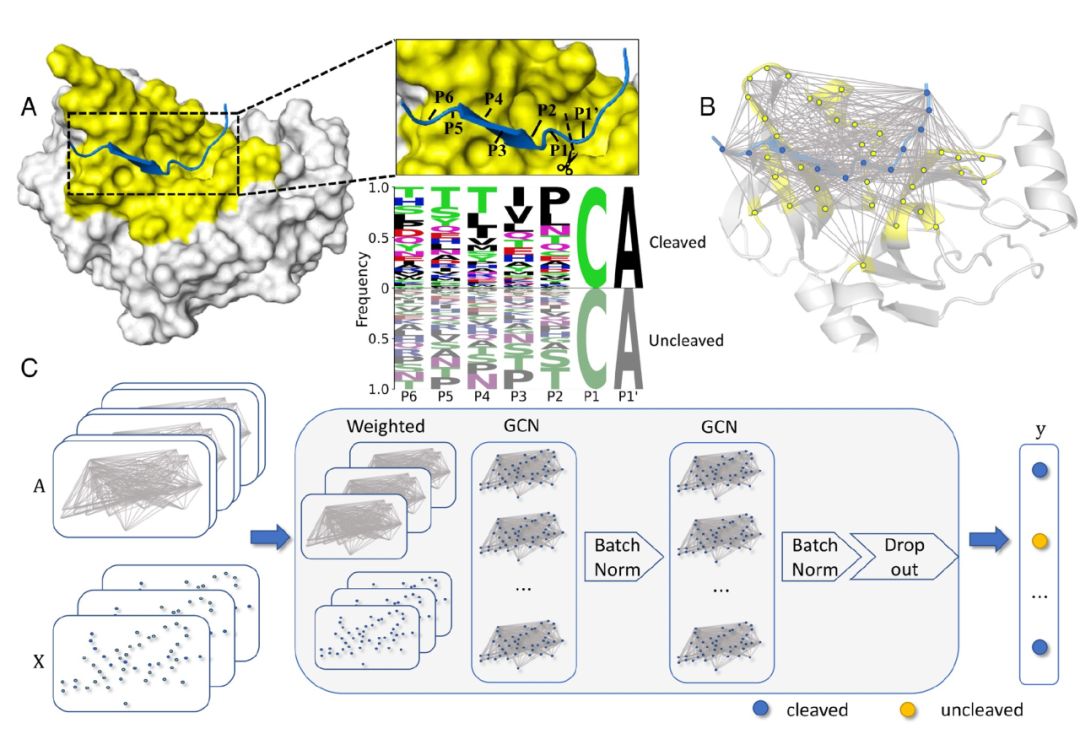
图 1
作者提出了一种名为PGCN的方法,它将蛋白质结构及其复合物建模为全连接的图,用于编码蛋白质序列、单个氨基酸相互作用能量以及成对相互作用能量,这些能量是使用Rosetta软件生成的。对于蛋白酶-底物复合物,底物肽被蛋白酶识别,然后在活性位点进行裂解或拒绝(如图1A所示)。酶-底物图(如图1B所示)被输入到图卷积神经网络中,该网络输出了给定复合物的裂解概率(如图1C所示)。作者的蛋白酶特异性数据集包括实验确定的裂解信息,即两种病毒蛋白酶NS3/4蛋白酶(以下简称为HCV蛋白酶)和TEV蛋白酶的野生型和变体的裂解和未裂解肽段列表。已确认的裂解和未裂解底物的样本集被随机分成了80%的训练集、10%的验证集和10%的测试集。
PGCN在使用不同特征编码进行底物特异性预测时表现优于基线机器学习模型
为了评估PGCN在预测底物特异性方面的性能,作者首先训练并测试了WT(野生型)和三种HCV蛋白酶变种(A171T、D183A和R170K/A171T/D183A)的特异性景观模型(表格1)。作者进一步合并了所有HCV蛋白酶变种的数据,并在这个组合数据集上训练和测试了一个单一的PGCN模型,以探索PGCN在识别蛋白酶中小结构变化引起的特异性变化方面的敏感性。
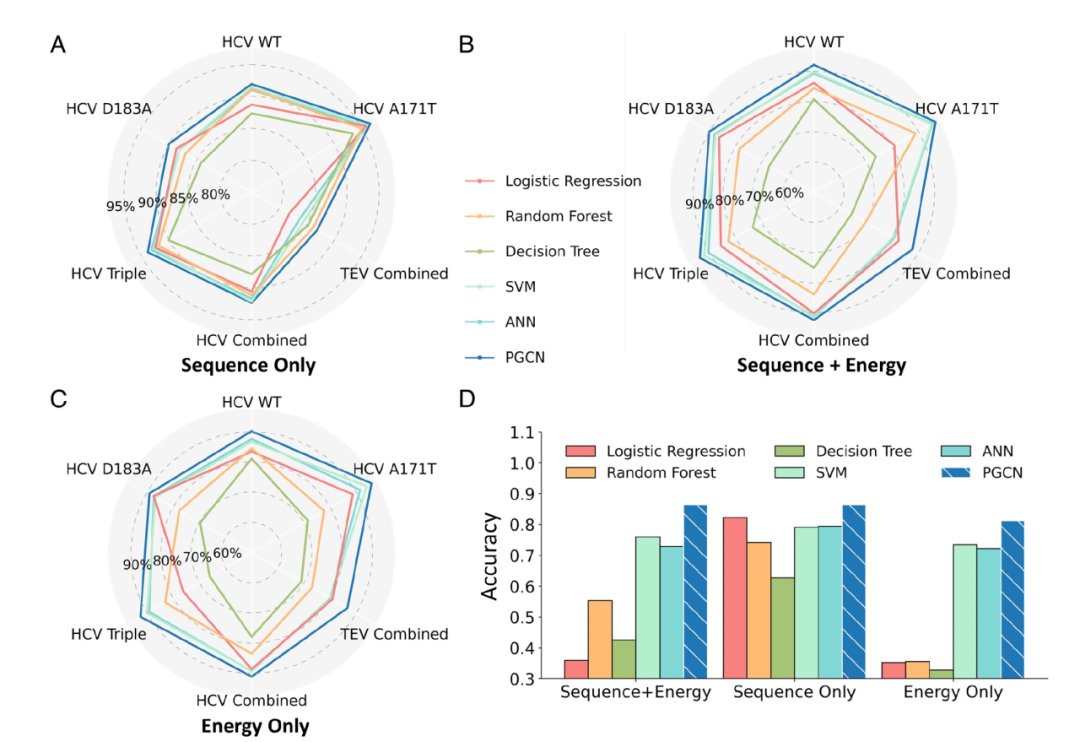
图 2
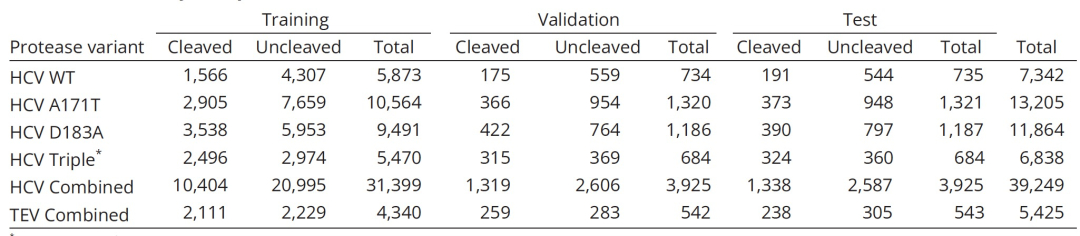
表 1
在基准测试中,PGCN在仅使用序列特征的情况下(图2A)表现出色,胜过了其他ML模型,对所有数据集,包括组合数据集,都达到了90%以上的测试准确度。除了准确度之外,作者还使用了其他不同的指标来评估PGCN的性能,包括F1分数、精确度、召回率、曲线下面积(AUC)和平均精度(AP),这些都是处理不平衡数据的ML任务的标准评估指标。PGCN在这些基准方法中具有最高的F1、召回率和AP分数。
然后,作者评估了PGCN在使用能量特征时的性能。在这些测试中仅使用Rosetta能量信息,或者将序列和Rosetta能量信息一起作为PGCN中使用的特征。如图2 B和C所示,PGCN始终在仅使用能量特征或完整序列和能量特征时表现最佳。这个结果非常显著,因为以前基于能量的蛋白酶-肽相互作用评分方法,涉及到不同能量项的加权和,性能不如基于序列的学习方法。其他基于能量的模型和PGCN之间的一个关键区别是,除了PGCN之外的所有模型在学习中使用的能量都是简单线性组合,而PGCN利用图表示法来以隐式非线性关系编码分子间的能量。因此,作者的结果表明,基于图的各个能量项的卷积是一种有前途的方法,可以结合生物物理分析和数据驱动建模,以一种方式来解决每种方法的局限性。
所有的机器学习模型在仅考虑序列特征时都能够学习一些TEV数据的模式,但在考虑能量特征时,基于树的方法、支持向量机(SVM)和人工神经网络(ANN)在准确度上表现较低(图2 B和C)。PGCN在不同特征编码之间的性能稳定,使用仅序列信息(图2A)、仅能量信息(图2C)和序列+能量特征(图2B)时的准确度分别为86.86%、86.62%和87.72%。PGCN利用了将残基级别的成对能量编码到图的边中,使得PGCN能够在每个GCN层中学习残基的局部环境。此外,使用完整的序列和能量特征的PGCN优于使用减少特征的模型,这支持了我们的假设,即蛋白酶特异性的预测受益于底物序列和酶与底物之间的物理相互作用能量。
为了确保PGCN的性能,特别是在使用序列特征时,不会在训练过程中受到底物序列模式的记忆的影响,作者还使用了基于K均值聚类的底物序列的训练、验证和测试集划分策略,使切割和未切割底物池中的底物序列在每个集合中与其他两个集合的序列远离。作者发现,无论考虑哪种特征编码,PGCN在TEV组合数据集中仍然具有最高的性能,它在使用序列+能量特征时以86.41%的准确度主导了预测,相对于其他ML模型(最佳准确度为SVM的75.96%)(图2D)。HCV蛋白酶也获得了类似的结果。因此,作者得出结论, PGCN不是基于记忆或学习(最近邻)底物序列模式,因此采用了节点-边消融测试来进一步研究PGCN性能的来源。
节点-边重要性分析以从PGCN中获得物理洞察力
PGCN的一个优点是,节点和边直接对应于物理氨基酸残基及其相互关系。因此,作者认为通过识别对PGCN性能至关重要的节点和边,就可以确定重要的残基和相互作用。为了通过PGCN确定每个图组件的预测强度,作者在所有样本图中扰动了每个节点(或边)的特征值,然后重新计算准确性。扰动后的准确性降低用于衡量PGCN图中节点i(或边j)的(相对)重要性。
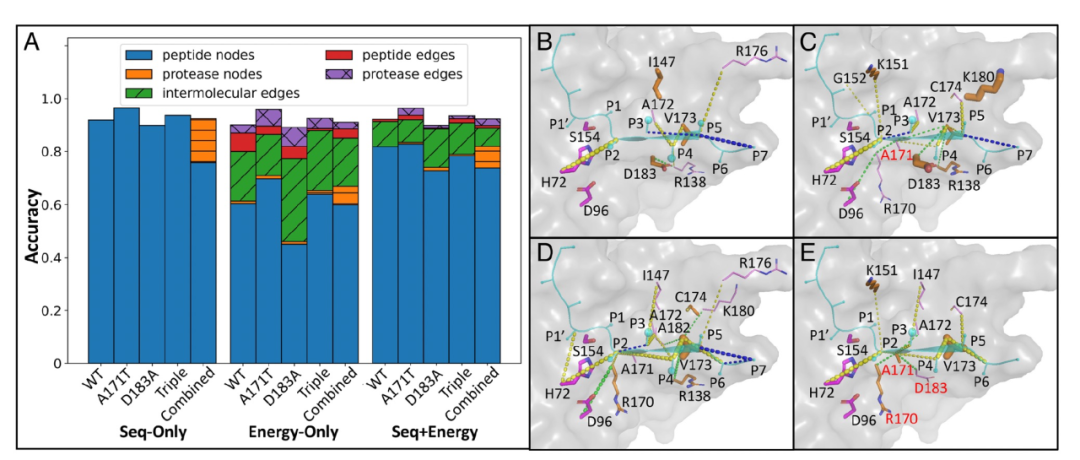
图 3
作者通过预测的整体准确性对节点/边的计算重要性进行了归一化,并按特征类型(节点或边)汇总了归一化的重要性,以查看PGCN用于训练的特征如何影响分类。根据连接给定边的节点类型,有两种类型的节点(蛋白酶、底物)和三种类型的边(蛋白酶-蛋白酶、底物-底物和分子间),当仅考虑序列特征时(边上没有任何特征),如预期的,只有肽节点对单变体集的准确性有贡献(图3A)。然而,对于也采样了蛋白酶多样性的数据集(图3A中的“组合”数据集),通常发生替代的蛋白酶节点也被检测为准确性的贡献者。当考虑能量特征时,无论是仅有能量特征还是与序列一起考虑,蛋白酶节点都作出了显著更大的贡献(图3A),这表明蛋白酶残基能量对其环境的变化非常敏感。同样,当排除序列信息时,对边特征的依赖性增加,而预测的整体准确性没有受到显著影响。利用能量信息允许更广泛地关注残基-残基相互作用,因为更多的边被认为是显著重要的。这些观察结果表明,序列是PGCN在可用时使用的一种抽象表示,但是相同的信息也可以从能量中学习。
接下来,作者可视化了HCV蛋白酶结构中重要节点和边的位置。对于WT和每个变体蛋白酶,一个关键的边位于底物的P2残基和催化碱基H72之间,这可能反映了底物在活性位点中的适当定位。作者还观察到一些重要的节点/边在WT和变体蛋白酶之间是不同的。例如,蛋白酶边R138-D183在野生型中非常显著(图3B),但在A171T(图3C)或D183A(图3D)或Triple变体A171T、D183A和R170K(图3E)中并不是一个重要的相互作用。当引入D183A突变时(图3D和E),一些分子间边(例如P6-R138)的侧链取向不同,尽管蛋白酶节点D183A本身并没有显著影响底物特异性的分类。相反,作者发现一些其他分子间边,例如P3-I147、P2-A171(T)、P4-A171(T)、P4-V173和P6-V173,对于在D183A和Triple变体上训练的模型来说,至少重要性是野生型和A171T数据上训练的模型的两倍。此外,只有当D183A发生突变时,蛋白酶节点R170(K)才显示出其重要性(图3D和E)。然而,重要的边和节点列表并不是可加的:例如,对于Triple变体来说,边138到173和96到170都不重要(图3E),而它们是在D183A上训练的模型中最重要的两个蛋白酶边(图3D)。考虑到在HCV组合集上训练的模型,尽管对于单一变体集的重要节点和边对于组合集同样重要(例如节点V173、边P2-H72、P4-V173等),但也有一些只对个体变体或野生型内的底物特异性预测有用的重要节点/边,例如P3-I147。因此,该模型能够使用有重叠但不同的节点和边特征集来实现近似相同水平的准确性分类。
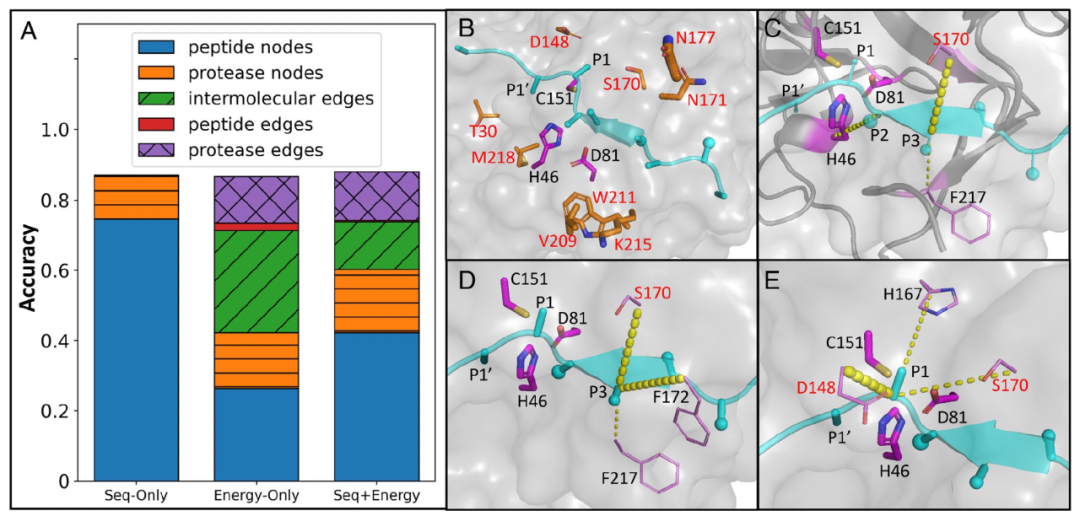
图 4
在TEV蛋白酶预测的节点/边敏感性分析中,出现了类似的趋势(图4A)。一些氨基酸替代位点在TEV变体中具有很高的重要性,例如D148、S170和N177(图4B)。还确定了一些重要相互作用的强烈信号。例如,底物的P2残基与催化碱基H46之间的相互作用在所有TEV变体和野生型中始终很重要(图4C),与上述HCV预测相同。此外,还发现了一些其他边,例如P3-S170(图4C),S3口袋周围的蛋白酶内部边(图4D)和S1口袋(图4E)也具有很高的重要性。
参考资料
Lu, C., Lubin, J. H., Sarma, V. V., Stentz, S. Z., Wang, G., Wang, S., & Khare, S. D. (2023). Prediction and design of protease enzyme specificity using a structure-aware graph convolutional network. Proceedings of the National Academy of Sciences, 120(39), e2303590120.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


