编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Giuseppe Felice Mangiatordi团队的一篇论文。此项研究引入了一种名为”GENERA”的全新设计算法,它将自动药物类似物设计的深度学习算法”DeLA-Drug”的能力与生成具有目标定向性质分子的遗传算法相结合。GENERA被应用于血管紧张素转化酶2(ACE2)靶点,该靶点与许多病理条件(包括COVID-19)有关。通过两个分子对接程序,PLANTS和GLIDE,评估了GENERA在为特定靶点全新设计有前景的候选分子的能力。

药物发现(Drug Discovery,DD)成本高昂且耗时。平均而言,将一种新药带到市场上需要10年时间和大约27亿美元。考虑到这些限制,现代制药行业优先使用计算工具来最小化必须经受昂贵的临床前和临床测试的候选物数量,从而节省资金并加速流程。近年来,出现了新的基于结构和配体的模型来应对这一挑战性任务。这些预测模型使得虚拟筛选(VS)策略能够从大量的预先合成和易于合成的化合物库中识别出有前景的候选物。
作者在最近的论文中提出了一种名为DeLA-Drug的新的深度学习(DL)算法,用于药物类似物的数据驱动生成。该模型使用从ChEMBL提取的100多万个化合物的SMILES字符串语法进行训练,使用一种名为“采样与替代”的新方法从单个查询中生成药物类似物分子。与在全新药物设计中采用的其他方法不同,该算法不涉及精细调整步骤来引导生成阶段,使其(i)适用于数据较少的情况,在该情况下,没有针对所关心的目标获得已知实验数据的大量化合物数据集,以及(ii)可以在易于使用的网络平台上实施,只需要一个种子(起始)化合物的2D结构或SMILES字符串。然而,DeLA-Drug从整个ChEMBL中学到的结构模式是通用的,使得该工具能够为初步筛选生成广泛多样的库,而不是特定的目标定向库。为了克服这一限制,作者将DeLA-Drug与遗传算法(GA)相结合,以引导生成(预测的)面向目标的化合物,其中化合物与目标结合的倾向性通过对接分数进行估计。由此产生的计算工作流程称为GENERA,被应用于全新抑制剂设计,目标是ACE2酶,在各种病理条件中起关键作用,包括COVID-19。通过从有限的一组化合物开始(例如,一组已被证明对所关心的目标具有活性的分子),GENERA能够迅速生成新的候选物,这些候选物(i)在化学上是有效的,(ii)探索了新的化学空间,并且(iii)在进一步的体外研究中非常有前景,因为它们的对接分数甚至比已确认的活性物质还要好。
方法部分
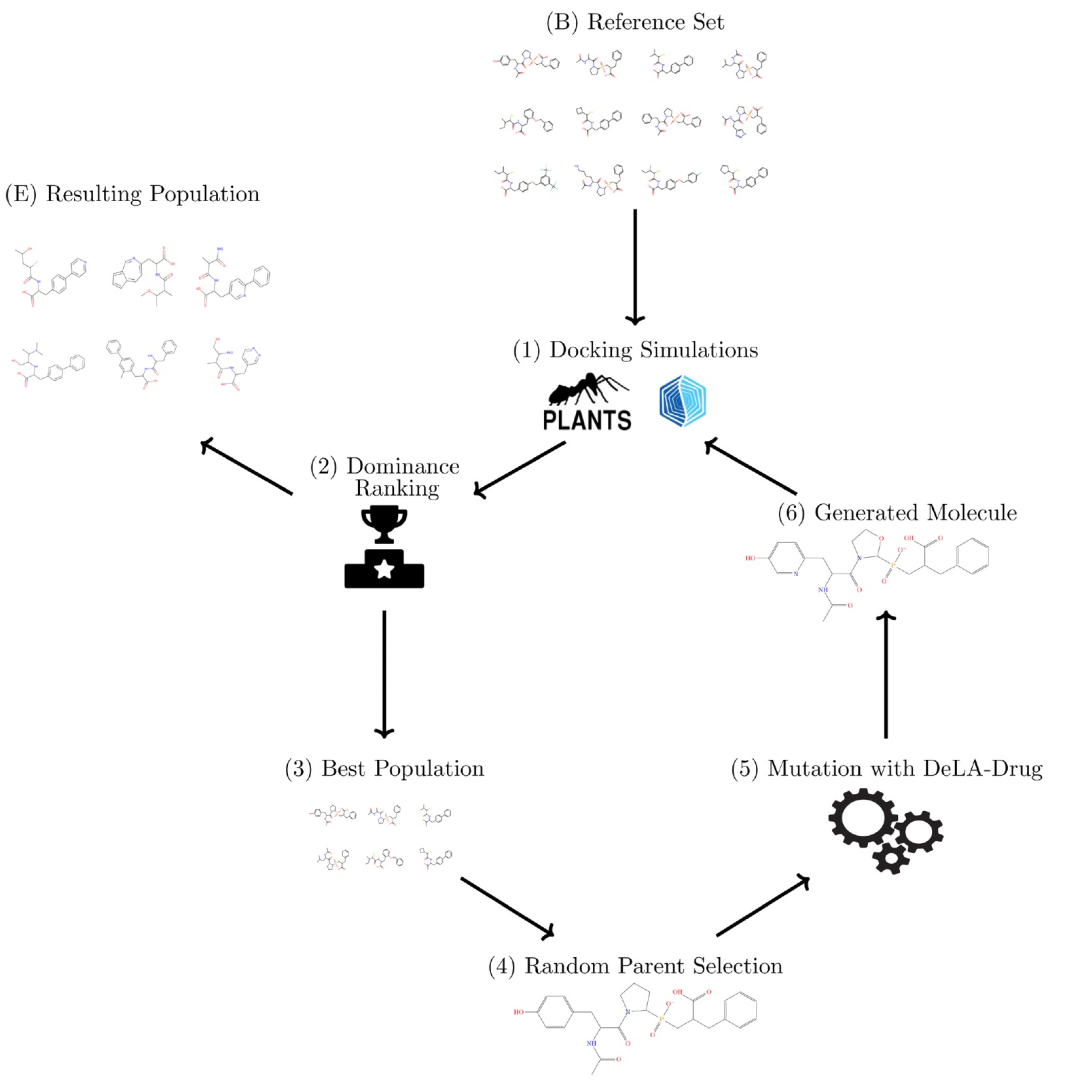
图 1
DeLA-Drug是一个深度生成模型,它学习了来自ChEMBL的1,092,285个化合物的SMILES字符串的语法,并从给定的查询分子开始生成药物类似物。该模型基于一个由两个LSTM层组成的RNN。
将DeLA-Drug与GA相结合产生了一种新的体系结构,其主要步骤如图1所示,以下进行详述:在1到5之间随机选择特定数量的替代物,并将其应用于可用的SMILES之一的随机位置。对于每个随机选择的父化合物,都会生成一个子化合物。请注意,DeLA-Drug会对生成的结构进行验证。如果在1000次尝试后没有生成有效的结构,则算法将继续选择新的替代位置组合。首先,将子SMILES标准化为其规范形式,然后在已处理项的存储库中检查其唯一性,该存储库由GENERA脚本保持更新。如果当前的SMILES已经被访问过,则放弃当前尝试。否则,使用RDKit作为工具,通过一系列快速的亚结构搜索来识别反应性或化学不稳定的基团。如果找到了这些基团,就会丢弃候选化合物,并将其多个适应度分数设置为非常低的水平。否则,算法会继续进行配体准备并在感兴趣的结合位点上进行对接,同时使用PLANTS和GLIDE检索相应的适应度分数。
作者使用GENERA进行了两轮多目标优化:(i)仅基于PLANTS,以及(ii)基于PLANTS和GLIDE的“共识”对接。这些优化在一个多节点、多核的Linux集群上异步运行。这些运行在执行时会获取包含当前“最佳”可生成后代的SMILES字符串的文件的当前状态,然后随机选择一个分子,利用DeLA-Drug生成“突变体”后代,接着依次运行PLANTS和GLIDE,计算上述提到的各项指标。接着,设计者脚本将一个包含后代SMILES及其目标得分值的一行文本写入GENERA工作目录。主GENERA脚本定期检查这些新的条目。一旦发现,这些新条目会被添加到一个包含所有迄今为止生成的SMILES及其适应性分数的存储库中。然后,当前“最佳”SMILES文件和新条目会合并,并提交给帕累托前沿工具。这个工具会检测并删除任何在输入中被其他项目“支配”的项目。在技术上,如果存在至少一个其他项目在所有目标上都更好(严格>),那么该项目就会被认为是“支配”的。帕累托前沿,也就是在所有目标上都没有被“支配”的项目的子集,将会由帕累托前沿工具输出,然后被重命名为新的“最佳”项目池。
实验部分
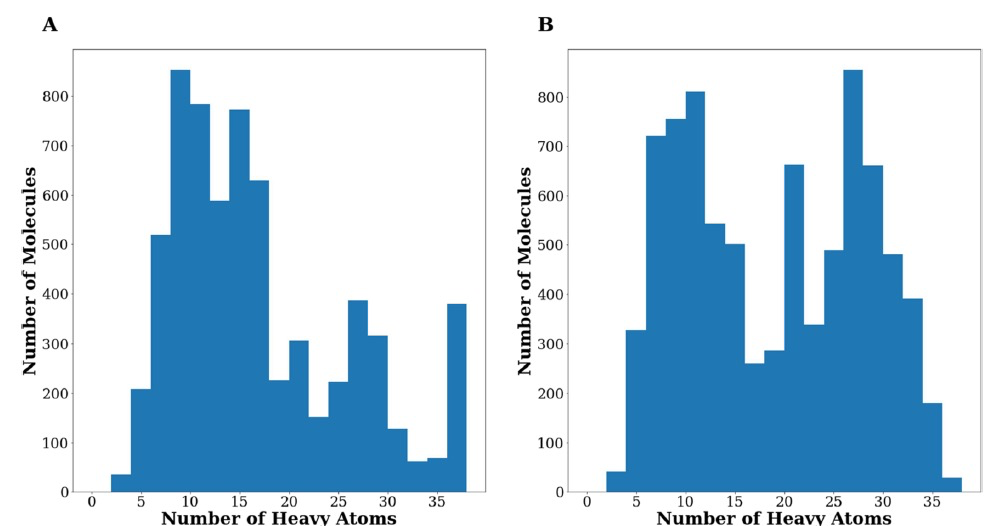
图 2
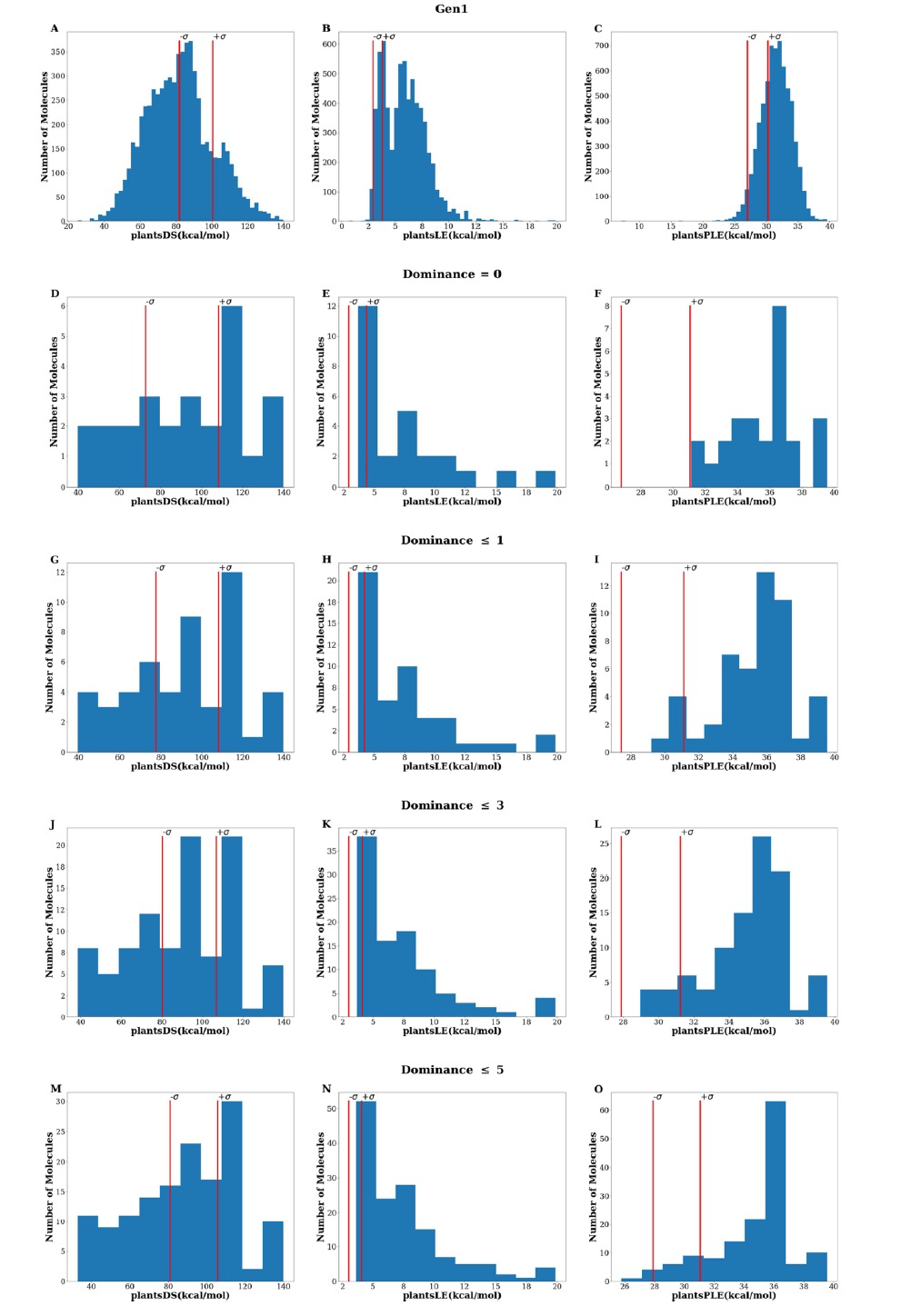
图 3
GENERA从针对ACE-2的初始活性分子开始,设计了6648个独特且化学上有效的分子(以下称为Gen1集)。帕累托前沿的部分目标取决于分子的大小。具体来说,对接得分偏好较大的分子,而配体效能偏好较小的分子。图2A显示了Gen1集中重原子的分布情况。从图中可以看出,GENERA可以设计不同大小的分子,范围从具有少于5个重原子的片段到具有超过35个重原子的化合物。图3显示了生成化合物返回的PLANTS得分分布。对整个Gen1集的初步检查证明了GENERA生成对接化合物的能力。尤其是当观察图3C时,这种能力特别明显,显示出大多数生成的化合物的plantsPLE值优于初始参考集。
仅基于分子对接模拟选择候选物存在许多限制,因为评分函数的准确性强烈依赖于所研究的特定靶点。这些弱点可以通过结合多种软件工具来进行至少部分缓解。实际上,这种方法可以提高虚拟筛选活动的命中率,正如先前的研究所报道的。基于这些证据,作者挑战使用GENERA将PLANTS和GLIDE进行组合,两者均作为帕累托前沿的输入。在生成过程中使用了EVS、plantsDS、plantsLE、glideDS和glideLE作为目标。作者使用了与之前相同的活性化合物参考集,并生成了一个新的8336个独特且化学上有效的分子集(从现在起称为Gen2集)。值得注意的是,Gen2集平均来说由较重的化合物组成。实际上,属于Gen2的化合物中有56%(与Gen1中的43%相比)的化合物具有超过15个重原子。通过比较Gen1和Gen2集合返回的重原子分布(图2A vs 2B),这种差异也是明显的。值得注意的是,GENERA能够设计出在PLANTS和GLIDE都预测要比起始活性分子更好的化合物。当根据计算出的支配读(Dom)选择子集时,这一点尤为明显(图4)。
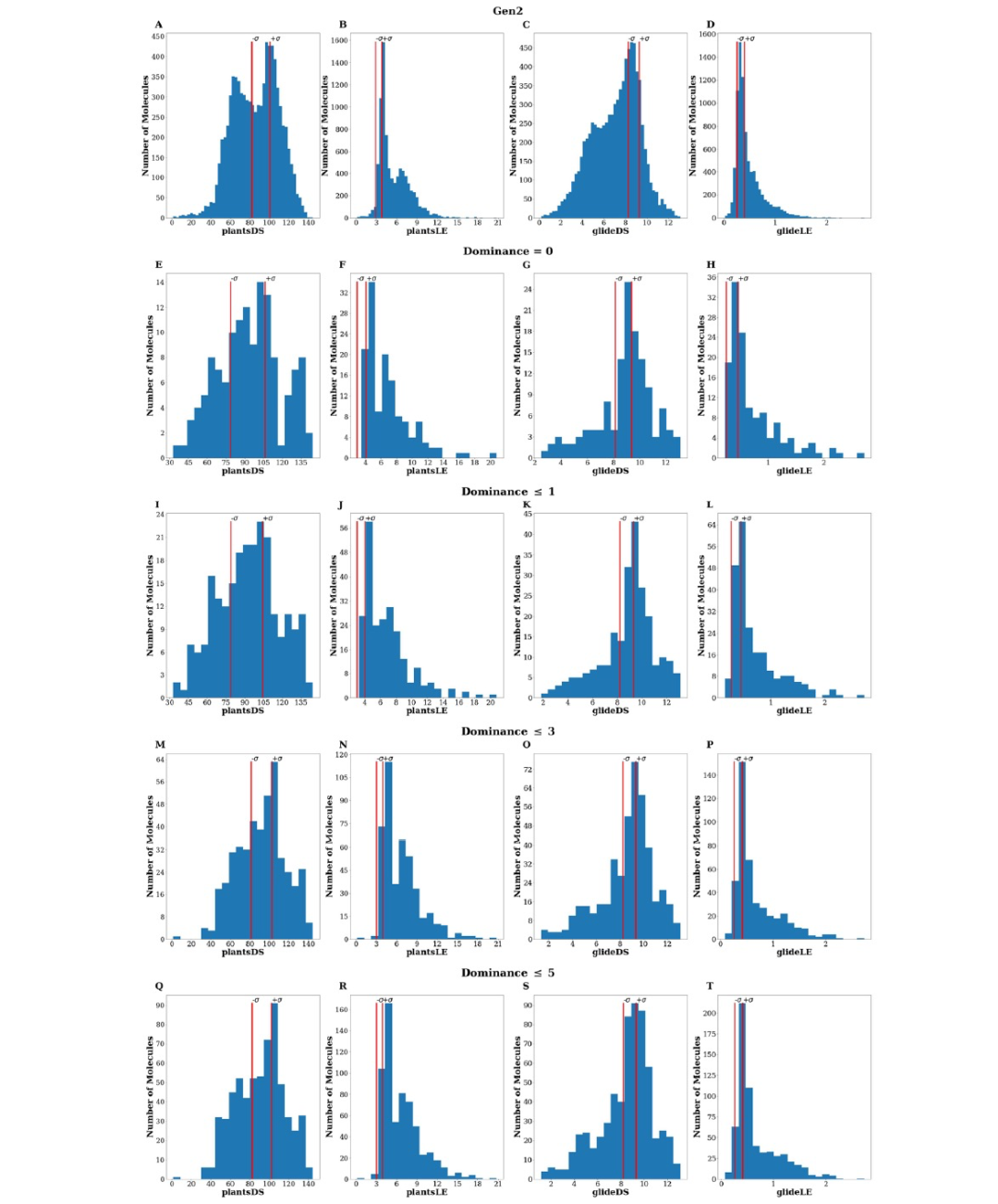
图 4
结论
作者的研究引入了GENERA,这是一种新颖的算法,将深度学习生成模型DeLA-Drug与遗传算法框架相结合。作者对GENERA生成的专有库的分析揭示了其迅速优化用户定义属性的能力。与旨在“易于”修改的定制分子表示相比,使用DeLA-Drug具有明显的优势。首先,它可以“即插即用”地嵌入到任何进化算法模板中,而无需设计任何特定于数据结构的突变过程。最重要的是,使用DeLA-Drug可以让用户从其训练过程中学习到的“化学知识”中受益。将深度学习中学习的“化学知识”用于强化化学空间采样的方法具有重要的灵活性,因为它在使用方面没有任何先决条件。因为目标函数可以无缝地与该工具相结合,任何接受SMILES作为输入并返回良好度量分数作为输出的可执行文件或脚本都可以,该方法支持对任何性质的终点进行(多目标)优化。
参考资料
Lamanna, G., Delre, P., Marcou, G., Saviano, M., Varnek, A., Horvath, D., & Mangiatordi, G. F. (2023). GENERA: A Combined Genetic/Deep-Learning Algorithm for Multiobjective Target-Oriented De Novo Design. Journal of Chemical Information and Modeling.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


