编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自 Guo-Wei Wei团队的一篇论文。

阿片类物质使用障碍(OUD)已成为一个重要的全球公共卫生问题。针对OUD的主要治疗方法通常包括药物和行为干预的综合应用,旨在解决成瘾的生理和心理方面,促进康复,并防止复发。美国食品药品监督管理局(FDA)已批准了三种药物,包括美沙酮、布比洛芬和纳曲酮,用于OUD的治疗。这些药物通过与大脑中的阿片受体结合发挥作用,即μ阿片受体(MOR)、kappa阿片受体(KOR)和delta阿片受体(DOR)。美沙酮是一种长效阿片受体激动剂,主要作用于MOR。它有助于减轻戒断症状和渴望。布比洛芬则作为部分性阿片受体激动剂,主要针对MOR。它缓解戒断症状和渴望,同时产生较少的欣快感,并且与美沙酮相比,呼吸抑制的风险较低。纳曲酮被归类为阿片受体拮抗剂,它阻止了阿片类药物的作用,并减少了愉悦效应。它的作用机制主要涉及MOR,但它也对KOR具有一定的亲和力。在这项研究中,作者提出了一个深度生成模型,将基于随机微分方程(SDE)的扩散模型与预训练的自编码器相结合。分子生成器能够高效地生成针对多种阿片受体的分子。
多目标随机生成网络复合体(MTSGNC)的结构
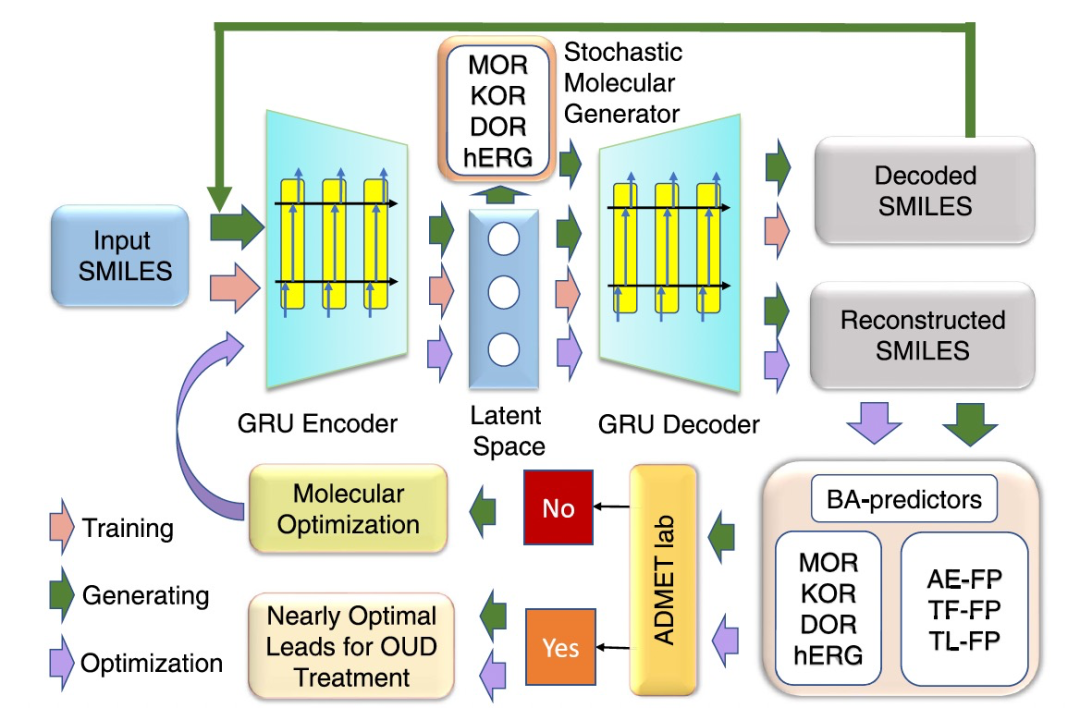
图 1
在分子科学中,自编码器可用于分子表示学习,其过程涉及将分子编码为低维空间,同时保留其结构和功能特性。SMILES是常用的分子表示之一。作者训练了seq2seq自编码器,将输入和输出的SMILES之间进行转换,从而使潜在向量携带了准确的分子信息。因此,潜在空间可以用于表示化学空间,并用作机器学习建模的分子指纹。自编码器还可用于生成建模,在这种情况下,解码器可以通过从潜在空间中进行采样来生成新的数据点。自编码器(AE)的结构如图1所示(左上方)。研究中,作者使用预训练的seq2seq自编码器来设计用于OUD治疗的新型分子。
MOR、KOR 和 DOR 是治疗 OUD 的三个关键药物靶点。此外,hERG 是在药物发现中需要避免的一个关键靶点。作者从 ChEMBL 数据库中收集了这四个靶点的抑制剂数据集。这些数据点包括分子化合物的 SMILES 字符串,以及以 IC50 或 Ki 形式的相应结合标签。这些数据集在两个主要方面得到了利用。首先,分子化合物,特别是那些在 MOR、KOR 和 DOR 上表现出强效的化合物,被用作生成新化合物的参考和种子。其次,这四个数据集被用作训练集,构建用于预测结合亲和力的机器学习模型。
生成模型是生成潜在新药分子的强大工具。在作者之前的工作中,作者引入了用于生成新型药物样分子的GNC模型。作者考虑了三种潜在空间扰动模型,包括(1)称为“随机输出”的随机噪声扩散模型,(2)称为“受控输出”的梯度下降模型,以及(3)称为“优化输出”的多目标优化模型。第一个模型通过高斯白噪声扰动潜在空间分子向量,然后类似于蒙特卡洛方法,选择具有改进性能的分子。这个模型确保了生成分子的新颖性,但可能无法在达到理想的药物样性质方面有效。第二个模型通过一个受力驱动项改进生成分子的特定分子性质。然而,产生的新分子可能不会保留其他重要性质,缺乏新颖性。第三种方法旨在通过多目标损失函数同时优化生成分子的多个分子性质。然而,生成分子的新颖性可能无法得到保证。为了提高GNC模型的性能,作者提出将随机噪声扩散模型与多目标优化模型结合起来。
图1展示了GNC中的化合物生成过程。作者的目标是设计对MOR、KOR和DOR具有有效性的新型药物样化合物,同时不具有hERG副作用。分子生成过程包括四个关键步骤。1.从收集的数据集中分别选择在MOR、KOR和DOR上有效的三种化合物作为参考化合物。还从这些数据集中选择在三种受体中多种化合物上有效的种子化合物。然后,通过预训练的编码器将参考和种子化合物的SMILES字符串编码为潜在向量。2.将参考和种子化合物的潜在向量输入到随机分子生成器中,产生大量新的潜在向量。这些新的潜在向量作为潜在分子的表示,受到了MOR、KOR和DOR的结合亲和力约束,以及hERG的结合亲和力约束。预训练的结合亲和力预测器在结合亲和力约束评估中被使用。3.具有期望结合亲和力性质的潜在向量被解码成有效的SMILES。然后,这些SMILES通过预训练的编码器和解码器再次输入,如图1所示,以识别可以成功重构的分子。那些重构的分子被认为在结构上是稳定的,并且可以被自编码器网络很好地解释。4.那些重构的分子在结合亲和力和ADMET性质方面进行重新评估,以识别药物样化合物。随后可以实施分子优化过程,设计更多具有所需可药性特征的药物候选化合物。
在GNC生成器中,作者整合了四个结合亲和力(BA)预测器,以评估生成的潜在分子在四个关键靶点上的结合亲和力。为构建这些预测器,作者使用从自编码器网络的潜在空间中得出的分子指纹。这些指纹称为AE-FP,得到的四个BA预测器被表示为AE-BPs。AE-BPs通过将AE-FPs与深度神经网络(DNN)算法进行整合来完成。模型被用于进一步评估那些可以重构为SMILES的分子的结合亲和力,如图1右下所示。另外还使用了两种分子指纹,即Transformer指纹(TF-FP)和拓扑拉普拉斯指纹(TL-FP)。它们是通过预训练的Transformer模型和作者最近提出的拓扑拉普拉斯理论设计的。
BA预测器通过将TF-FP与深度神经网络(DNN)算法整合,以及将TL-FP与梯度提升决策树(GBDT)算法融合来构建,分别称为TF-BP和TL-BP。共识模型或预测通过对AE-BP、TF-BP和TL-BP的预测进行平均得到。这种策略可以增强机器学习预测,通常优于单独的模型。这种方法在以前的关于OUD的研究中得到了应用,涉及将DrugBank化合物进行机器学习再利用以用于OUD治疗,以及对OUD相互作用网络进行机器学习分析。模型的预测性能通过5折交叉验证进行了评估。所获得的平均皮尔逊相关系数(R)分别为MOR、KOR、DOR和hERG数据集分别为0.824、0.840、0.845和0.756。此外,相同数据集的平均均方根误差(RMSE)值分别为1.010、1.027、1.006和0.801 kcal/mol。
生成用于MOR、KOR和DOR的新型多靶点抑制剂
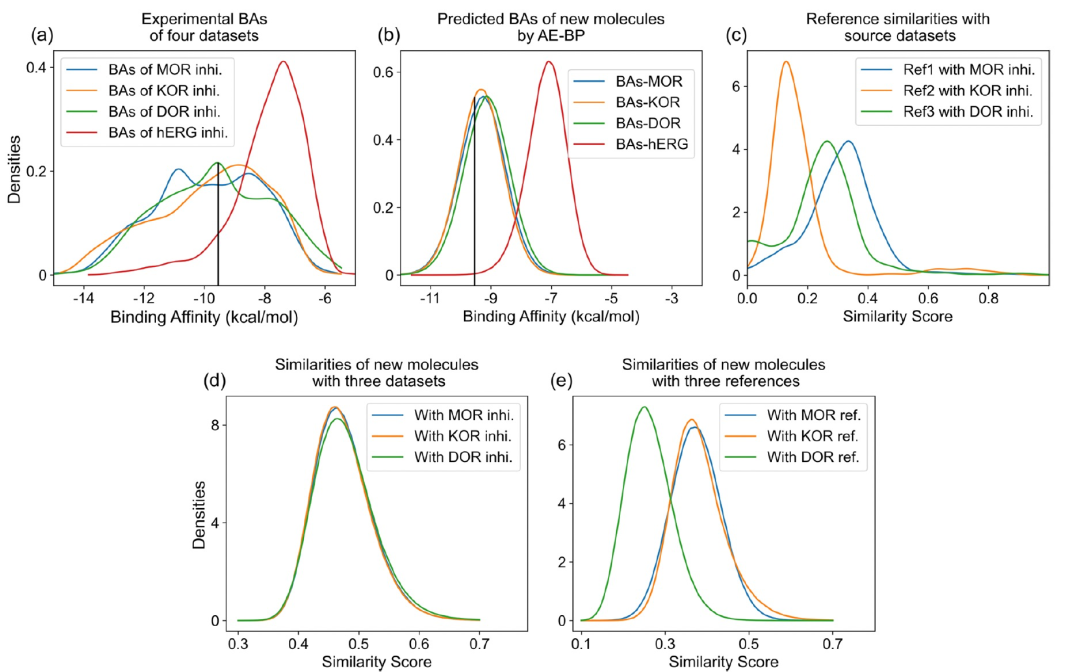
图 2
针对OUD治疗的FDA批准药物在MOR、KOR和DOR上具有高度的效力。GNC可以设计更多同时对这三种受体有效的分子。与此同时避免具有潜在hERG副作用的抑制剂。选择适当的参考和种子化合物对于生成有效的分子至关重要。作者将在收集的数据集中表现出对阿片受体有效性的化合物作为我们的参考。这是因为生成的化合物,继承这些参考的药效团,更有可能在受体上表现出效力。图2a显示了这些数据集中抑制剂的结合亲和力(BA)分布。显然,在MOR、KOR和DOR数据集中都有大量的有效分子,其结合亲和力值都在-9.54 kcal/mol以下。这个阈值被广泛接受用于鉴定活性化合物。这三个数据集共包含2152个共同的化合物。作者依靠机器学习模型来预测生成化合物的结合亲和力值。这三个数据集展示了广泛的BA分布,范围从-14到-6 kcal/mol,这表明存在高度多样的分子。此外,与-9.54 kcal/mol的BA阈值相比,BA数据呈平衡分布。这种训练数据的平衡分布有助于无偏的BA预测。
在第一个实验中,作者从抑制剂数据集中选择了三种化合物,分别是ChEMBL2048770、ChEMBL3349979和ChEMBL494462。这些化合物在MOR、KOR和DOR上都表现出有效性,其相应的结合亲和力值分别为-11.51、-11.58和-11.92 kcal/mol。因此将它们分别用作各自受体的参考化合物。种子化合物在MOR、KOR和DOR上的结合亲和力值分别为-10.44、-10.5和-8.96 kcal/mol。它在DOR上作为弱抑制剂。通过使用ChEMBL494462作为DOR的参考化合物,生成的化合物可以包含ChEMBL494462中存在的某些基团。因此,这些生成的分子可能在DOR上表现出有效性。利用参考和种子化合物,GNC在短短几个小时内利用超级计算机生成了一百多万个新的有效分子。随后,这些化合物通过编码-解码网络,保留了那些可以成功重构的分子,产生了大量的新型分子库。首先使用AE-BPs评估生成化合物的结合亲和力,鉴定出那些多靶点活性分子。图2b显示了通过AE-BPs重构的分子的结合亲和力分布,表明三个阿片受体的活性化合物数量很大。此外,只有极少数这些分子可能会引起hERG副作用。从不同角度调查分子相似性分数至关重要,因为它们与机器学习预测相关。一个重要的相似性分数是参考化合物与各自源数据集之间的相似性分数。这些参考化合物是从MOR、KOR和DOR抑制剂数据集中选择的,这些数据集被用作开发BA预测器的训练数据。生成的分子与参考化合物之间存在相似性。图2c展示了参考化合物与其源数据集之间的相似性分数分布。图2d展示了生成的分子与三个抑制剂数据集之间的相似性分数分布。每个相似性分数由生成分子的AE潜在向量与相应数据集中所有分子计算出的最高Tanimoto系数确定。大部分相似性分数都在0.4-0.6的范围内,表明生成的分子之间具有很高的新颖性。
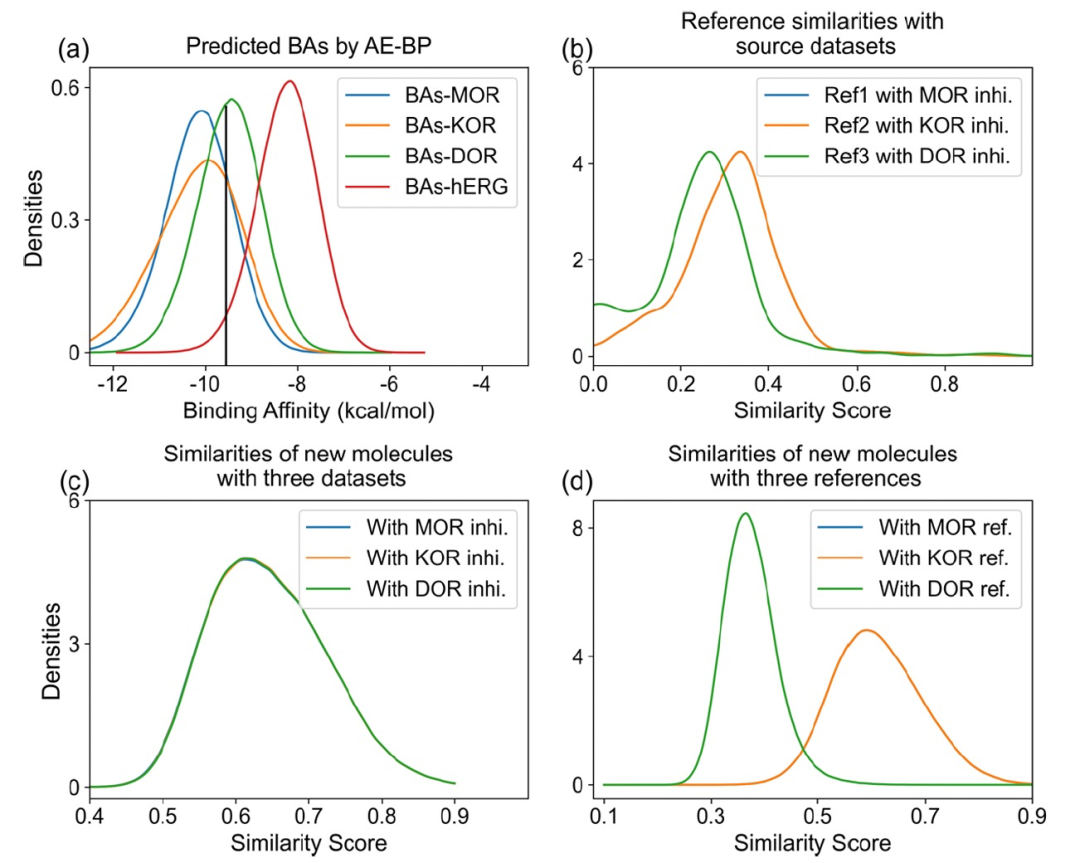
图 3
作者进行了第二个实验,使用两个参考化合物生成新分子,旨在提高与训练数据的分子相似性,并提高BA预测的准确性。在之前的实验中使用的同样两个化合物ChEMBL2048770和ChEMBL494462被利用。ChEMBL2048770作为MOR和KOR的抑制剂,具有分别为-11.51和-11.78 kcal/mol的结合亲和力。ChEMBL494462则专门与DOR结合,其BA值为-11.92 kcal/mol。在这个测试中,ChEMBL2048770被选为MOR和KOR的参考化合物,而ChEMBL494462作为DOR的参考化合物。继续使用ChEMBL243195作为种子化合物。模型生成了数百万个新的有效分子。图3展示了第二个实验中分子生成的结果。与前一个实验类似,GNC成功地为MOR、KOR和DOR目标生成了大量的活性分子,同时在hERG副作用方面表现出较弱的影响,如图3a所示。如预期的,生成的分子与训练数据之间的相似性得分得到了提高,如图3c所示。平均相似性得分约为0.6,高于在之前的实验中使用三个参考化合物获得的大约0.45的分数,如图2d所示。此外,在图3d中可以观察到MOR和KOR参考化合物的相似性得分高于DOR参考化合物,这与图3b一致。值得注意的是,在图3b和d中,MOR和KOR参考化合物的分布曲线重叠在一起,因为ChEMBL2048770被同时用作MOR和KOR目标的参考化合物。通过比较这两个实验,可以看出使用两个参考化合物可以是一种有效的方法,用来设计新颖的分子。
结论
作者开发了一种高度有效的深度生成模型,用于生成对多个靶点具有作用的新型分子,包括MOR、KOR和DOR。分子生成器是通过将基于随机微分方程(SDE)的扩散方法整合到预训练的自编码器模型的潜在空间中而设计的。通过精选适当的参考化合物并遵循一系列的新颖性标准,可以生成大量具有理想结合亲和力的新型化合物,这些化合物对MOR、KOR和DOR具有结合亲和力,同时具备其他药物样性质。
参考资料
J. Med. Chem. 2023, 66, 17, 12479–12498 Publication Date:August 25, 2023
https://doi.org/10.1021/acs.jmedchem.3c01053



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


