2023年10月5日,Nature杂志发表了一篇题为AlphaFold touted as next big thing for drug discovery — but is it的文章,评述了AlphaFold等AI工具在制药行业的价值。文章认为,AlphaFold能否彻底改变药物发现,仍需实践检验。

蛋白质结构预测
在谷歌Deepmind的AlphaFold于2020年证明它能高精度地预测蛋白质的3D形状之后,化学家们开始对利用这一开源人工智能程序更快、更便宜地发现药物的前景感到兴奋。大多数药物是通过与蛋白质上的不同位点结合发挥作用的,而AlphaFold可以预测蛋白质的结构,科学家们以前对这些蛋白质知之甚少。 AlphaFold预测蛋白质的结构
AlphaFold预测蛋白质的结构
一些企业在AlphaFold技术成就的基础上,积极探索药物发现的新方法。这其中就包括Recursion。
Recursion:在AlphaFold的基础上,连接蛋白质与化学宇宙
2023年8月8日,处于临床阶段的领先生物技术公司Recursion(NASDAQ: RXRX)宣布,该公司利用最近从Cyclica收购的MatchMaker技术,对Enamine REAL Space化学库进行筛选,计算出了360亿种潜在药用化合物与15000多种人类蛋白质的结合情况。这一成果的取得得益于几个方面,包括从AlphaFold2数据库中获得的15000多种人类蛋白质的预测结构(包含8万多个潜在的结合口袋),以及据说是世界上最大的可搜索化学库(包含约360亿个按需制造的分子)的Enamine REAL Space。
很多人都预测过分子如何与蛋白质结合,但这么多的预测是史无前例的。
—Recursion联合创始人兼首席执行官Chris Gibson
Recursion联合创始人兼首席执行官Chris Gibson博士说:“这一成就标志着我们向实现解码生物学和化学的使命迈出了令人振奋的重要一步。在此之前,生物学和化学领域取得的突破性进展(即AlphaFold、Enamine虚拟化学库以及大规模计算和新机器学习方法的快速发展)在很大程度上是相互孤立的,或者是在相对较小的范围内实现的。利用Recursion的机器学习和计算专长以及英伟达的技术,我们将这些进步层层叠加在一起,来预测这个庞大的化学宇宙中的每种分子如何与蛋白质宇宙相互作用。”
评论褒贬不一
并不是每个人都对AlphaFold彻底改变药物发现持乐观态度。就在Recursion宣布这一消息的前一天,加州斯坦福大学的一个科学家团队在eLife上发表了一篇论文,指出AlphaFold在预测蛋白质结构方面的优势还不能转化为配体结合研究的可靠leads。

斯坦福大学的生物物理学家、该论文的另一个作者Masha Karelina说:“像AlphaFold这样的模型在蛋白质结构方面确实很出色,但我们需要考虑如何将它们用于药物发现。”
接受Nature杂志采访的其他人也认为,这类工作提供了大量令人印象深刻的数据,但他们还不能确定这些数据的质量。像Recursion这样的生物技术公司通常不会提供验证数据,即从实验室实验中证实模型准确预测了结合。计算出的相互作用也是基于预测的蛋白质结构,而不是实验确定的蛋白质结构,这种结构可能不具备原子级分辨率,而药物开发人员需要这种分辨率来精确定位可能发生最强结合的位置。
加利福尼亚大学旧金山分校的药物化学家Brian Shoichet说:“更重要的是,预测的相互作用的数量之大(Recursion预测了28万亿次)意味着,即使是很小比例的假阳性hit也会导致代价高昂的延误,而科学家们却要浪费宝贵的时间来验证它们。结果是令人兴奋的,但也有很多问题。”
面临的问题
南加州大学洛杉矶分校的计算生物学家Vsevolod Katritch说:“在药物发现中使用计算工具的理念是’更容易、更快、更便宜地处理好药物的所有参数’。通过使用人工智能模型寻找leads,制药公司可能只需要在实验室测试几百种化合物,而不是几千种。这可以节省数百万美元的成本,并在数年而不是数十年内将化合物推向市场。”
AlphaFold和类似的程序,例如由华盛顿大学西雅图蛋白质设计研究所研究人员领导的国际团队开发的RoseTTAFold,有望进一步撼动制药业,因为许多人类蛋白质的结构一直缺乏,导致很难找到治疗某些疾病的方法。在去年存入数据库的2亿个蛋白质结构中,欧洲分子生物学实验室的欧洲生物信息学研究所认为35%的结构高度准确–与实验确定的结构一样好,另外45%的结构准确度足以满足某些应用的需要。
Karelina说:“从AlphaFold和RoseTTAFold的蛋白质结构到配体结合的预测,从表面上看似乎并不是一个很大的飞跃。”她最初认为,模拟小分子如何与预测的蛋白质结构”对接”(通常涉及估算释放的能量)会很容易。但当她着手进行测试时,却发现与AlphaFold模型对接的准确性远远低于与实验确定的蛋白质结构对接的准确性。Karelina还不能完全确定原因,但她认为,模型中氨基酸侧链的方向与实验结构的方向之间的微小变化可能是造成差距的原因。当药物结合时,它们也会轻微改变蛋白质的形状,而这正是AlphaFold结构所不能反映的。
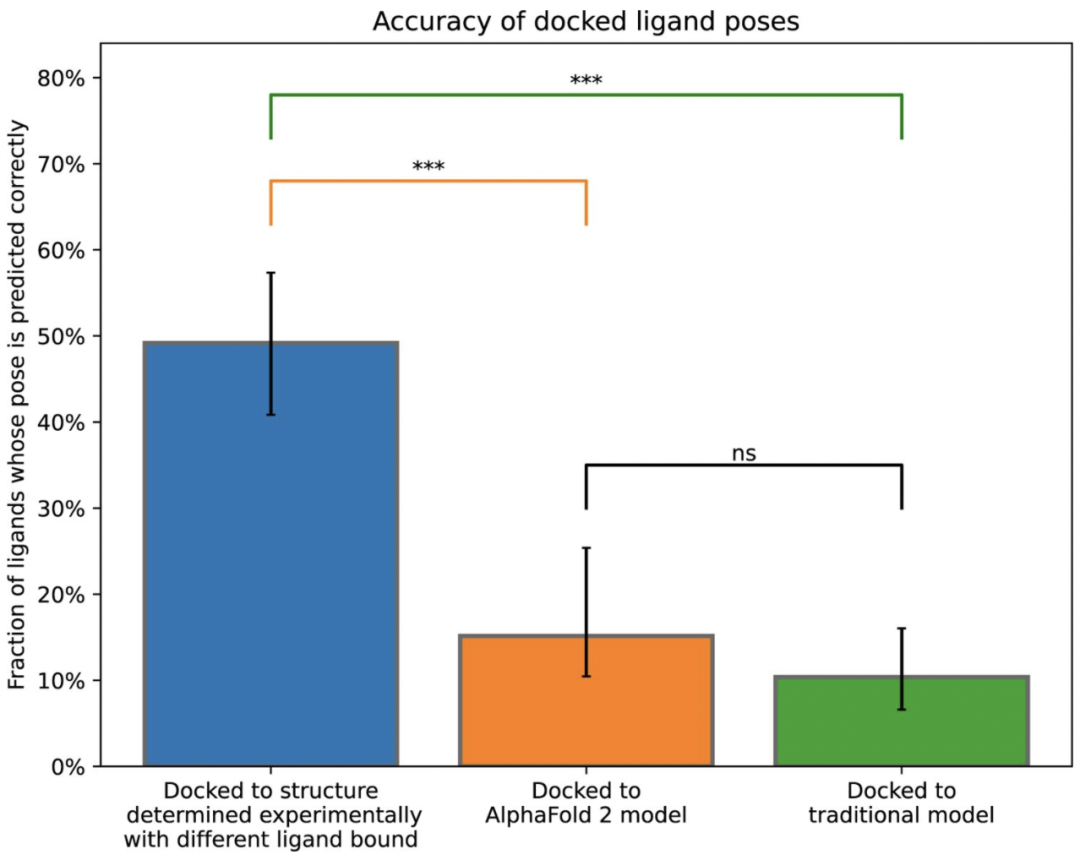 通过计算对接 AlphaFold 2 模型、传统模板模型或实验确定的与不同于对接配体的配体复合的蛋白质结构,预测配体结合位置的准确性。
通过计算对接 AlphaFold 2 模型、传统模板模型或实验确定的与不同于对接配体的配体复合的蛋白质结构,预测配体结合位置的准确性。
总部位于伦敦的Charm Therapeutics首席执行官兼联合创始人Laksh Aithani同意Karelina的研究结果,认为RoseTTAFold和AlphaFold在确定小分子对接时表现不佳。
Charm正在尝试一种不同的方法来评估蛋白质与药物的结合。这项技术使用了一种名为DragonFold的人工智能工具,该工具以RoseTTAFold为骨架。Aithani说,它可以模拟蛋白质和配体结合在一起时的三维形状,从而使Charm能够考虑到配体结合时蛋白质形状的变化,并对可能的药物进行修改,以产生更紧密、更具选择性的结合。Aithani还没有透露太多的细节,但他说这个项目已经吸引了制药公司百时美施贵宝的兴趣。
未来的路
Shoichet说:“这些研究小组面临的挑战最终不是设计一个能识别分子结合程度的模型,而是创建一个能识别与蛋白质强结合的化合物的系统,而我们对这些蛋白质知之甚少。要想取得进展,就必须在实验室中进行验证。”
剑桥麻省理工学院的数学家Bonnie Berger说:“工业界应该能够进行验证。但目前,如果企业正在进行验证,也不会共享这些数据。像Recursion这样的公司缺乏透明度。”
Recursion回应到:已经在最近的两项研究中分享了MatchMaker的验证数据。
Recursion发言人Ryan Kelly 说:“实时分享这些令人兴奋的技术里程碑,是我们与社区和广大公众分享药物发现思路的方式。”
Berger表示,像AlphaFold这样的竞赛不仅有助于推动药物发现的发展,还能让更多人了解业界的方法。AlphaFold在2020年两年一度的蛋白质结构预测关键评估(CASP)竞赛中获胜,成为头条新闻,在该竞赛中,研究人员必须用一组已通过实验确定结构但尚未公开发布的蛋白质来测试他们的预测模型。同样,人工智能工具的药物-蛋白质相互作用结果也可以与实验室的结合结果进行比较。
Shoichet表示:为了利用AlphaFold等模型进行药物发现,我们正在进行大量的努力。但是,一切都还处于起步阶段。
参考资料:
https://www-nature-com.2730.top/articles/d41586-023-02984-whttps://ir.recursion.com/news-releases/news-release-details/recursion-bridges-protein-and-chemical-space-massive-protein
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


