今天为大家带来的是一篇于2023年发表在Applied
Intelligence上的一篇关于改进蛋白质相互作用预测的论文。蛋白质相互作用(PPIs)的预测在许多生物学应用中至关重要,包括蛋白质功能鉴定、药物设计和疾病检测。本文提出了一种结合特征的蛋白质表示和直接从蛋白质序列预测PPI的方法。首先利用5种蛋白质序列提取器提取手工特征。接下来应用自然语言处理技术Word2vec,通过将蛋白质序列嵌入到特征空间中来生成学习特征。最后,采用深度神经网络结构对两类特征进行组合识别。

1.介绍
PPIs在生物体的细胞系统中起着重要的作用。因此,PPIs的鉴定可以提供生物学进展的全局图景,并有助于获得对蛋白质功能、生物过程、药物设计和其他生物学应用的生物学见解。用于PPI鉴定的高通量生物学和大规模实验为PPI网络提供了有价值的信息,但在确定不同物种之间的PPI时昂贵、耗时且有限。因此,精确而强大的PPI预测计算方法被提出并进行了广泛的研究。计算方法可为生物实验设计提供参考和指导,有助于实验室验证。其中一个受到广泛关注的基于计算的方法是机器学习方法。
基于机器学习(ML)的方法是基于不同来源的生物信息开发的,如蛋白质序列、蛋白质结构信息、基因本体注释和蛋白质的语义相似度。此外,蛋白质序列数据正在迅速增长,这比其他生物信息来源更有优势。
基于ML的直接从蛋白质序列中预测PPI的方法有两个主要目标:一是创建有效的方法将氨基酸序列转换为特征向量;二是设计一个鲁棒的ML模型,准确快速地将蛋白质对分类为相互作用或非相互作用类。蛋白质序列表示方法可以分为两组,一组是手工制作的特征,即由数据科学家手工设计的特征,另一组是学习的特征,即从机器学习算法中自动获得的特征。
到目前为止,许多基于ML方法的工作已经创建了各种仅从蛋白质序列预测ppi的高性能模型。这些方法的成功主要依赖于表示蛋白质序列和选择合适的学习模型。手工制作的特征已经实现了几十年,当与机器学习分类器结合使用时,仍然是一个强大的工具。同时,学习到的特征对于解决特定的任务非常有效。结合这两种类型的特征来表示蛋白质序列比只使用一种特征更能提高性能。受此启发,本文提出了是否有可能有效地结合这两种类型的特征以及它对预测PPI的有效性的想法。
2.材料与方法
1.数据集
在本文中使用了10个基准PPI数据集。首先,采用DIP的酵母核心数据集。在该数据集中去除氨基酸数小于50且序列同一性大于等于40%的氨基酸序列,选择5594对蛋白质相互作用对作为阳性样本,5594对具有不同亚细胞位置信息的蛋白质相互作用对作为阴性样本。其次,人类数据集来自人类蛋白质参考数据库,其中包含3899对阳性蛋白和4262对阴性蛋白。
第三,从DIP数据库下载5个PPI跨物种数据集,构建独立数据集。在这些数据集中,仅保留直接物理相互作用对,包括秀丽隐杆线虫(Celeg),大肠杆菌(Ecoli),智人(Hsapi),幽门螺杆菌(Hpylo)和小家鼠(Mmusc)。这些数据集分别由4013对交互对、6954对交互对、1412对交互对、1420对交互对和313对交互对组成。第四,三个ppi网络数据集由One-core网络(CD9)、Wnt相关通路交叉网络(Wnt)和Cancer特异性网络(Cancer)组成。这3个PPI网络数据集和5个PPI跨物种数据集被构建为独立的测试数据集。
2.方法
本文所提出方法的流程如图1所示。在步骤1中输入蛋白质序列对,每个序列在步骤2中被提取成手工制作的特征。同时,每个序列在步骤3a中进行预处理,通过预训练的Word2vec模型生成步骤3b中学习到的特征。在步骤4中,使用DNN模型组合输入特征,最终将蛋白质对分类到所需的类别中。在步骤5中使用上述数据集评估分类性能。
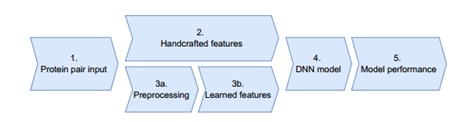
图1 方法流程图
3.手工提取的特征
为了将蛋白质序列转化为特征向量,如图2所示文章构建了序列转换模块。序列转换模块包括5种特征提取方法,氨基酸组成(AAC)、伪氨基酸组成(PseAAC)、两亲性PseAAC (APAAC)、准序列顺序(QSO)和二肽组成(DPC)。AAC和DPC是描述蛋白质序列中氨基酸组成频率的两种方法。AAC和DPC方法的优点是简单,除了序列外不需要其他信息。此外,与CTD相比,AAC和DPC产生的特征向量维数、多尺度连续和不连续都不大,特征转化时间短。氨基酸序列的顺序是一个重要的因素,改变顺序会产生不同的蛋白质。利用QSO方法捕捉这一重要信息。每个氨基酸具有不同的物理化学性质,因此使用这些性质来表示蛋白质序列可以帮助模型更好地区分不同的蛋白质序列。由此,利用PseAAC和APAAC方法来描述氨基酸的理化信息,以及反映蛋白质序列内所有氨基酸对之间序列顺序相关性的因子。
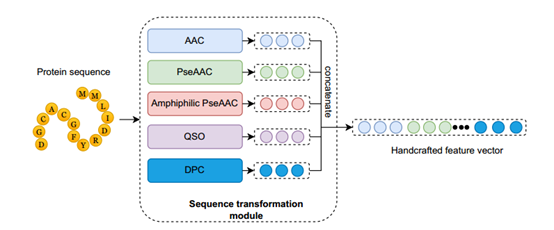
图2 序列转换模块的工作流程
这五种方法的构建是为了将蛋白质序列转化为手工制作的特征,这些特征能够描述来自蛋白质序列的各种信息。
4.学习到的特征
为了利用氨基酸之间的语义关系作为表示蛋白质序列的有用信息,本文使用Word2vec模型来学习这种关系。Word2vec模型已被证明对各种NLP任务非常有用,Word2vec模型可以利用语料库中词的共现信息来反映词义。为了应用这种方法,本文将蛋白质序列表示为一个句子,并将一组蛋白质序列表示为语料库。为了表示单词,把氨基酸序列分成若干单位。.
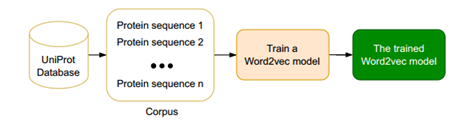
图3 Word2vec模型训练过程
经过训练的Word2vec模型可用于将蛋白质序列编码为数字特征向量,称为学习特征。因为蛋白质序列的长度是可变的,所以此模型需要固定蛋白质序列的长度。
本文中用N的值固定蛋白质序列的长度。长度大于N的氨基酸序列将向右截断;否则,长度小于N的氨基酸序列在其最终向量上加零值。这样就有了一个固定长度的学习特征向量来表示一个蛋白质序列。训练Word2vec模型和转化蛋白序列的整个过程如图3和图4所示。这些特征与手工制作的特征相结合,用于馈送神经网络。
图4 蛋白质序列转化过程
5.深度学习和注意机制
深度学习是一种试图模拟人类大脑行为的技术,使其能够从大量数据中学习。深度学习是生物信息学领域的一种成功方法。神经网络的主要架构,如DNN、CNN和RNN,可用于预测PPI。然而,一些蛋白质序列非常长,例如酵母核心和人类数据集中最长的序列分别包含4,910和33,423个氨基酸。因此,在使用RNN时,将蛋白质序列视为一个以氨基酸为单词的句子会导致非常长的训练时间问题或梯度消失问题。CNN可以很好地利用特征之间的空间关系,例如图像中像素的位置或序列顺序信息。事实上,CNN可以用来利用学习到的特征。然而,学习到的特征不包含其元素之间的位置关系,序列顺序信息已经通过QSO方法编码在手工制作的特征中。DNNs是一种具有大量层数的神经网络。深度神经网络的主要作用是提取高级特征、消除噪声和降低数据维数。此外,深度神经网络适用于不同的应用。因此本文选择DNN作为设计PPI预测模型的架构,命名为DeepCF-PPI。DeepCF-PPI架构如图5所示,该架构包括三个主要模块:特征提取、特征组合和分类。
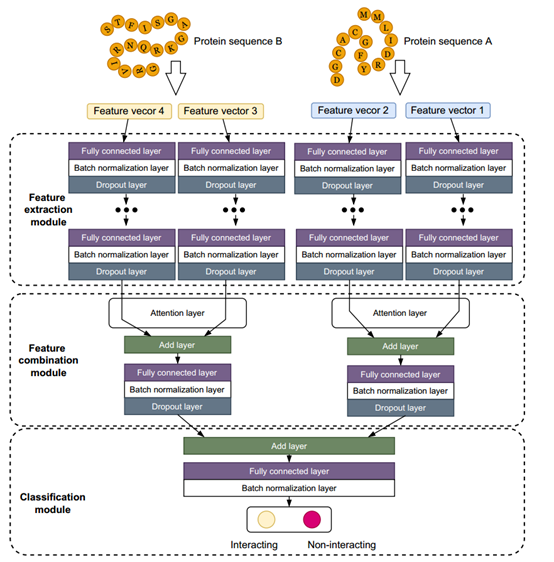
图5 DeepCF-PPI架构的总体框架
特征提取模块特征提取模块有四个分支,接收四个原始特征向量作为输入,将每个原始特征向量传递到每个分支。这些分支具有相同的参数,即神经元数量、学习率、批处理大小和激活函数。每个分支都由层叠的组组成,每个组有三个层:完全连接层、批处理规范化层和Droupout层。
全连接层也被称为密集层或前馈层,其中每个神经元接收来自前一层的所有神经元的输入。批规范化层对输入进行标准化。架构使用这一层来提高神经网络的训练速度。Dropout是神经网络避免过拟合的一种简单而强大的正则化技术。这种技术在一层中随机抽取一些神经元。
为了学习输入之间的非线性关系,在全连接层之后加入ReLU激活函数。如果ReLU函数的输入为正,则将其直接输出,否则将输出零,从而引入非线性。ReLU函数使神经网络的训练变得更容易,并且往往能达到更好的性能,因此它已经成为许多类型神经网络的默认激活函数。
本文提出的方法的深度神经网络使用两种类型的特征作为其输入,包括手工特征和学习特征。从不同方法提取的特征类型可以保存不同重要程度的信息。因此需要使用注意力机制来关注重要的功能。架构通过注意层将注意机制集成到特征组合模块中。该模块由Attention层、Add层、全连接层、批规范化层和Dropout层组成。特征组合模块将其输入特征向量合并为单个特征向量,并由注意层学习表征输入特征向量重要性的注意权值。
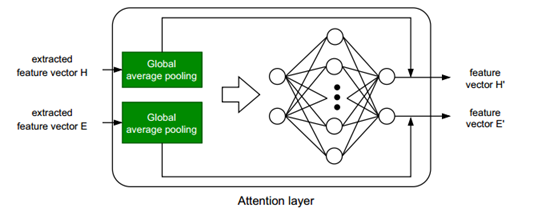
图6 Attention层的结构
注意层的架构如图6所示。注意层接收前两个DNN特征提取模块的两个输出向量,使用全局平均池化层对两个向量生成统计,再传递到由两个完全连接层组成的子网络中,分别有8个神经元和2个神经元。得到特征向量的注意权值后,将向量乘以它们的注意权值,得到两个新的向量。
Add层将前一层的两个输出作为输入,其输出是其输入的总和。Add层的输出随后被传递到下一层。最后,构建代表输入蛋白对的特征向量,并由分类模块用于预测交互作用。
分类模块分类模块由Add层、全连接层和批处理归一化层组成,然后是一个带有两个神经元的输出层。输出层使用2类softmax激活函数,判断蛋白质是否能相互作用。
6.模型训练
在本方法中有两个模型需要训练,包括Word2vec模型和DNN模型。这两个模型是相互独立训练的。
训练好的Word2vec模型用于将蛋白质序列转换为学习到的特征向量。为了得到代表氨基酸的特征嵌入,本文在Swiss-Prot数据库上训练了一个模型,其中包括562,253个蛋白质序列。在酵母核心数据集上探讨了不同参数组合对PPI预测的影响。本文使用Gensim库实现了Word2vec。
蛋白质序列长度是直接影响模型性能的一个因素。在实验中,选择N的值作为训练集中蛋白质序列的平均长度。如果N的值很大,相当于在特征向量上加了很多个零,导致生成的特征向量相对相似。若N的值很小由于序列截断操作,会丢失大量的序列信息。
选择N值后,使用分类交叉熵损失函数和Adam梯度下降优化算法对DNN模型进行训练。将Adam梯度下降优化算法的学习率设置为0.001,衰减值设置为0.001,其他参数均设置为默认值。该框架的实现是在Python库上进行的,包括TensorFlow和Scikit-learn。
3.结果
1.实验设置
使用酵母核心数据集为所提出的模型找到最佳配置。将优化配置的DeepCF-PPI模型与其他已有模型在10个基准数据集上进行对比。进行了两个实验:一是在酵母核心和人类数据集上使用交叉5倍交叉验证方法;二是在其余8个数据集上独立测试模型的性能。
在第二个实验中使用酵母核心数据集的所有样本来训练所提出的模型,然后比较了在8个数据集(Celeg、Ecoli、Hsapi、Hpylo、Mmusc、CD9、Wnt和Cancer)上预测的模型的准确性。
2.超参数选择
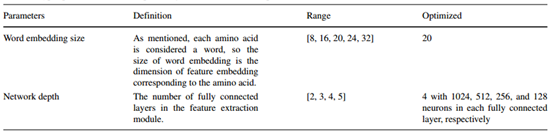
有两组超参数需要确认:用于训练Word2vec模型的超参数集和用于配置神经网络的超参数集。由于不同的超参数组合呈指数增长,并且在实践中不可能尝试所有的超参数,因此作者只选择了两个最重要的超参数:词嵌入大小和网络深度。使用词嵌入大小超参数控制训练Word2vec模型,使用网络深度配置神经网络。表1显示了实验中超参数的值。
表1 用于优化网络的超参数和最优值
本文将酵母核心数据集分成三个比例,70%用于训练集,10%用于验证集,20%用于测试集。这个过程重复了五次。然后通过比较测试集上的准确性来评估最佳模型配置。表2显示了此方法在不同网络深度和词嵌入大小组合下对酵母核心数据集的预测性能。
表2 此方法在不同组合的酵母核心数据集上的预测性能
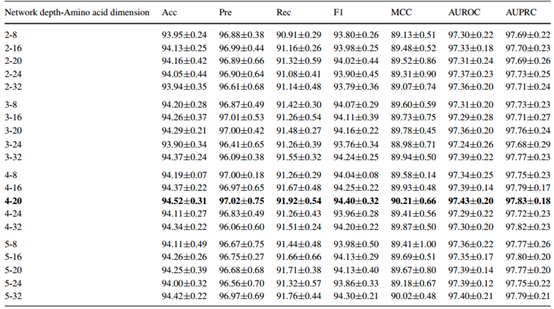
实验结果表明,当词嵌入大小为20,网络深度为4时,模型性能最佳,平均Acc为94.52%,平均Pre为97.02%,平均Rec为91.92%,平均MCC为90.21%。F1、AUROC和AUPRC的其他指标分别为94.40%、97.43%和97.83%。
3. 与传统机器学习模型的比较
本文通过五重交叉验证,将DeepCFPPI的预测结果与酵母核心数据集上的六种传统ML模型进行比较,包括朴素贝叶斯(NB)、AdaBoost (Ada)、支持向量机(SVM)、决策树(DT)、k近邻(KNN)和随机森林(RF)。对比结果如图7和图8所示。
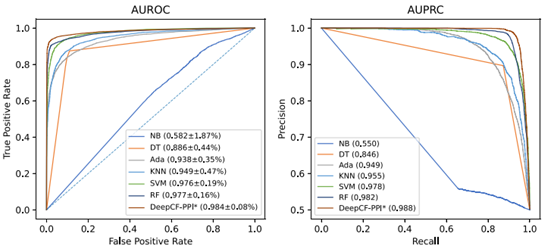
图7 不同模型在酵母核心数据库上进行5倍交叉验证的ROC和PR曲线比较
如图7所示,DeepCF-PPI模型的预测性能优于NB、DT、Ada、KNN、SVM和RF模型。该模型的AUROC和AUPRC值分别比其他6种传统ML模型高0.7% ~ 40.2%和0.6% ~ 43.8%。这些结果表明,本文提出的方法可以准确地检测酵母核心蛋白对之间的相互作用。此外,从结果中可以清楚地看出,将两种类型的特征结合到基于dnn的架构中,在识别ppi方面是有效且有前途的。
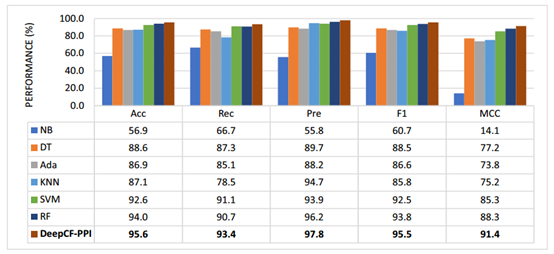
图8 不同模型在酵母核心数据库上进行5倍交叉验证的其他性能指标
4.不同方法在酵母核心数据集上的性能
为了此方法的性能,本文将其与其他现有方法进行了比较。在这项工作中,通过5倍交叉验证,对酵母核心数据集进行了比较方法和DeepCF-PPI模型。各方法对该数据集的预测结果如表3所示。
表3 DeepCF-PPI与其他现有方法在酵母核心数据集上的预测性能比较
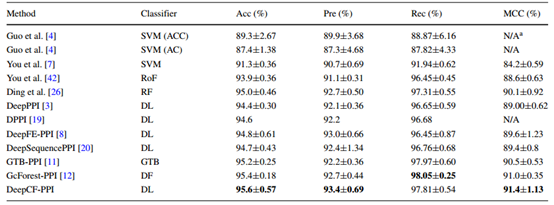
从表3中可以看出,DeepCFPPI模型具有较高的预测性能,Acc为95.6%±0.57%,Rec为93.4%±0.69%,Pre为97.81%±0.54,MMC为91.4%±1.13%。
5.不同方法在人类数据集上的性能
为了进一步证明此方法的有效性,文章进一步将其与人类数据集上的其他现有方法进行了5倍交叉验证。各方法预测结果如表4所示。
表4 DeepCF-PPI与其他现有方法在人类数据集上的预测性能比较
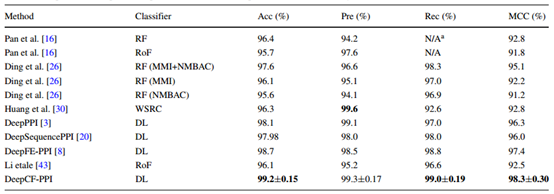
如表4所示,本文提出的方法在Acc(99.2%)、Rec(99.0%)和MCC(98.3%)上的性能最高。与比较方法相比,此方法获得的Acc、Rec和MCC指标分别提高了0.5%-3.6%、0.2%-6.4%和0.9%-7.1%。
6.独立测试数据集的预测结果
为了验证该方法的有效性,在8个独立的数据集上进一步测试了该模型,其中包括5个跨物种数据集和3个PPIs网络数据集。在本实验中使用酵母核心数据集的全部11,188个样本作为训练集,然后在8个独立数据集上获得预测精度。比较模型的通用性在此精度上进行了评价。跨物种数据集的测试至关重要。
表5 不同方法在8个独立数据集上的预测精度
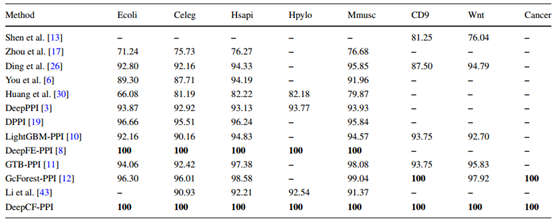
从表5中可以看出,DeepCF-PPI模型的预测精度非常高,对所有五个跨物种数据集的预测精度都达到了100%。与其他方法相比,此方法在5个跨物种数据集上获得了最高的精度,等于DeepFE-PPI,然而DeepFE-PPI模型尚未在其他独立数据集上进行测试。
4.结论
基于机器学习的蛋白质相互作用预测方法已经得到了广泛的发展。为了改进PPI预测,本文提出了一个基于深度神经网络的新框架,该框架能够将手工制作的特征与学习的特征相结合,直接从它们的氨基酸序列中预测PPI。利用该框架,蛋白质序列可以通过强大的蛋白质描述符和自然语言处理中的词嵌入技术来表示。
蛋白质描述符从序列信息中捕获物理化学和组成特征,而词嵌入技术捕获蛋白质序列中氨基酸之间的语义关系。将这两种类型的特征集成到一个深度学习架构中,可以有效地表示蛋白质序列,从而提高PPI的预测性能。
结果表明,多种信息在蛋白质表示中的组合对于预测蛋白质之间的相互作用非常有用。DeepCF-PPI模型总体上优于现有方法,特别是在泛化能力方面。在不同数据集上的成功预测表明,本文提出的蛋白质表示方法比其他特征提取方法具有更好的性能,并且此模型在许多PPI预测任务中是有用的。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


