2023年10月17日,哈尔滨工业大学(深圳)李君一团队在Bioinformatics上发表文章Struct2GO: protein function prediction based on graph pooling algorithm and AlphaFold2 structure information。
 作者提出了一种新的基于图池化算法和AlphaFold2结构信息的蛋白质功能预测器(protein function prediction based on graph pooling algorithm and AlphaFold2 structure information)。
作者提出了一种新的基于图池化算法和AlphaFold2结构信息的蛋白质功能预测器(protein function prediction based on graph pooling algorithm and AlphaFold2 structure information)。
Gene Ontology(GO)数据库是目前最常用的描述基因和蛋白质功能的数据库。本文利用蛋白质结构信息对GO数据库中的蛋白质功能进行预测,因而称为Struct2GO。作者通过图表示学习获得蛋白质结构中的氨基酸残基嵌入,利用基于自注意力机制的图池化算法获得整个图结构特征,并将其与蛋白质语言模型获得的序列特征融合。结果表明,与传统的基于蛋白质序列的功能预测模型相比,Struct2GO模型取得了更好的结果。
背景
AlphaFold2模型将蛋白质结构预测的精度提高到了原子水平,与之相比,基于深度学习的蛋白质功能预测模型通常是从蛋白质序列中提取特征,并将其与蛋白质-蛋白质相互作用网络相结合,以获得较好的结果。然而,对于未在蛋白-蛋白相互作用网络中的新测序蛋白,这种模型无法进行有效的预测。针对这一问题,本文提出了结合蛋白质结构和序列数据的Struct2GO模型,以提高蛋白质功能预测的精度和模型的通用性。
方法
蛋白质功能预测问题可以看作是一个多标签的二值分类问题,即通过提取给定蛋白质的特征并将其映射到蛋白质功能标签的空间。可利用多种数据源获取蛋白质功能预测特征,如蛋白质序列、蛋白质结构、蛋白质家族、蛋白质-蛋白质相互作用网络等。最常用的信息源是蛋白质序列和相互作用网络。蛋白质功能标签可以通过Gene Ontology数据库获取。根据功能范围的不同,包括三个独立的分支:细胞成分(Cellular Component Ontology,CCO)、分子功能(Molecular Function Ontology,MFO)和生物过程(Biological Process Ontology,BPO)。
本文从EMBL-EBI数据库中获得了AlphaFold2预测的人类蛋白质结构数据,包括23391个蛋白质结构。本文从与人类蛋白质对应的GO数据库标注中筛选出56万余条数据,并通过实验获得标注,即提取“IDA”、“IPI”、“EXP”、“IGI”、“IMP”、“IEP”、“IC”或“TA”的证据码(evidence Code),其中人类数据集包括20395条数据。同时,下载并解析了官方基因本体网站发布的最新基因本体数据,根据解析的BPO、CCO、MFO分支构建了有向无环图,并完成了标签。需要注意的是,大多数功能术语在数据集中没有出现或只注释了少数蛋白质,因此本文对每个分支以低于一定阈值的频率过滤掉GO名词,以降低标签的稀疏性。完成后BPO、MFO、CCO标签数量分别为809个、273个、298个。
蛋白质的结构与功能密切相关。为了更好地从蛋白质结构信息中推断蛋白质的相关功能,本文将三维蛋白质结构转化为二维蛋白质接触图,构建蛋白质结构网络,对相邻残基信息进行聚合,最终获得蛋白质结构特征。如图1所示。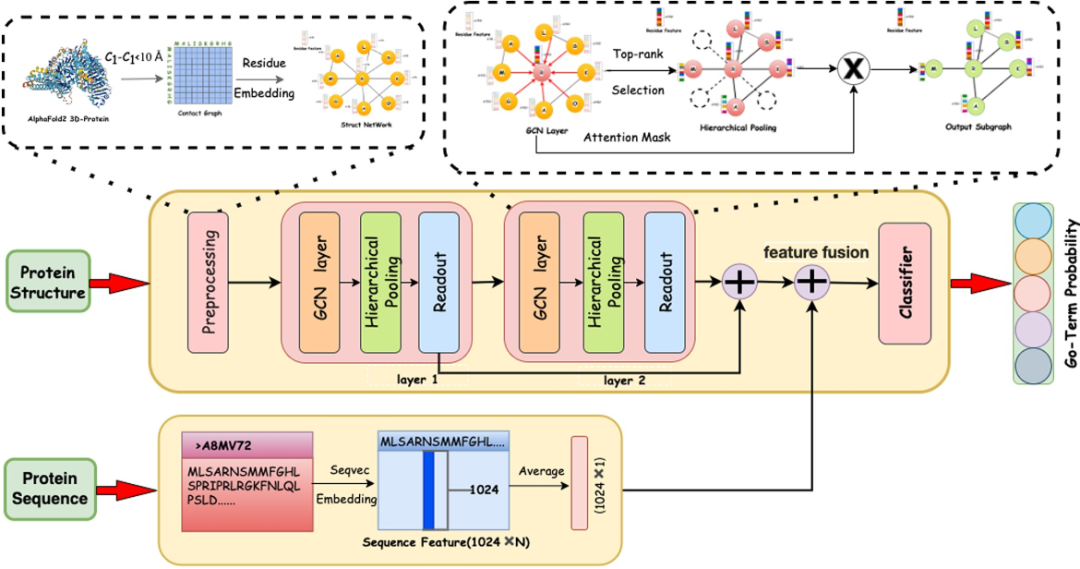 图1 Struct2GO结构图
图1 Struct2GO结构图
在具体实现方面,可以通过AlphaFold2获得蛋白质结构的三维原子坐标,然后计算氨基酸残基之间的相对距离。如果它们之间的Cα原子小于10埃,则认为两个残基之间存在直接连接的边。
在蛋白质结构网络中,每个节点都是一个氨基酸残基。为了获得节点的特征,最直观的方法是对20种不同的氨基酸进行独热编码,但这种方法无法捕获同一氨基酸在不同蛋白质网络中的位置信息。因此,本文利用图表示学习中的Node2vec模型来获取蛋白质网络中节点的结构信息。在具体实现上,本实验利用Spark提供的高效的数据存储、更新和共享服务,实现了用于图计算的node2vec算法,为蛋白质中的每个残基生成50维的特征向量。
在自然语言领域,BERT和XLNet等预训练大语言模型近年来发展迅速,许多研究人员将NLP领域的模型扩展到生物序列领域,提出了多种获取蛋白质序列分布式表示的预训练模型,其中SeqVec模型被广泛应用。具体来说,SeqVec模型首先在UniRef50数据库中预训练了大约33M个序列,然后使用CharCNN算法获取氨基酸的局部特征,然后使用BiLSTM算法构建语言模型。通过对场特征和语言模型进行平均,得到单氨基酸特征。最后,SeqVec模型将残基特征连接到一个矩阵,通过主成分分析或平均聚合生成矩阵的降维数矩阵。对于每个蛋白质序列,我们可以得到一个特征向量作为蛋白质序列特征,并结合其结构特征在后续模型中进行下游蛋白质功能预测。
由于同一蛋白质可能具有多种功能,因此该模型本质上是一个多标签分类任务。本文采用基于关注的图池化机制,将上述得到的蛋白质接触图和氨基酸残基特征作为输入,通过图卷积和分层池化提取蛋白质结构特征,并将上述序列特征集成为下游蛋白质功能预测多标签分类器的输入。同时,分类器中的网络层和后处理层保证了GO标签之间的层次关系。
图卷积层将蛋白质接触图作为邻接矩阵,将氨基酸残基特征作为图中的节点特征,通过图卷积在结构相似的残基和结构之间传播其特征。在每一层,通过邻居消息的传播和聚合得到一个新的隐藏表示。
近年来,自注意机制被广泛应用于深度学习模型中,使模型能够更多地关注重要特征。SAGPool将自注意力方法引入到图池化模型中,通过堆叠卷积层并将输出特征转换为一维来获得每个节点的重要性分数,用节点选择算法保留输入图的一些节点和边,生成新的子图作为下一层的输入。池化比率确定将保留的节点数量,然后选择节点由自注意卷积层得到的每个节点的重要度得分。本文使用双头注意机制,分别获得每个节点的两个重要性分数,并计算其平均值作为最终分数。接着,通过拼接和池化和最大池化来提取这一层的图特征,最后将多层的图特征求和作为蛋白质的结构特征。
结果
作者将Struct2GO与一些具有代表性的方法进行了比较。表1列出了三个不同的指标,包括AUC, AUPR和宏F1值(Fmax)。对于BPO, CCO, MFO这所有的三个分支,Struct2GO在各个指标上超越了现有方法。
表1 与其他方法对比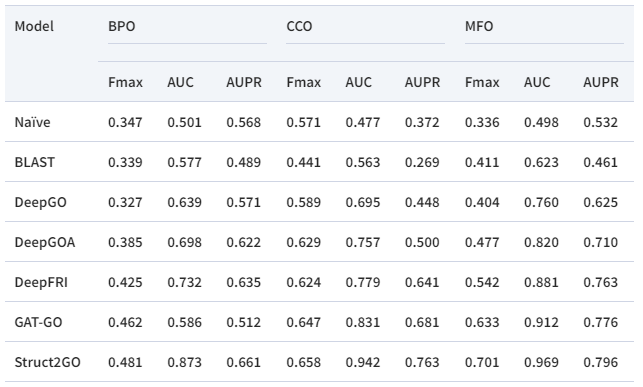
作者设计了模型消融实验,在四种变体中删除模型的不同部分:(i)没有结构信息,(ii)没有独热编码, (iii)没有Node2vec,(iv)没有序列信息(预训练大语言模型)。如表2所示,Struct2GO的表现超越了以上各个变体,表明模型中的各个组成部分均能有效提升模型性能。
表2 消融实验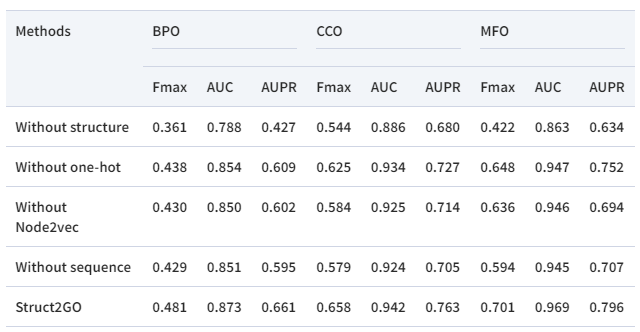
作者还进行了案例分析,在蛋白质图上选择不同的建图策略,如图2所示。本文采用CA-CA方法进行建图:如果两个残基之间的Cα原子小于10埃,则认为两个残基之间存在直接连接的边。
在案例分析中,作者讨论了两种不同的建图方法。
1) ANY-ANY:如果两个残基的任意原子中的距离小于10埃,则认为两个残基之间存在直接连接的边。
2) NBR (neighbor radius):如果两个残基的Rosetta相邻原子之间的距离小于氨基酸对相邻半径的和,则认为两个残基之间存在直接连接的边。除了使用Cα的甘氨酸外,所有氨基酸的Rosetta相邻原子都定义为Cβ原子。
图2表明,使用ANY-ANY方法会使得过多的原子被认为是接触的,失去了有效过滤有意义的相互作用的能力。相比之下,CA-CA和NBR方法取得了更好的Fmax结果,其中NBR略高于CA-CA,因为该方法更直接地考虑了氨基酸之间的侧链关系。然而,NBR需要使用RosettaFold来识别出Rosetta原子,所需要的计算时间远高于CA-CA,因此,本文采用CA-CA方法进行建图。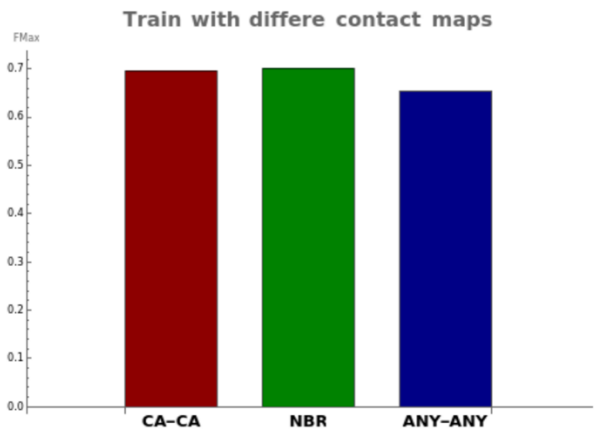 图2 案例分析
图2 案例分析
总结
在本文中,作者提出了一个强大的端到端图深度学习模型Struct2GO,该模型可以基于蛋白质结构和序列有效快速地标注蛋白质功能。具体而言,Struct2GO采用图池化模型从AlphaFold2预测的三维蛋白质结构中获取结构特征,并整合SeqVec提取的序列特征来训练蛋白质功能分类器。AlphaFold2预测的三维蛋白质结构数据为蛋白质功能预测提供了强有力的支持,可以有效提高模型的通用性。同时,与以往基于实验确定的蛋白质结构预测蛋白质功能的方法相比,AlphaFold2提供了足够的高分辨率结构信息,有效提高了预测的准确性。对比实验表明,Struct2Go达到了最先进的性能,从而最终证明了结构信息对蛋白质功能预测的有效支持。
在未来的工作中,可以继续研究新的方法,提高Struct2GO模型的通用性和精度。(1)AlphaFold2网站提供了2.17亿个多物种的蛋白质结构数据集,可以在未来的研究中用于尝试大规模的跨物种蛋白质功能模型训练,有效地提高模型的通用性。(2)为了在未来的工作中更多地关注细微的结构变化对蛋白质功能预测的影响,可以探索蛋白质结构特征提取的新方法。(3)可以研究将从序列模型中提取的氨基酸特征嵌入到蛋白质结构网络中,并探索新的随机游走模型,以更全面地挖掘蛋白质结构中的有价值信息。
(4)还可以构建基于结构相似性的蛋白质网络,以单个蛋白质为节点,利用网络传播中同源蛋白质的有效信息来提高模型预测的准确性。
参考文献
[1] Jiao et al. Struct2GO: protein function prediction based on graph pooling algorithm and AlphaFold2 structure information. Bioinformatics. 2023
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


