
药物的发现是昂贵、耗时并且科学上具有挑战性的。为了提高临床前药物发现路径的效率,计算药物发现方法,尤其是最近的基于机器学习的方法,越来越多地被用作为早期药物发现的有力工具。
在这篇论文中,我展示了三种互补的计算机辅助药物发现方法,重点是为了帮助发现新的潜在药物和优化潜在药物的性质。此外,这篇论文特别关注用于特征化机器学习模型的不同分子表示方法,以探索如何最好地捕获关于蛋白质、配体和3D蛋白质-配体复合体的有价值信息,以构建更稳健、更可解释和更准确的机器学习模型。
首先,我使用高斯过程(GP)开发了基于配体的模型,作为一个简单易行的工具来指导化学空间的探索,以优化蛋白质-配体的结合亲和力。我探索了不同的拓扑指纹和自编码器表示法进行贝叶斯优化(BO),并表明BO是一个强大的工具,可以帮助药物化学家优先考虑哪些新化合物用于单目标和多目标优化。当在一个众所周知的药物靶标基质金属蛋白酶-12的基准数据集和一个针对四种细菌金属-β-内酰胺酶的实际正在进行的药物优化数据集上进行测试时,该算法实现了对顶级化合物的高度富集。
接下来,我展示了一种基于知识的药物设计方法,结合新的蛋白质-配体相互作用指纹和基于片段的药物发现方法,以理解SARS-CoV-2 Mpro-底物的特异性并在硅内设计新的小分子抑制剂。结合基于片段的药物发现方法,我展示了这种基于知识的相互作用指纹驱动的方法如何揭示有成果的片段增长设计策略。
最后,我扩展了基于知识的接触指纹,创建了一个配体形状的分子图表示法(蛋白质配体相互作用图,PLIGs),以开发新的基于图深度学习蛋白质-配体结合亲和力评分函数。PLIGs在图的节点特征中编码了蛋白质-配体复合物中的所有分子间相互作用,因此它们是简单且完全可以解释的。我探索了与PLIGs结合的各种图神经网络架构,并发现图注意网络比其他GNN架构略微优越,其性能与最知名的蛋白质-配体结合亲和力评分函数相媲美。

论文题目:Machine learning approaches for computer aided drug discovery
作者:Marc Moesser
类型:2022年博士论文
学校:University of Oxford(英国牛津大学)
下载链接:
链接: https://pan.baidu.com/s/1NPQ4J5-Cyb94XmzkQPAXng?pwd=qu5k
硕博论文汇总:
链接: https://pan.baidu.com/s/1Gv3R58pgUfHPu4PYFhCSJw?pwd=svp5
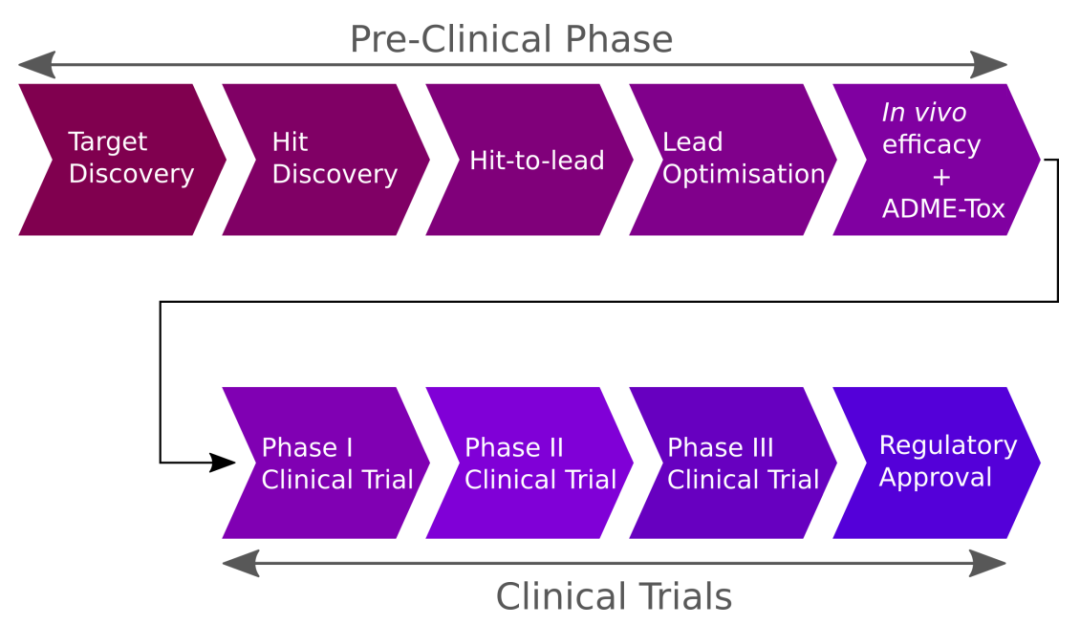
药物发现步骤:1)靶点发现,2)命中发现,3)命中先导,4)先导优化,5)体内疗效及adme毒理学研究,6)人体临床试验,分为3个临床试验阶段。步骤1-5被认为是“临床前”。本文的重点是针对步骤2-4的方法的发展。
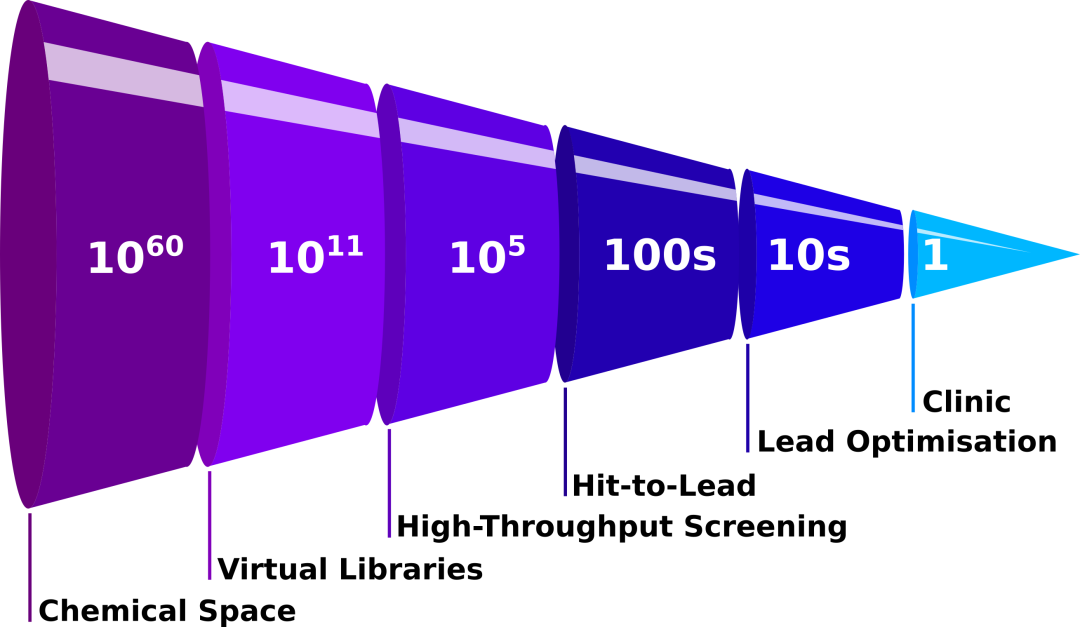
一般概述在一个典型的现代药物发现过程的每一步所使用的化合物的数量。在整个药物类化合物的化学空间中,巨大的虚拟文库被创建和过滤,在高通量筛选过程中创建数十万个化合物的文库,从中选择有前途的化合物并衍生出数百个化合物。然后选择数十种先导化合物,从中选择一种用于临床试验。
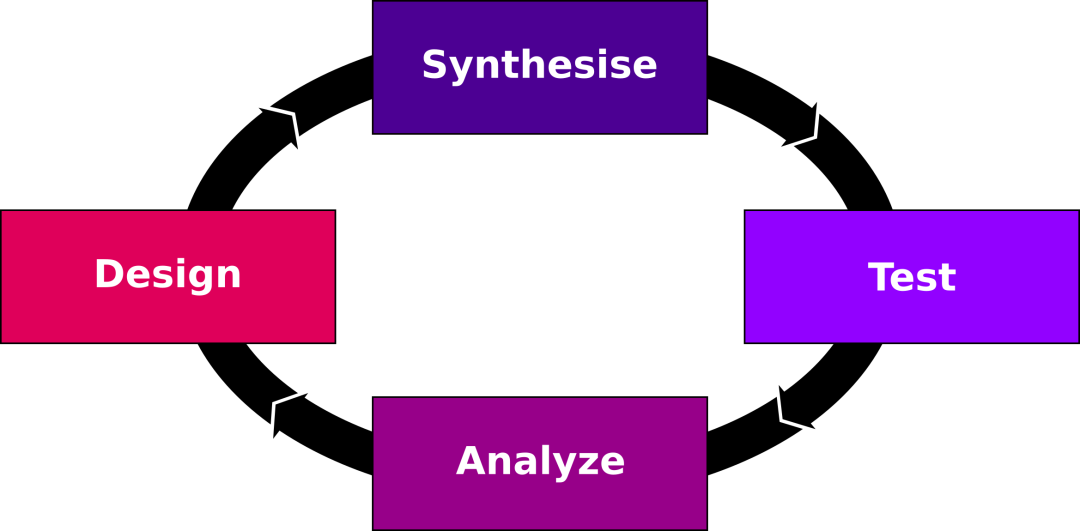
概述设计、制造、测试、分析(DMTA)周期。药物的设计基于一个有效的假设,以提高其性能。然后在实验室合成和测试。分析后,根据新的信息调整设计假设和相应的下一代化合物设计。
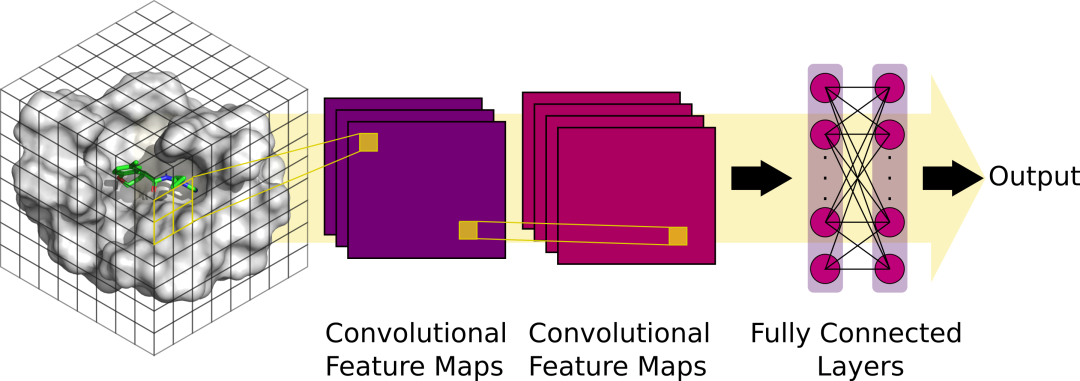
蛋白质-配体结合亲和力预测的3D CNN模型架构示意图。将3D体素化网格作为输入,应用N个不同的卷积来创建N个卷积地图,然后是第二个卷积层,最后是完全连接的层来生成预测。
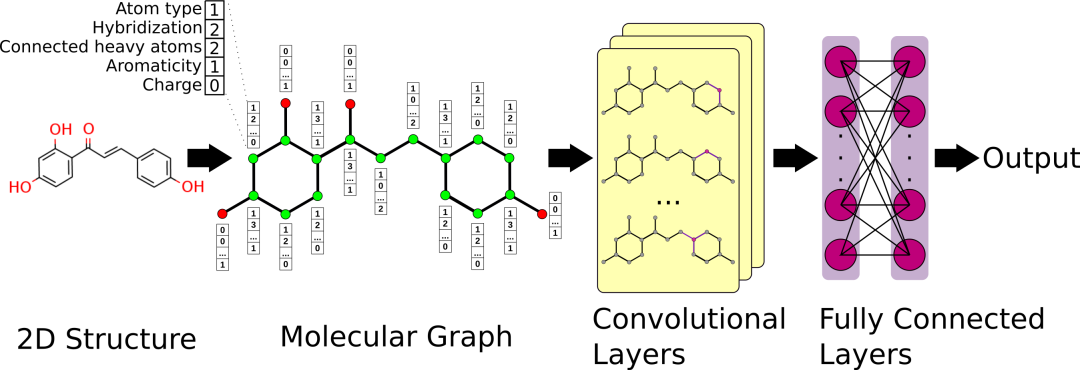
基于配体的图的生成和建立的图卷积网络(GCN)的示意图概述,用于蛋白质-配体结合亲和力预测。配体的二维结构被编码为一个图,其中one-hot编码的原子特征作为图的节点特征(这里的示例显示了5个流行的原子特征,但其他的也可能取决于设置)。GCN模型在图的节点上应用过滤函数,根据设置生成N个卷积层。卷积层连接到完全连接层以生成预测。
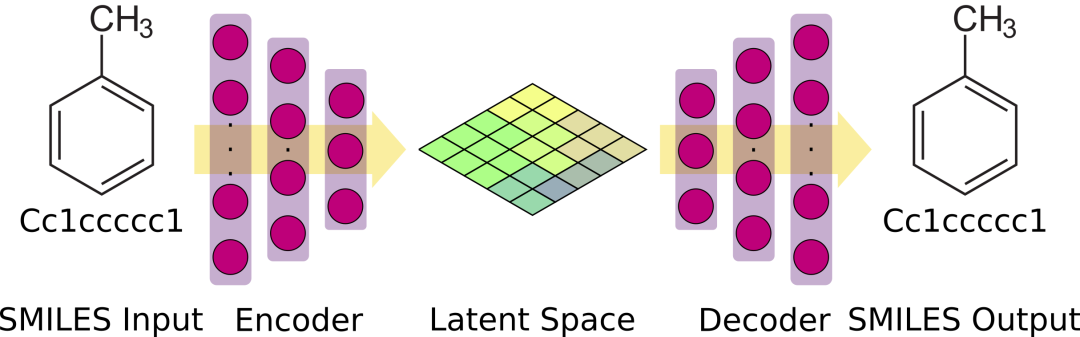
基于smiles的通用自动编码器的编码器和解码器设置示意图概述。训练深度神经网络将SMILES表示与编码器一起编码为潜在空间,编码器将潜在空间表示解码回SMILES。
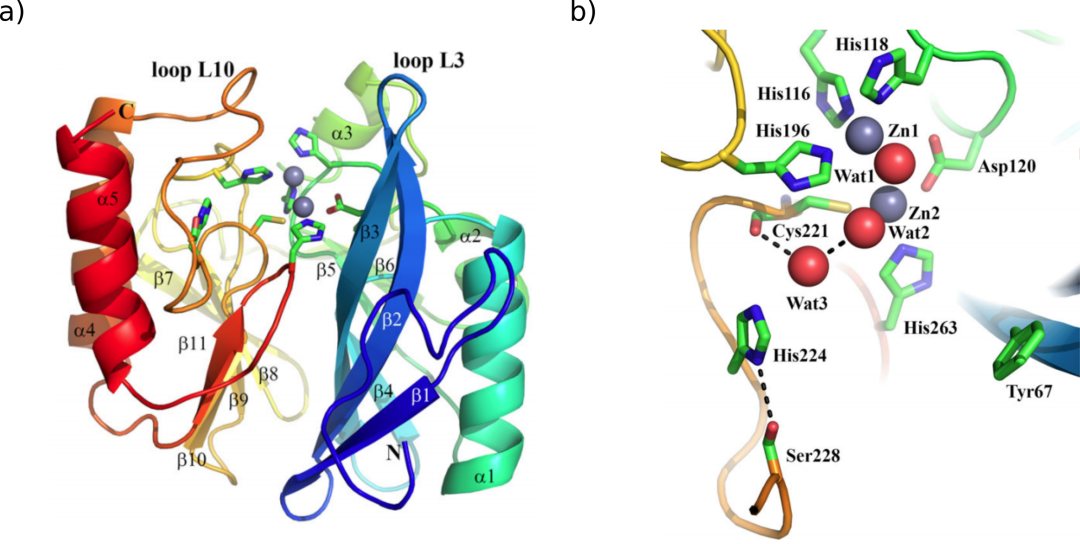
VIM-1晶体结构的视图由Salimraj等人发表(CC by 4.0许可)。(a) VIM-1三级结构,显示整体折叠和活性位点残基,颜色从蓝色(N端)到红色(C端)编码。锌离子显示为灰色球体,重要活性位点残基的侧链显示为绿色。(b) VIM-1活性位点,标记了关键活性位点氨基酸,水氧以红色球体表示。
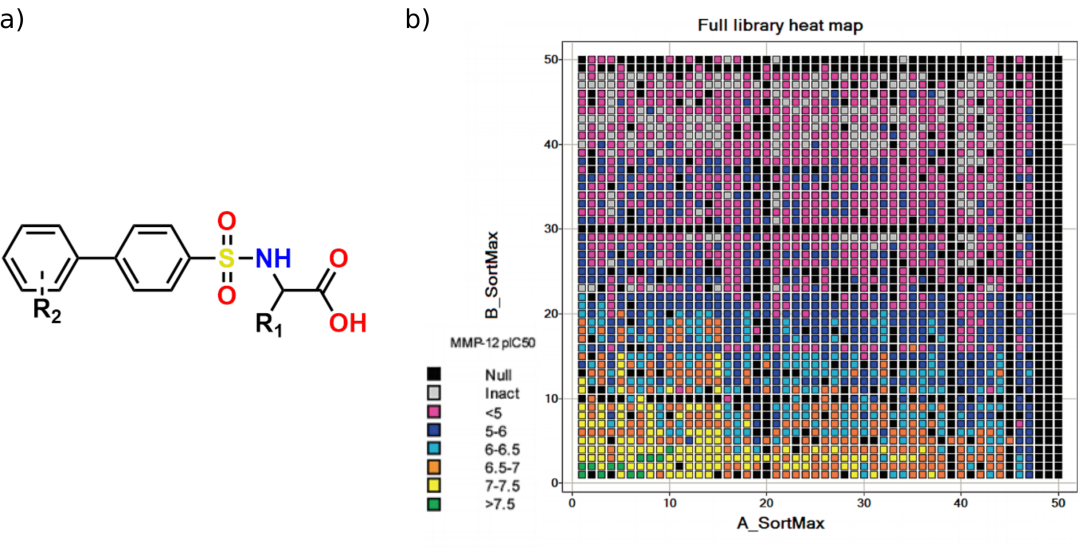
(a)在MMP-12数据集中的所有化合物中都存在联芳基磺酰胺核。(b) 50×50联芳基磺酰胺阵列热图。A SortMax对应R1的修改,B SortMax对应R2的修改。黑色的值被标记为“Null”,对应于化合物未合成或测定失败的化合物。灰色的值在分析中被发现是无活性的。
共同的吲哚-2-羧酸核心存在于MBL的每个分子数据集。在整个数据集中修改了取代基R1和R2,以探索SAR。
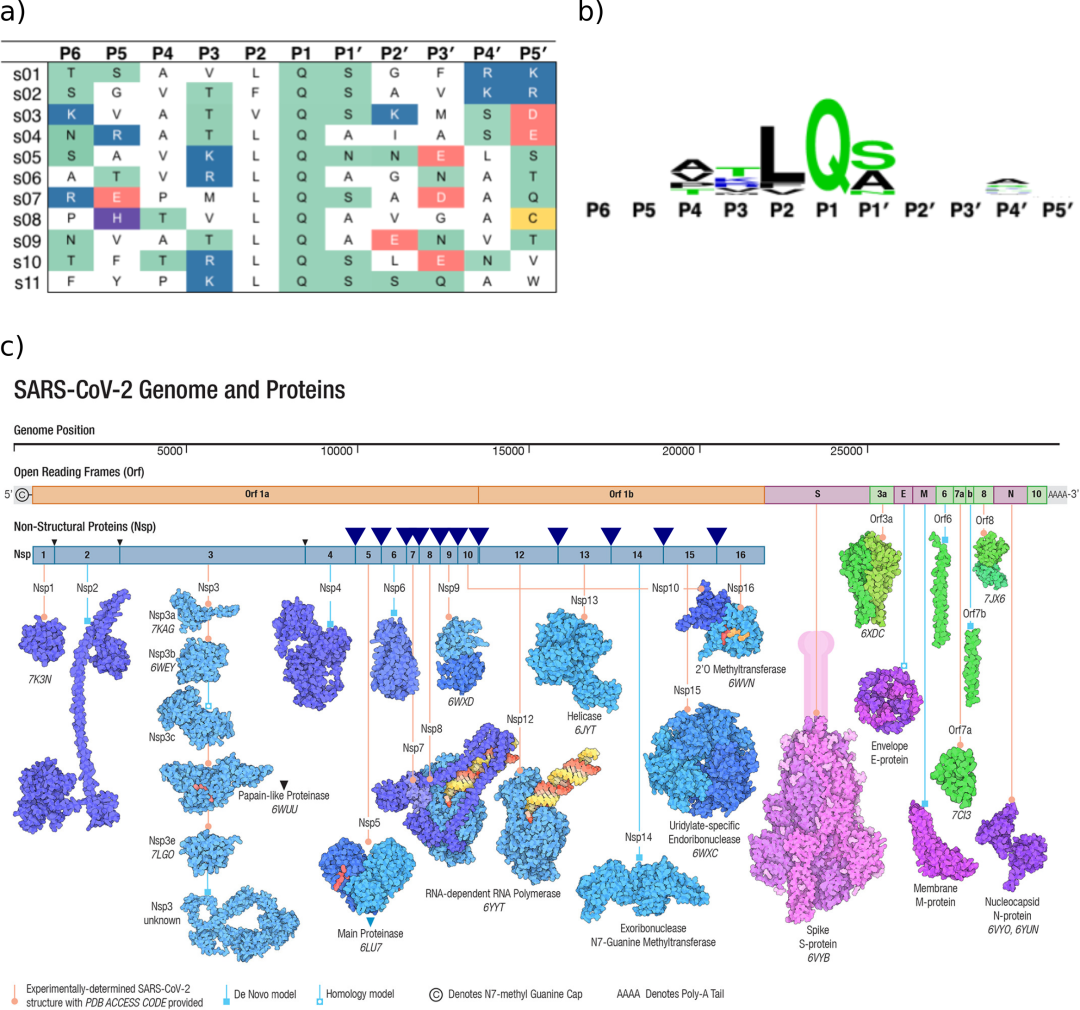
图a)和b)改编自该作品的原始出版物。a) 11个SARS-CoV-2 Mpro切割位点作为11个残基肽的序列(s01-s11)。带正电的氨基酸和带负电的氨基酸分别用蓝色和红色表示。极性氨基酸为绿色,半胱氨酸为黄色,组氨酸为紫色。b) WebLogo生成的每个位置残基相对丰度分析。谷氨酰胺在P1上是保守的,P2上的残基总是有一个疏水侧链。c)图经John Wiley和Sons(版权所有2021 John Wiley and Sons)许可转载(改编)。SARS-CoV-2基因组和蛋白质组综述病毒蛋白质组来源于非结构多蛋白pp1a和pp1ab(蓝色阴影),病毒粒子结构蛋白(粉色/紫色)和开放阅读框蛋白(Orfs,绿色阴影)。pp1a和pp1ab裂解位点用倒三角表示(黑色代表PLpro,蓝色/粗体代表Mpro)。底物s01-s11(如图a所示)是Mpro切割位点上按外观顺序分离Nsps的序列。
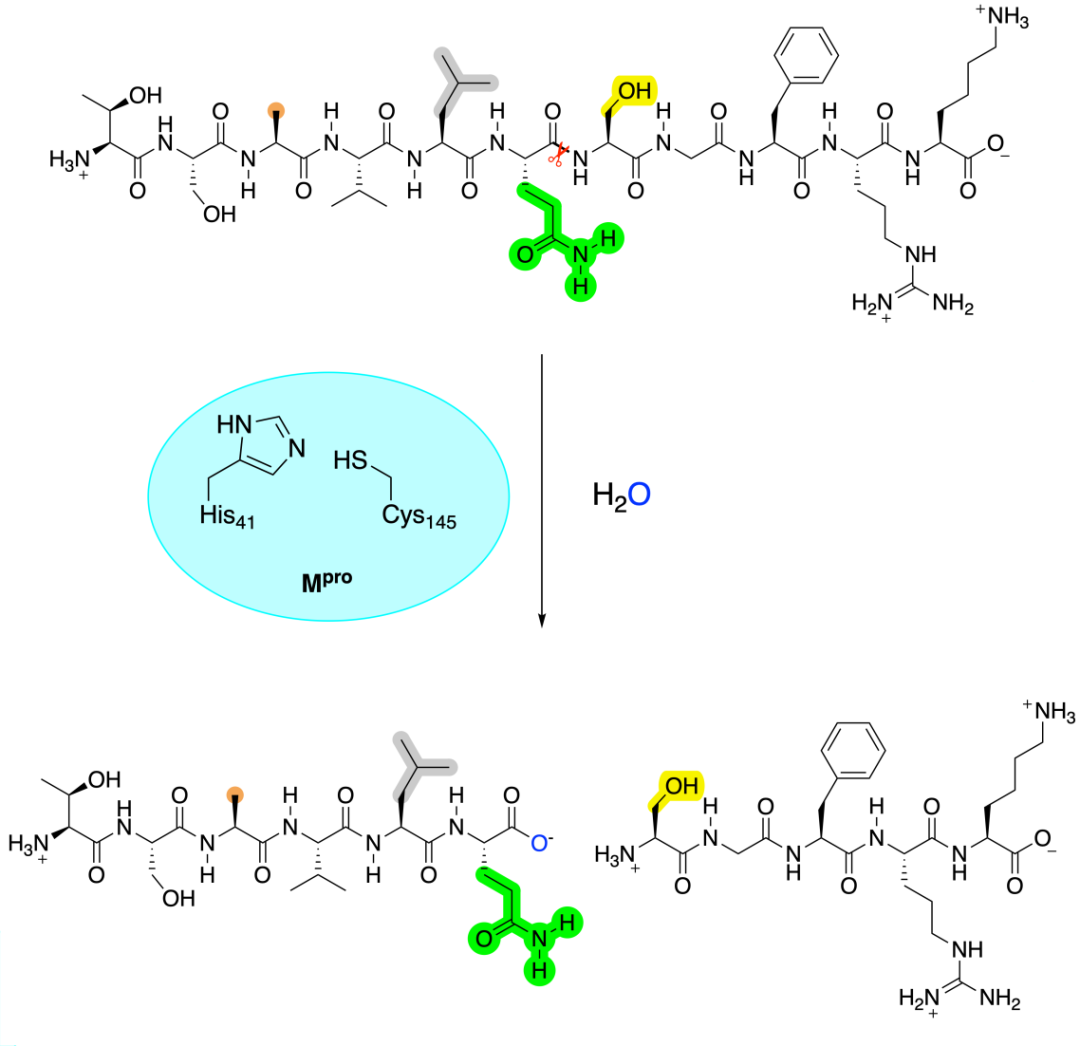
Mpro催化对天然底物s01的截短的11-mer序列催化底物裂解反应。图改编自该作品的原始出版物。切割位点用红色剪刀表示,以下是底物识别所必需的位置,颜色标记为:橙色(P4,“Small”),灰色(P2, Leu/Phe/Val/Met),绿色(P1, Glu),黄色(P1 ‘, Gly/Ala/Ser/Asn)。
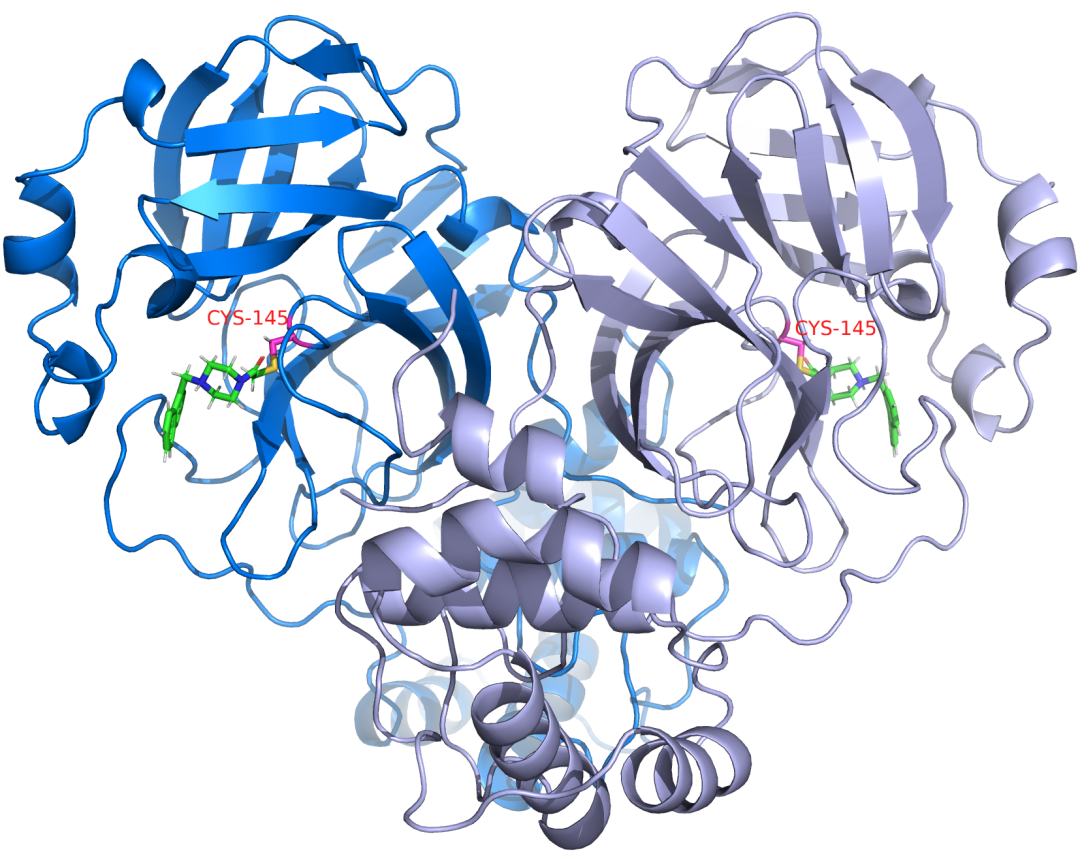
Mpro同型二聚体的晶体结构与片段x0830共晶。二聚体的两个亚基分别以蓝色和淡紫色表示,配体的碳、氮、氧和氢原子分别以绿色、蓝色、红色和白色表示。片段x0830共价结合活性位点半胱氨酸-145(洋红色)和硫(黄色)。该结构是从COVID Moonshot项目获得的,作为大规模晶体碎片筛选的一部分。该结构和COVID Moonshot项目创建的所有其他结构可以在上找到Fragalysis。
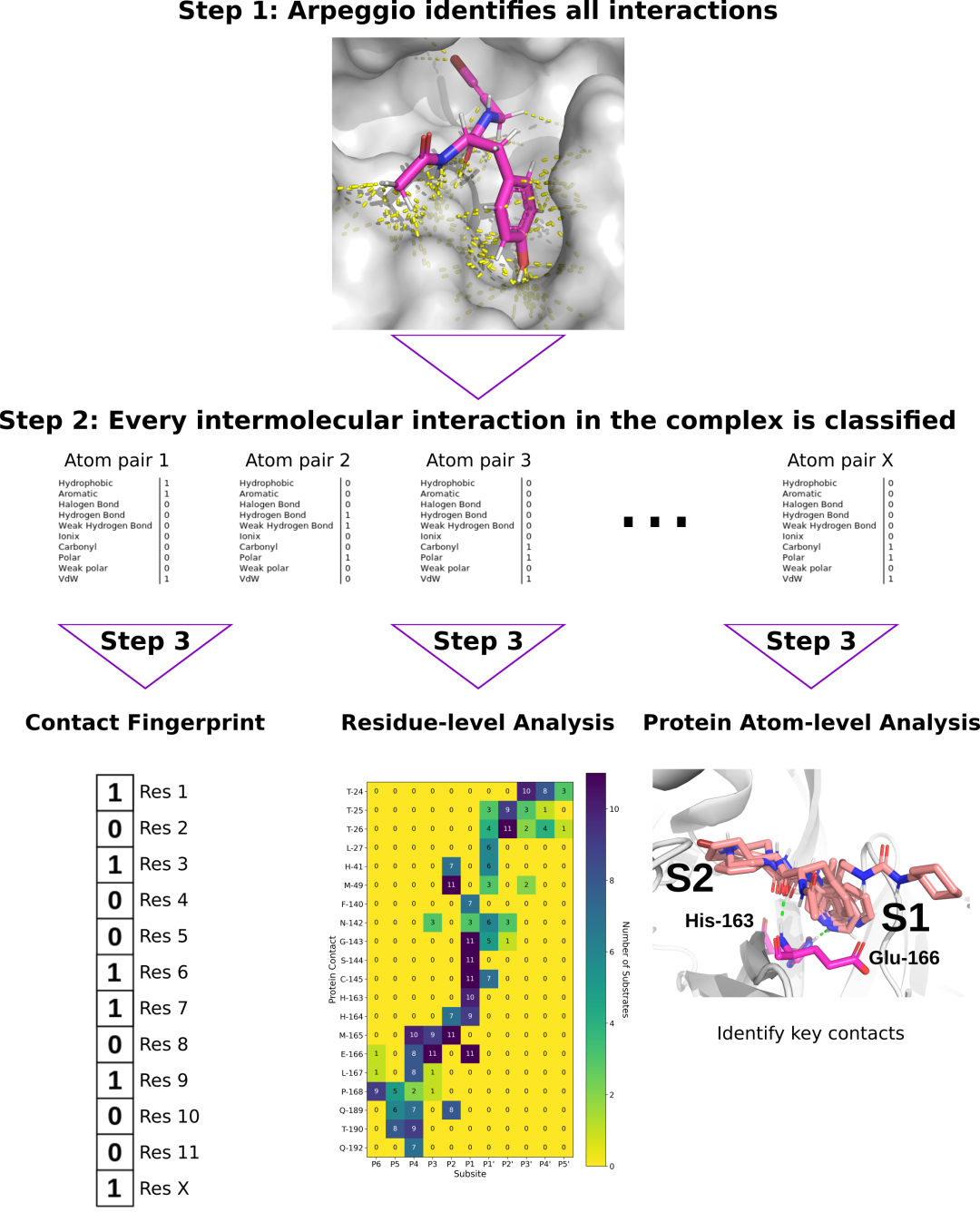
Mpro和配体或肽之间的琶音衍生分子间相互作用在三个细节水平上进行了分析。高水平的指纹被创建(左分支)基于存在的任何相互作用之间的配体和蛋白质残基。残馀水平相互作用分析(中间分支)获得活性位点子位点和在每个位点观察到的共同相互作用类型。原子水平分析(右分支)揭示了特定的相互作用,如氢键和疏水口袋,以指导抑制剂的设计。
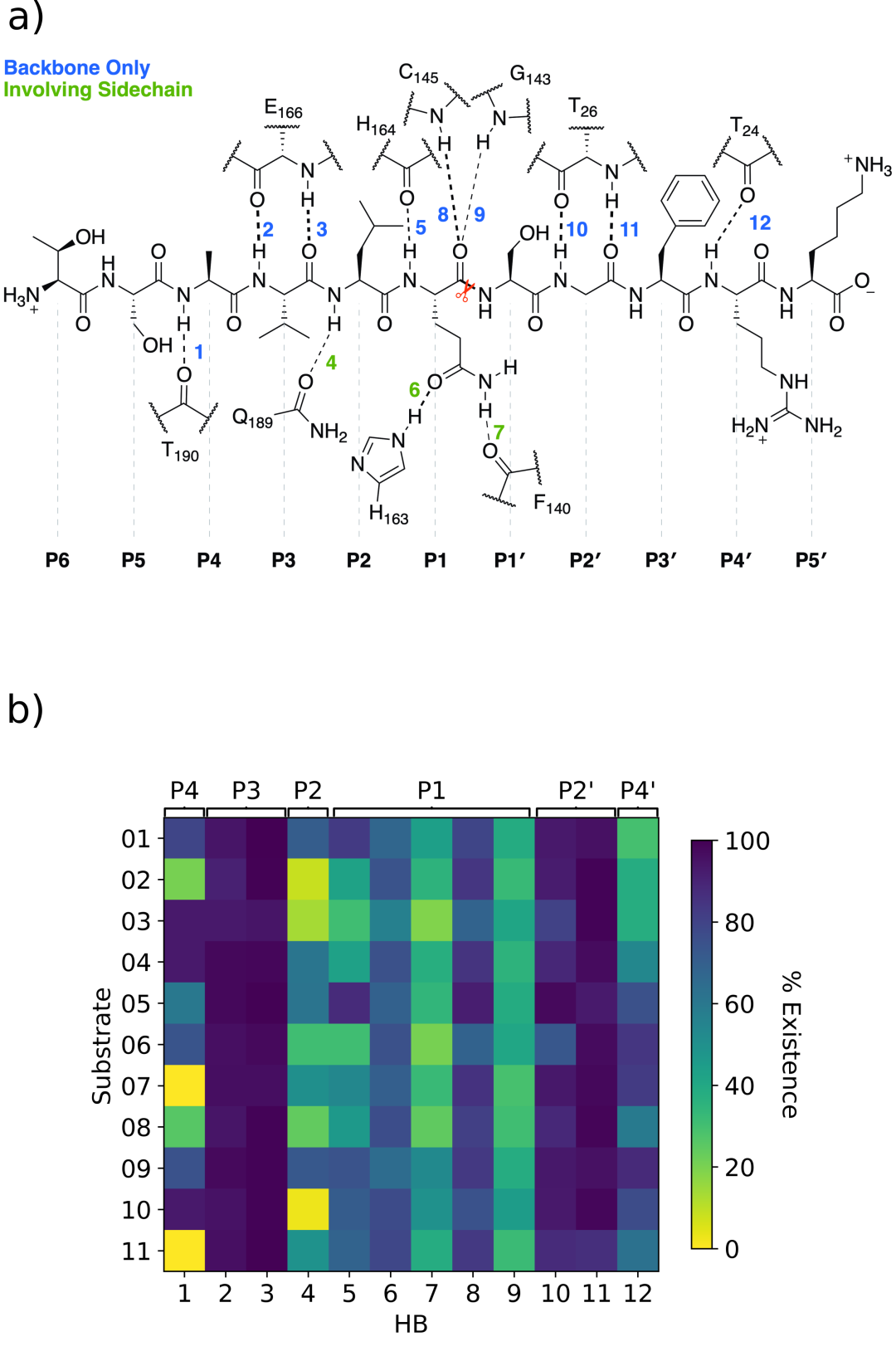
图片由H.T. Henry Chan创作,改编自该作品的原始出版物。a)已确定的12个主要氢键概述以底物s01为例。可剪酰胺用一把红色剪刀表示。b)每个底物的氢键1-12的观测率。从每个衬底的600 ns显式溶剂MD每纳秒拍摄快照。几乎100%的快照都观察到HBs 2-3和10-11,强烈地稳定了底物的姿态。HBs 6-9构成Gln识别基序。
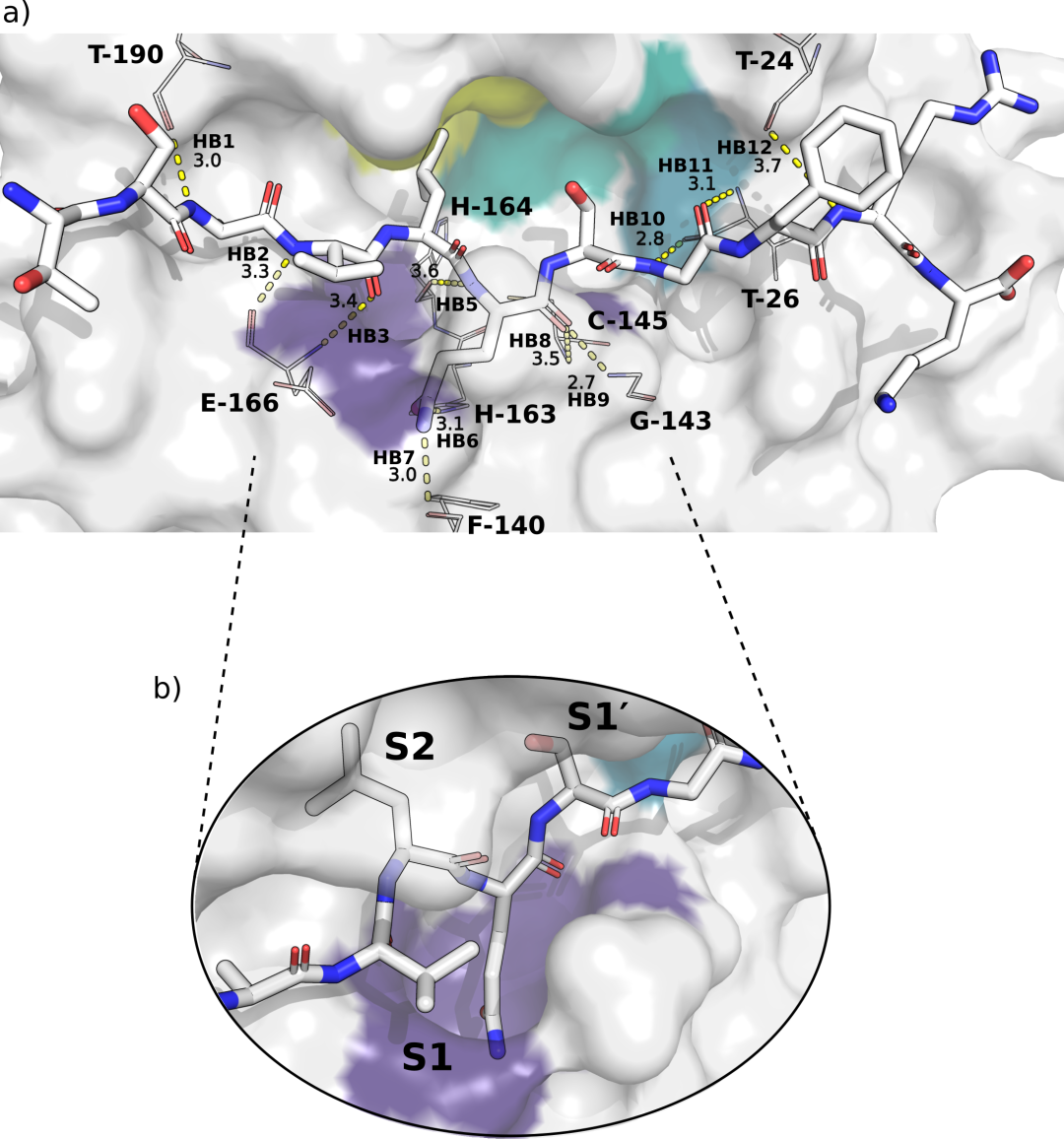
a)底物s01在Mpro活性位点的结合位综述。从MD中获得的用于相互作用分析的最具代表性的构象为,如第3.3.5节所述。参与结合的关键Mpro氨基酸标记为,参与s01识别的氢键1-4和6-12(见3.4.1.2节)用黄色虚线表示,并标记其距离。子站点S2-S2 ‘为,S2、S1、S1 ‘和S2 ‘分别用黄色、紫色、浅蓝色和绿松石色表示。b)可剪切酰胺键结合位点的旋转特写,显示S1处深埋的Gln残基。在P2上容纳s01 Leu残基的疏水S2口袋将Leu侧链深埋在口袋中,在那里它主要与Met-49和Met-165相互作用。
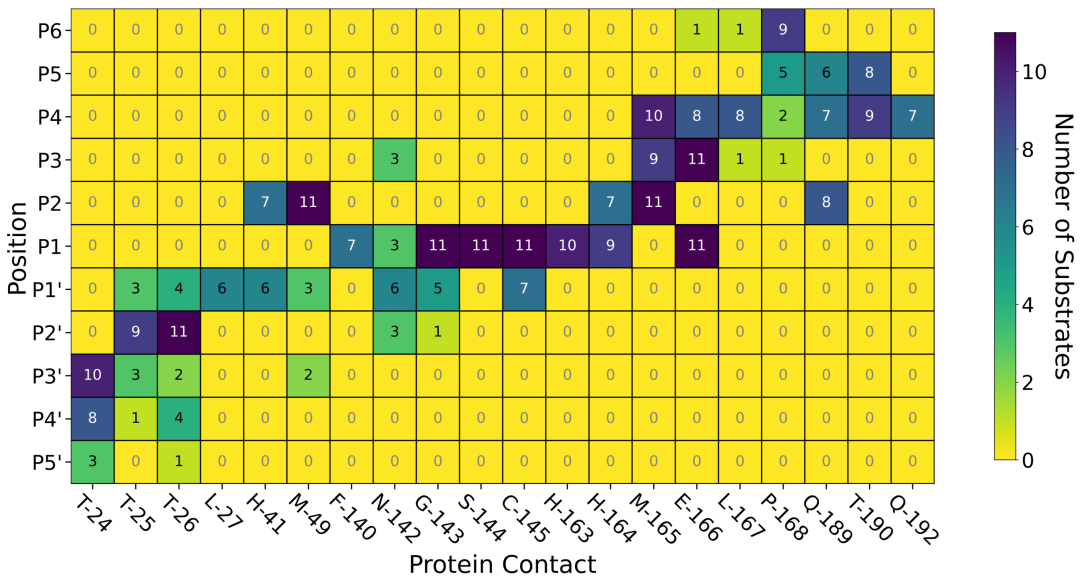
11种底物与Mpro之间的非共价相互作用图谱()来源于Arpeggio鉴定的相互作用[Jubb等人,2017]。每种底物最具代表性的位姿由3.3.5节中描述的显式溶剂MD获得。黄色表示没有底物在给定的子位点形成这种相互作用,而深蓝色表示相互作用由标记的大多数/所有底物形成。P1亚位点在所有11个底物之间具有高度保守的相互作用,主要与蛋白质中相同的氨基酸相互作用,表明P1处Gln残基的识别具有保守的,高度特异性的结合模式。
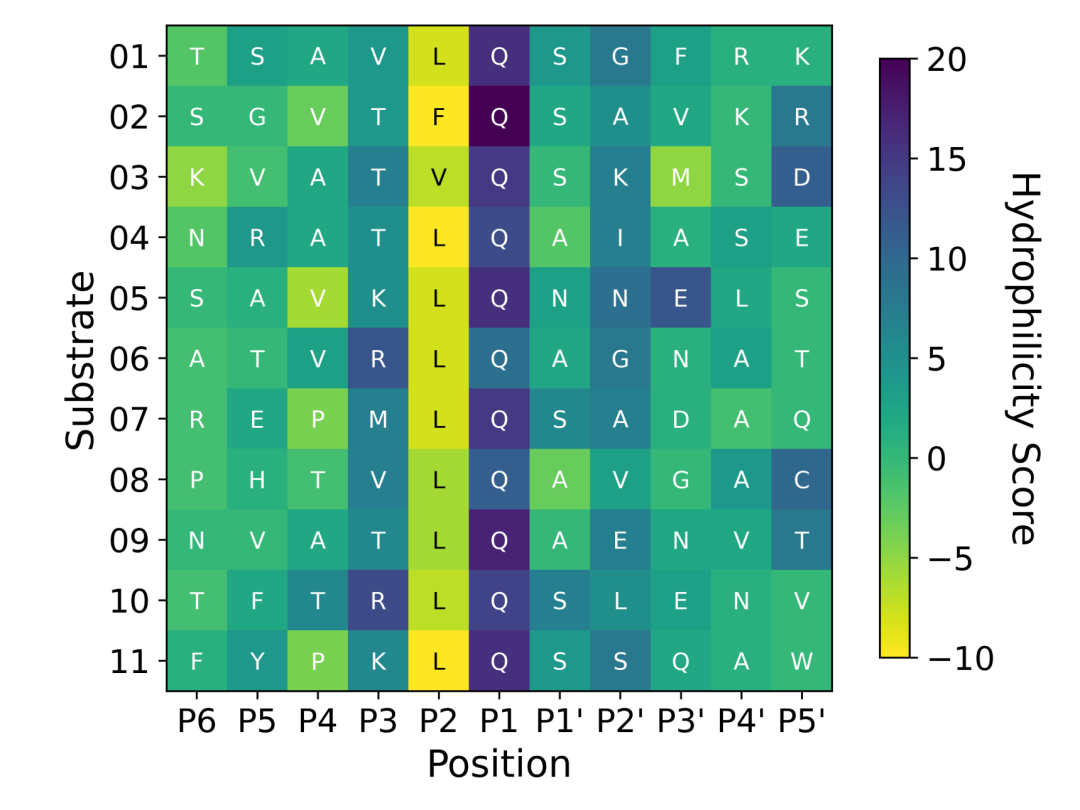
图显示了每个Mpro亚位点的亲疏水性。按照方法章节3.3中所述计算每种底物的亲水性评分。6 .使用琶音识别交互。得分越高表示口袋越亲水。子位P1具有高度亲水性,主要通过极性相互作用识别保守的Gln残基。观察到P2口袋为疏水口袋。其他亚位点没有表现出亲水或疏水相互作用的主要倾向。
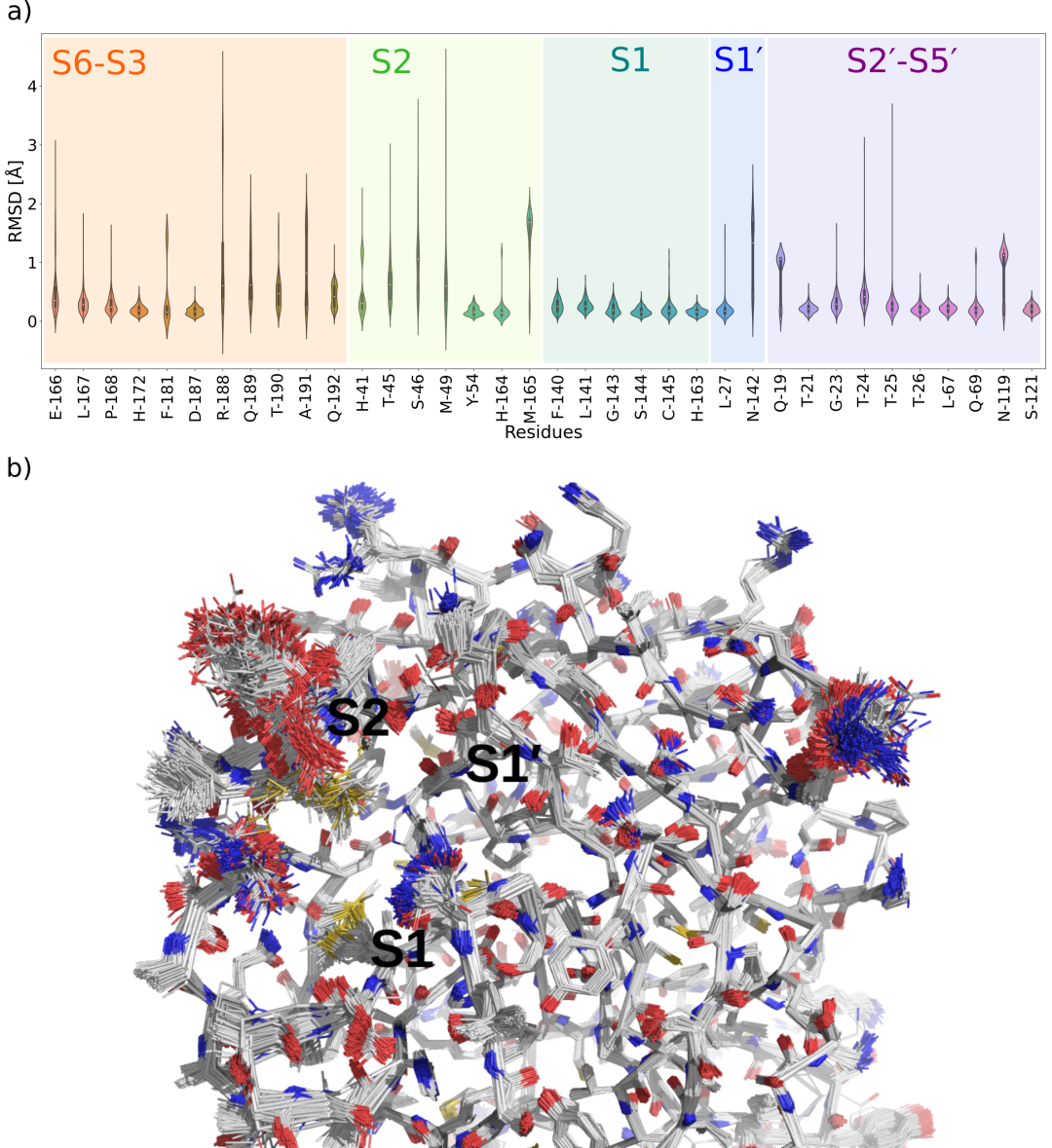
a) 333 Mpro共晶结构的活性位塑性分析。活性位点残基(19、21、23-27、41、45、46、49、54、67、69、119、121、140-145、163-168、172、181、187-192)是根据11个底物- Mpro模型()的MD分析选择的,它们对应于所有与底物相互作用的Mpro残基。小提琴图显示了333个m亲配体共晶结构53与参考非复杂结构(PDB 6yb7)之间每残基重原子RMSD值的分布。每个Mpro子站点都有颜色编码。b)所有333个Mpro共晶结构的线条叠加。标记了子位点S2, S1和S1 ‘。亚位S1′,尤其是S1具有较低的可塑性,在共晶结构之间几乎没有移动。然而,S2表现出大程度的灵活性,特别是在残基Ser-46和Met-49处,侧链的大运动会改变S2口袋的大小。
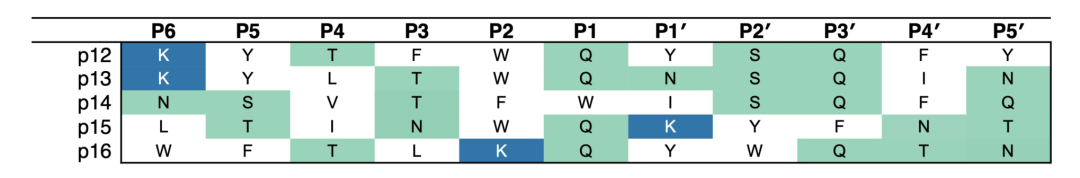
设计肽序列p12-p16。带正电的氨基酸是蓝色的。极性氨基酸呈绿色。设计的肽中没有带负电荷的氨基酸。引人注目的是,除了p15外,P2位置被占据,这是一个具有大芳香侧链的大块氨基酸(色氨酸或苯丙氨酸)。
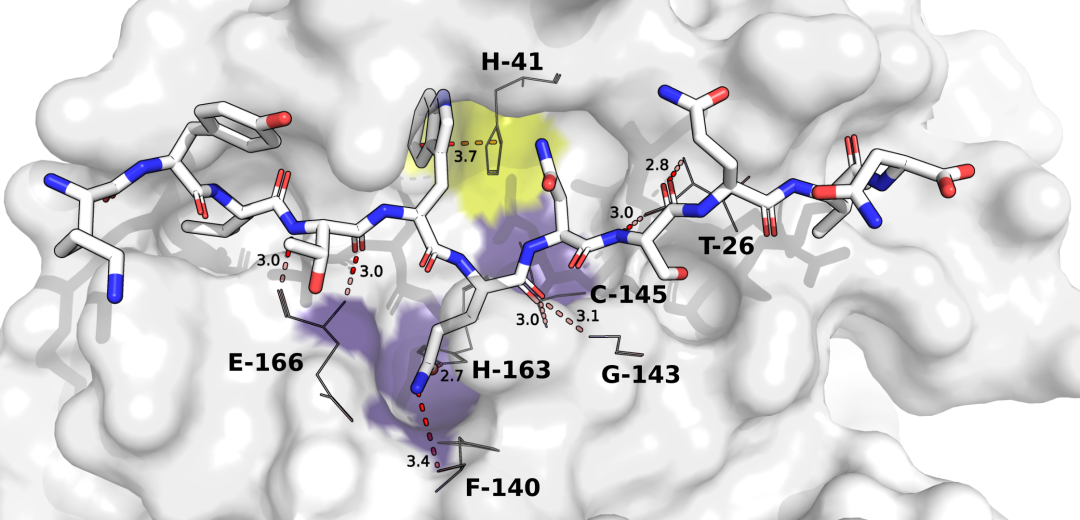
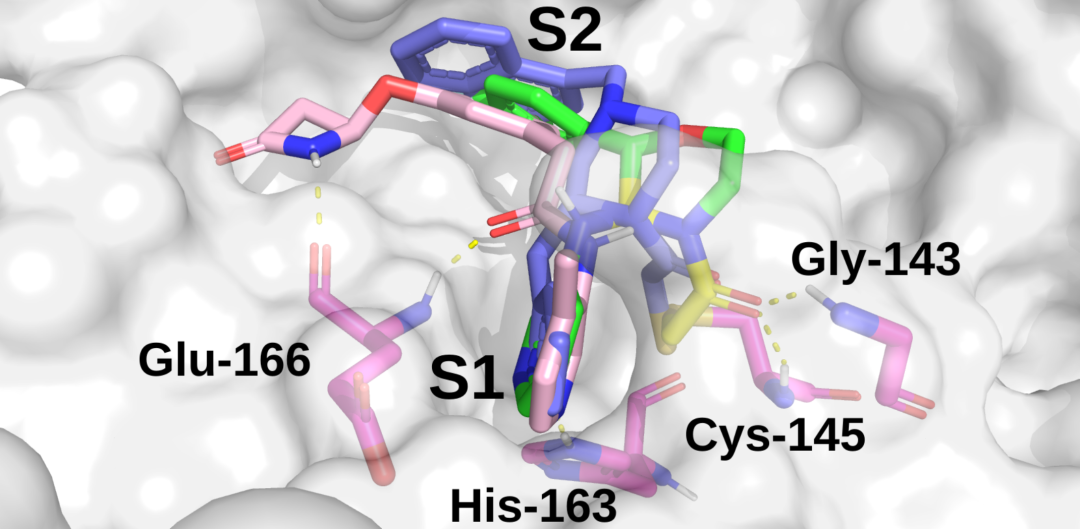
在Mpro的活性位点建模的肽抑制剂p13的结合模式概述。如图所示为在第3.3.5节中用于交互作用分析的MD中获得的最具代表性的位姿。参与结合的关键Mpro氨基酸被标记,关键氢键和与His-41的π-π堆叠被突出显示为红色虚线。子位点S2和S1分别用黄色和紫色表示。肽抑制剂p13占据了先前确定的所有关键氢键,用于紧密结合(Thr-26, Glu-166)以及谷氨酰胺识别(氧阴离子孔Cys-145, Gly-143和HBs与Phe-140和His-163)。此外,P2上的Trp残基与His-41形成π-π堆积相互作用,阻断催化二元体。
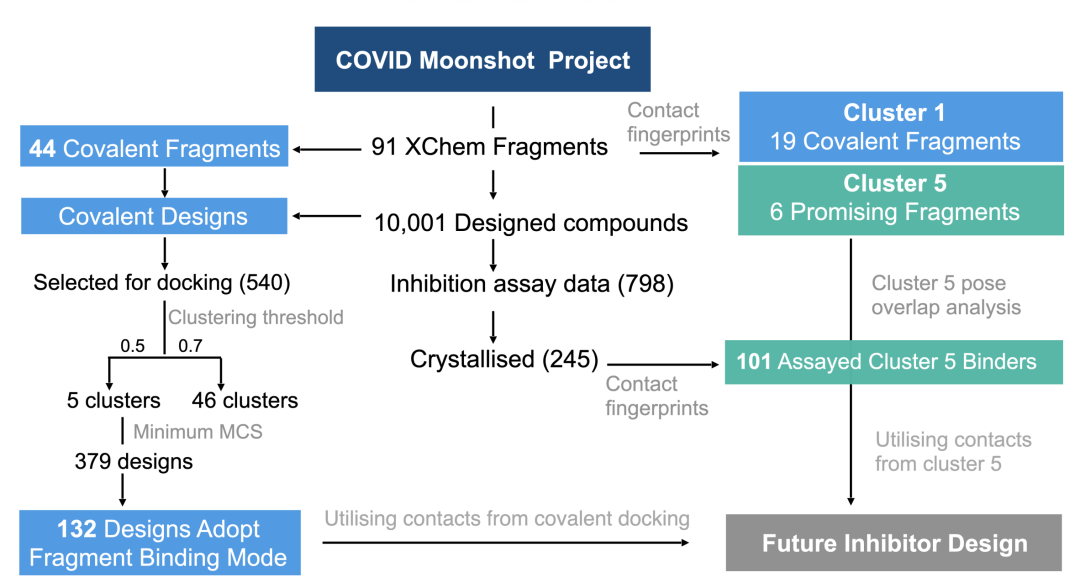
Moonshot项目和XChem片段筛选中片段和设计化合物的分析改编自该作品的原始出版物。工作流使用来识别有希望的片段并指导新颖的设计。使用COVID Moonshot Project数据库选择分析时存在的44个共价片段共晶结构和所有共价化合物设计(10,001个化合物)。所有基于特定共价片段的共价设计都被选中用于AGCD,并对得到的对接位进行过滤,以选择132个采用原始片段结合模式的高质量位姿的化合物。然后在已知的背景下分析这些设计,从11个底物模型和91个XChem片段结构中获得关键的相互作用。所有91个XChem片段使用Arpeggio分析,并创建交互指纹以结合模式聚类片段。最重要的簇(簇5)被发现占据了负责底物识别的关键相互作用,并被用作模板,以识别COVID Moonshot项目数据库中已知的簇5结合物,并指导片段的阐述。
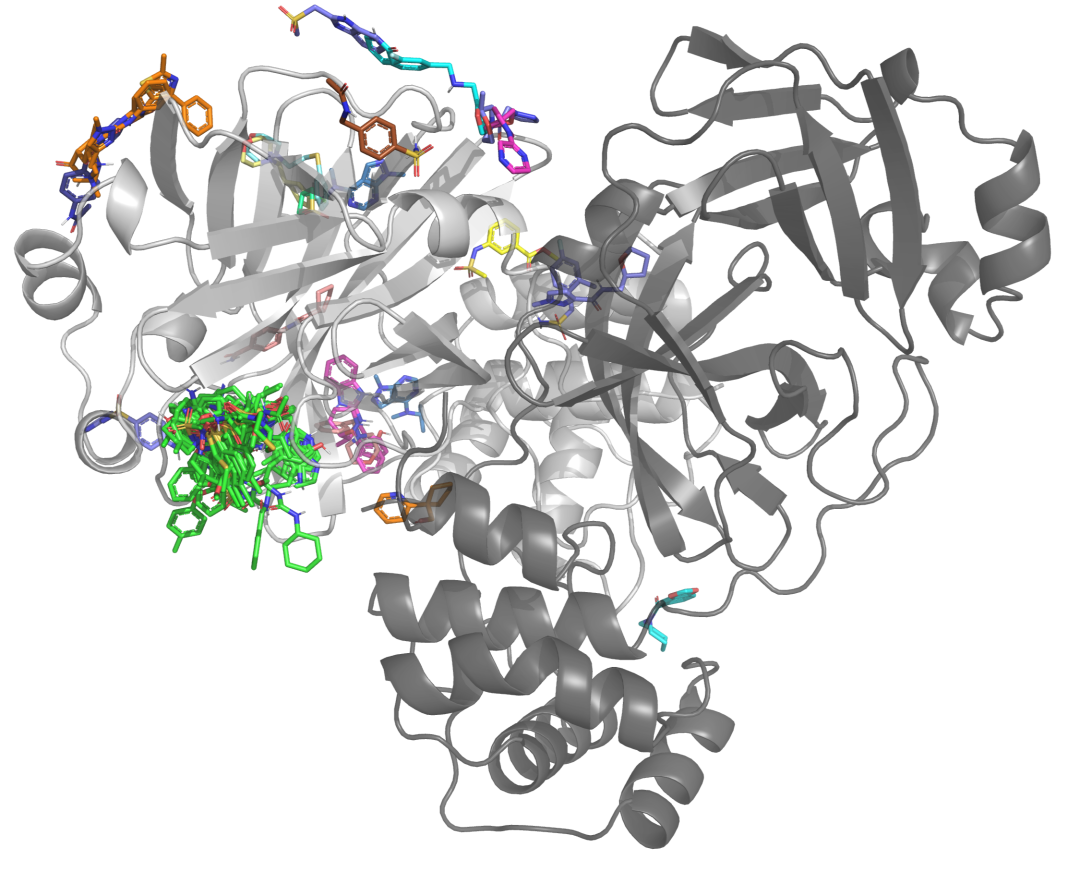
与Mpro二聚体结合的所有91个XChem片段的覆盖。图中显示了A链上所有的结合位点(白色)。作为具有代表性的结构,采用了x0830共晶结构的Mpro晶体结构。有66个片段结合到活性位点(绿色片段),25个片段结合在远程口袋中(片段1101结合在两个不同的远程位点)。非活性位点结合片段被广泛地着色以区分不同的结合位点。
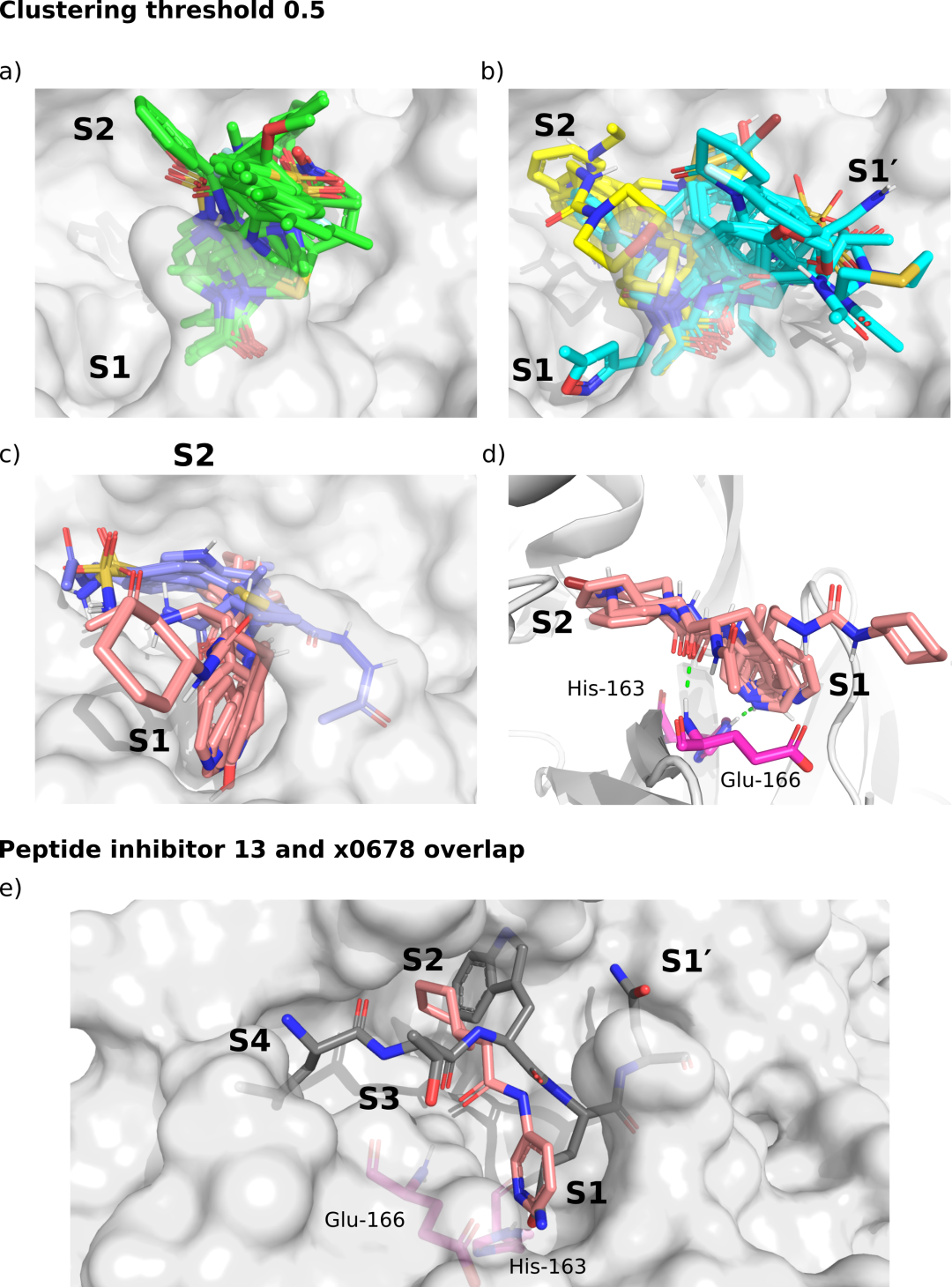
x0830结合的Mpro结构的分子表面(白色表面)和的前5个最密集的片段簇使用聚类阈值为0.5。a)聚类1个片段(绿色);B)簇2(青色)和3(黄色);C) 4号簇(淡紫色)和5号簇(粉色);d) 5号簇的特写。绿色虚线表示片段羰基氧与glue -166的主氮之间的两个关键HBs (HB 3,图3.6),以及片段His-163 n柱与杂环氮之间的两个关键HBs (HB 6,图3.6)。e)将p13的P4-P1′截断结构(灰色)和簇5粘结剂x0678(粉色)与x0678共晶Mpro结构(白色表面)叠加。簇5抑制剂非常适合模拟Gln在S1的识别,并延伸到疏水的S2口袋。
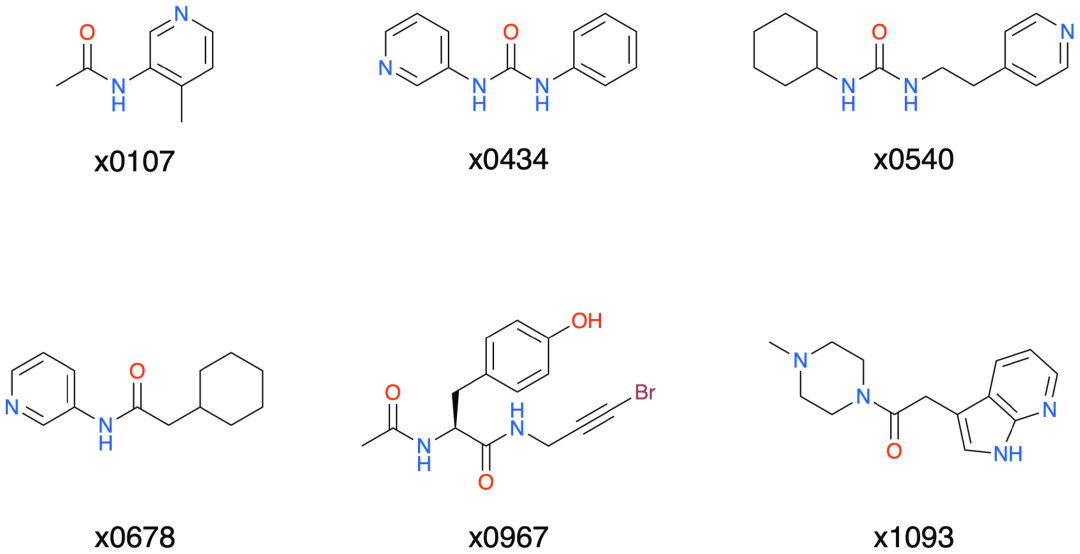
簇5个XChem片段的化学结构。请注意,普遍存在含氮杂环,或者在x0967的情况下,含有酪氨酸的酚衍生物结构,负责S1口袋中的氢键。
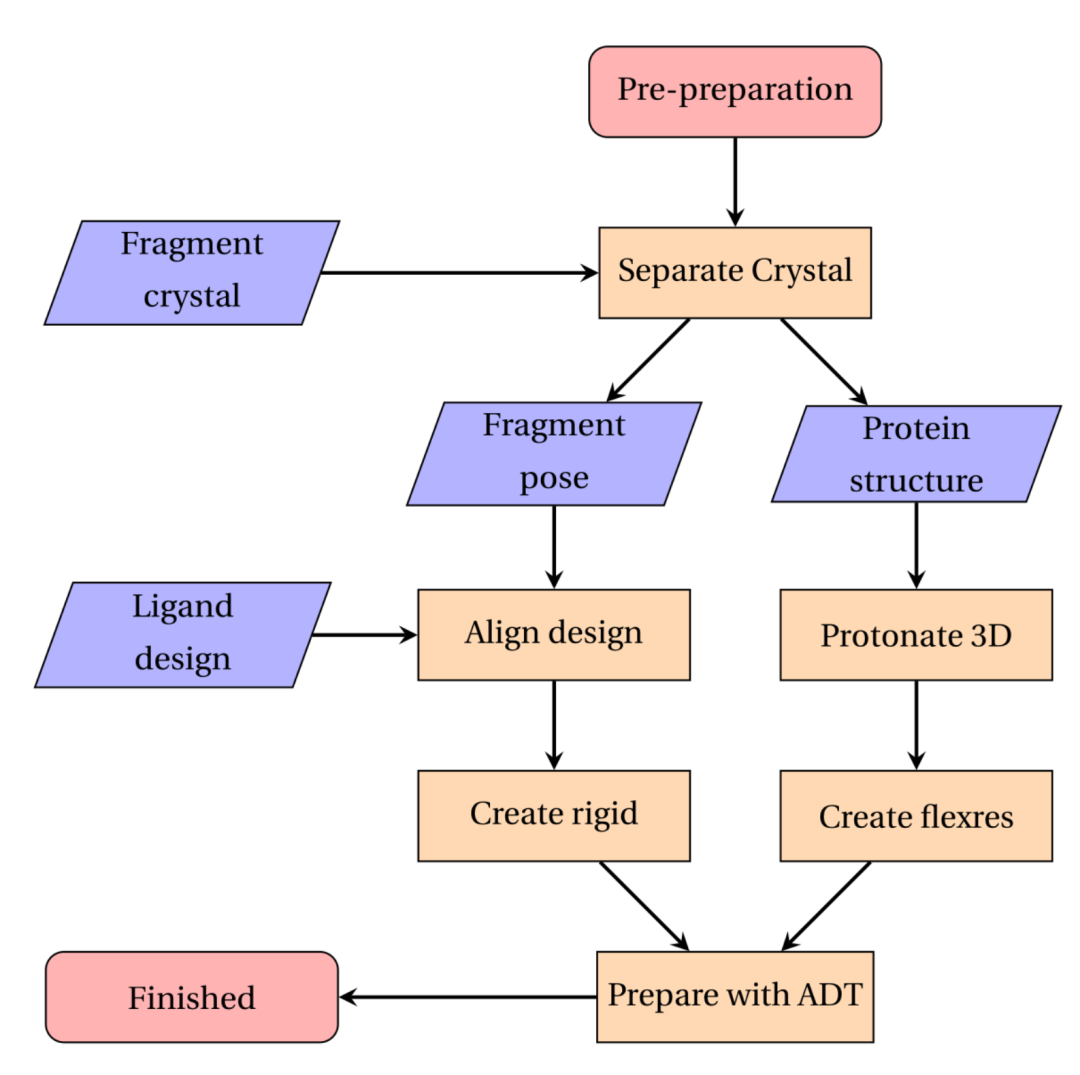
显示主动引导共价对接过程中使用的步骤和数据的流程图。
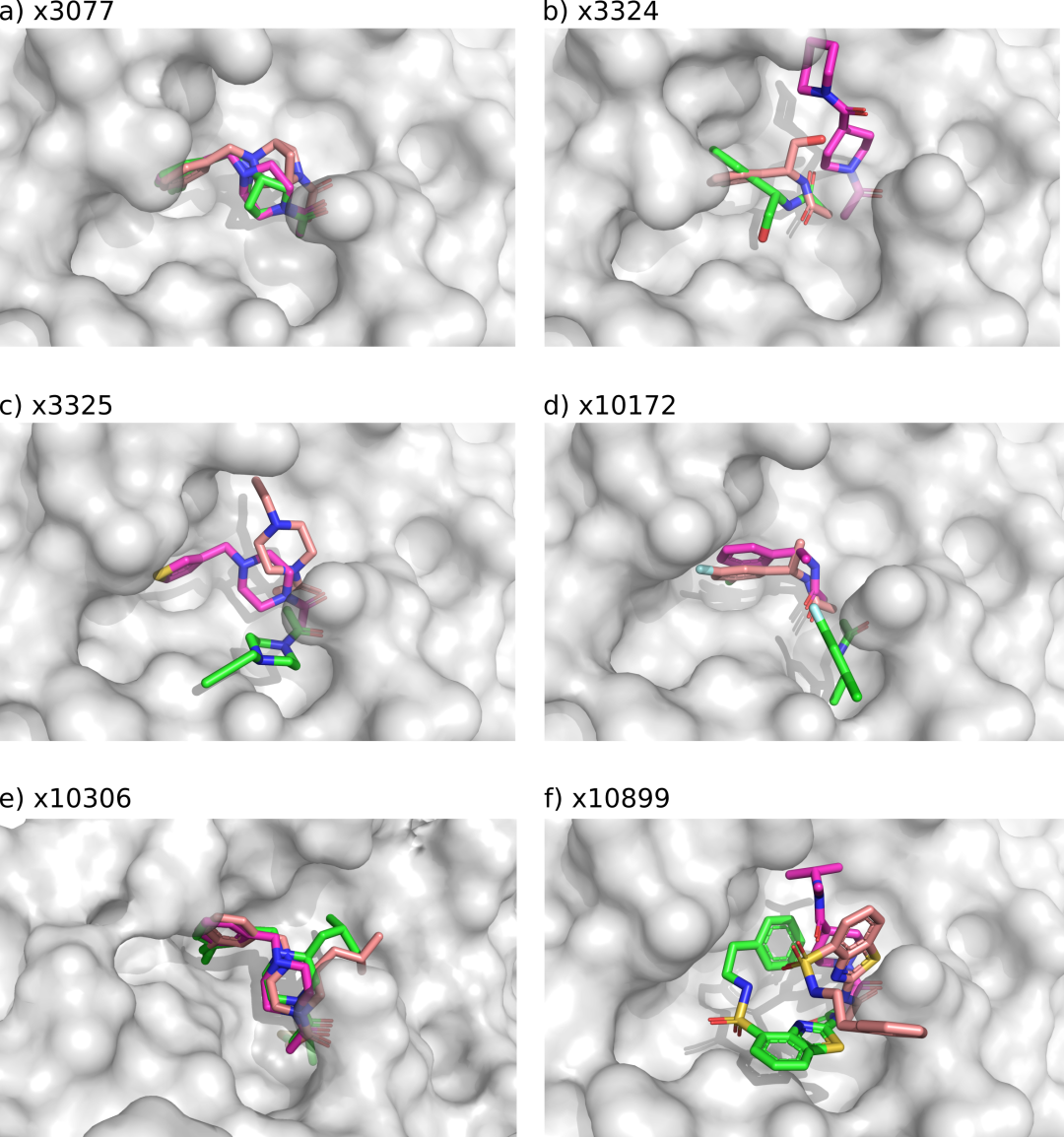
设计(绿色)的AD4共价对接过程中最高簇中最低能量位态与原始灵感片段(粉色)的晶体结构和设计(鲑鱼)的晶体结构的叠加。对于每次对接,都使用对应的启发片段共晶结构的Mpro蛋白结构。a)登月设计X3077(鲑鱼)与灵感片段X0770(粉色)和对接姿态X3077(绿色)。b) Moonshot设计的化合物X3324(三文鱼)与灵感片段X1380(粉色)和X3324对接姿态(绿色)。c)登月设计X3325(鲑鱼)与灵感片段X1386(粉红色)和的对接姿态X3325(绿色)。d)登月设计X10172(鲑鱼)与灵感片段X1382(粉红色)和对接姿态的X10172(绿色)。e)登月设计X10306(鲑鱼)与灵感片段X0770(粉色)和对接姿态X10306(绿色)。f)登月计划设计X10899(鲑鱼)与灵感片段X1458(粉色)和对接的姿态X10899(绿色)。姿势的设计x3077和x3324几乎完全类似原来的水晶姿势。
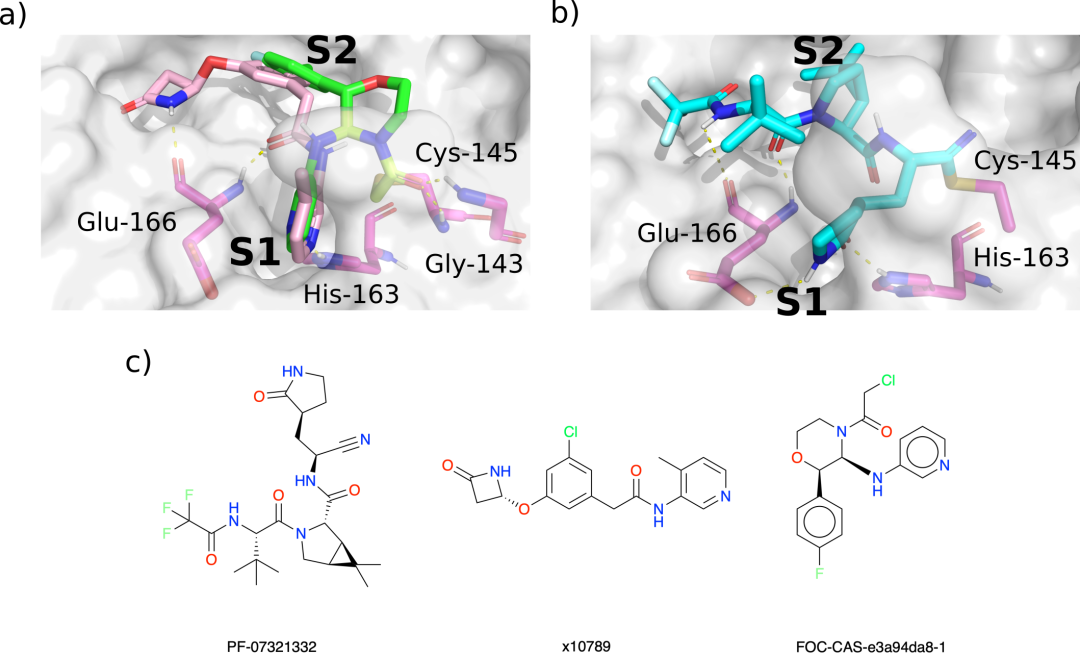
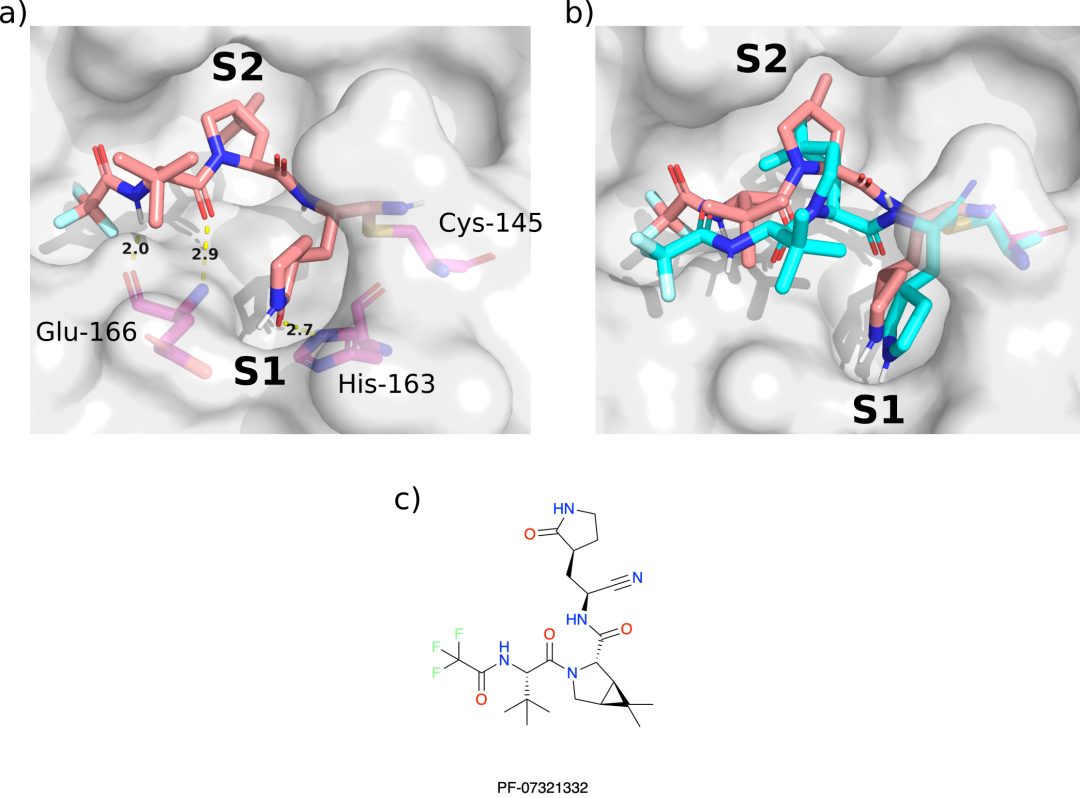
对接为新型抑制剂设计提供了信息。Mpro残基(洋红色)与配体之间的HBs用黄色虚线表示。a) focc – cas -e3a94da8-1的对接位态(绿色和黄绿色)与晶体结构x10789(粉色)在Mpro表面(PDB入口5RER)的叠加;1.88˚A分辨率)。衍生化x10789到氧阴离子孔可以通过连接一个亚甲酰胺基团存在于x0830(突出显示的黄绿色)。b)辉瑞FDA批准的药物化合物PF-07321332 (Nirmatrelvir)的对接姿势,共价对接到Mpro (6XHM);1.41˚A分辨率)。Nirmatrelvir (cyan)共价附着在Cys-145上。对接的Nirmatrelvir采用与x10789和x0830“组合”相同的主要相互作用,即与Glu-166主干的双HB,与S1亚位点的hs -163的HB,以及S2亚位点的一系列疏水相互作用。c) Moonshot设计的化合物FOC-CAS-e3a94da8-1、晶体学片段x10789和抑制剂Nirmatrelvir的结构。总的来说,Moonshot化合物系列的氧化阴离子孔扩展将使结合与批准的药物Nirmatrelvir完全相同的关键相互作用。
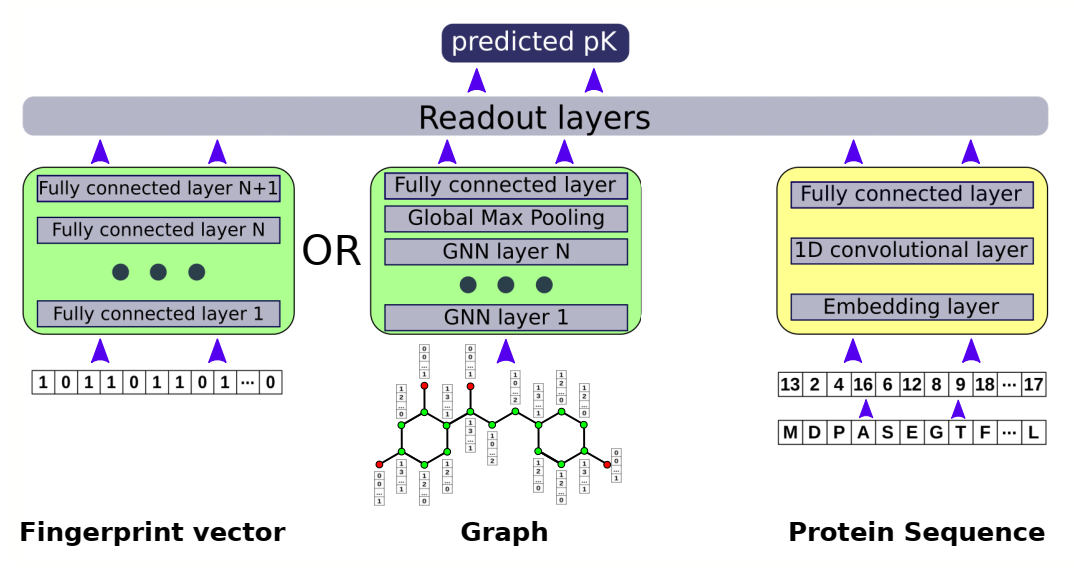
配体分支(绿色)和序列分支(黄色)的模型总体结构。配体要么作为指纹向量嵌入MLPNet分支(最左边的分支),要么为GNN分支(中间的分支)创建分子图。在右侧分支上,显示了蛋白质嵌入,其中1卷积层被嵌入的蛋白质氨基酸序列喂养。来自蛋白质和配体分支的输出被连接并馈送到三个完全连接的读出层中,并进行单个pK预测。
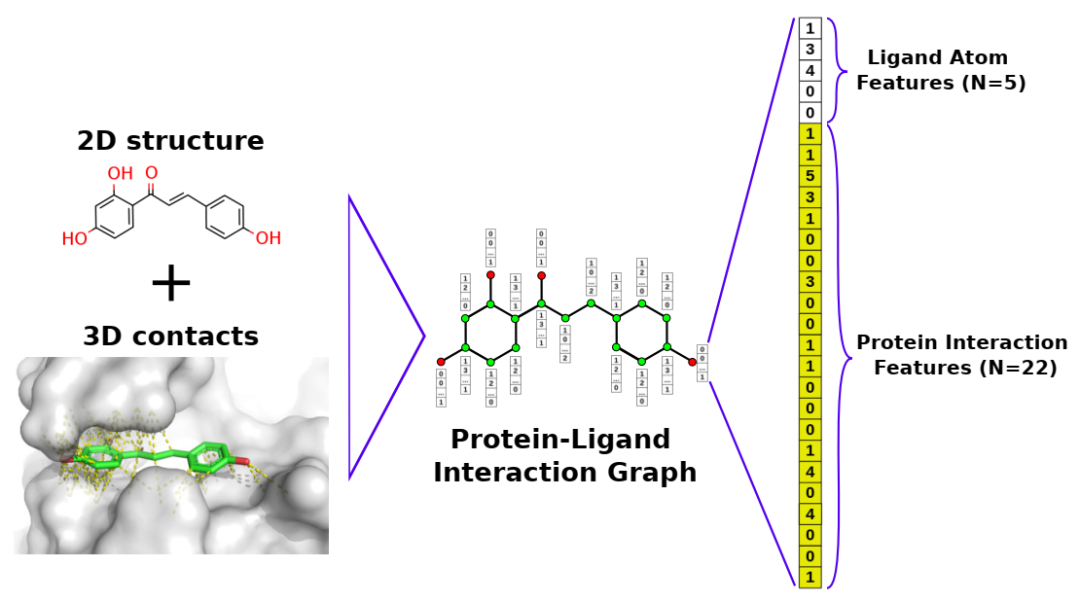
蛋白质-配体相互作用的形式化表示图。左图为6ajv配体的化学结构和结合位点的三维结构,每个配体原子在周围4˚A半径内的分子间相互作用用黄色标记。PLIGs是通过为每个节点分配一个整数特征向量来创建的,该特征向量由rdkit派生的原子描述符组成(相邻重原子的数量;邻氢数;明确的价;芳香性;和环成员)以及与每种蛋白质原子类型(22种唯一原子类型各一个)的相互作用的数量,这些相互作用在预定义的距离阈值内,从而为PLIG中的每个原子节点创建最终的27维特征向量。
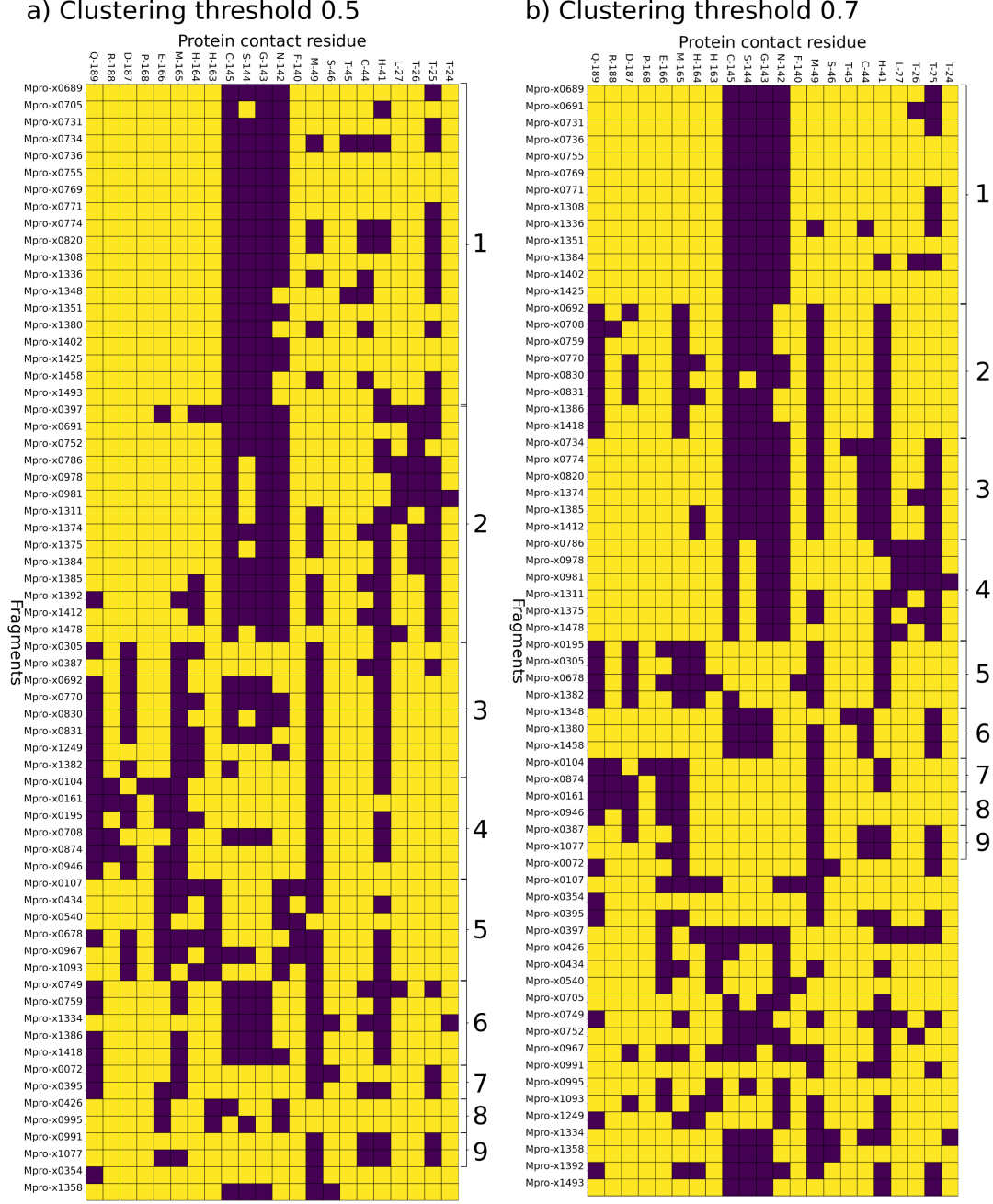
图改编自本作品的原始出版物。66个活性位点XChem片段的接触矩阵,按照a)较宽的聚类阈值0.5和b)较紧的聚类阈值0.7进行排序。触点显示为紫色,无触点显示为黄色。含有1个以上分子的簇用括号标记(x0354和x1358除外)。
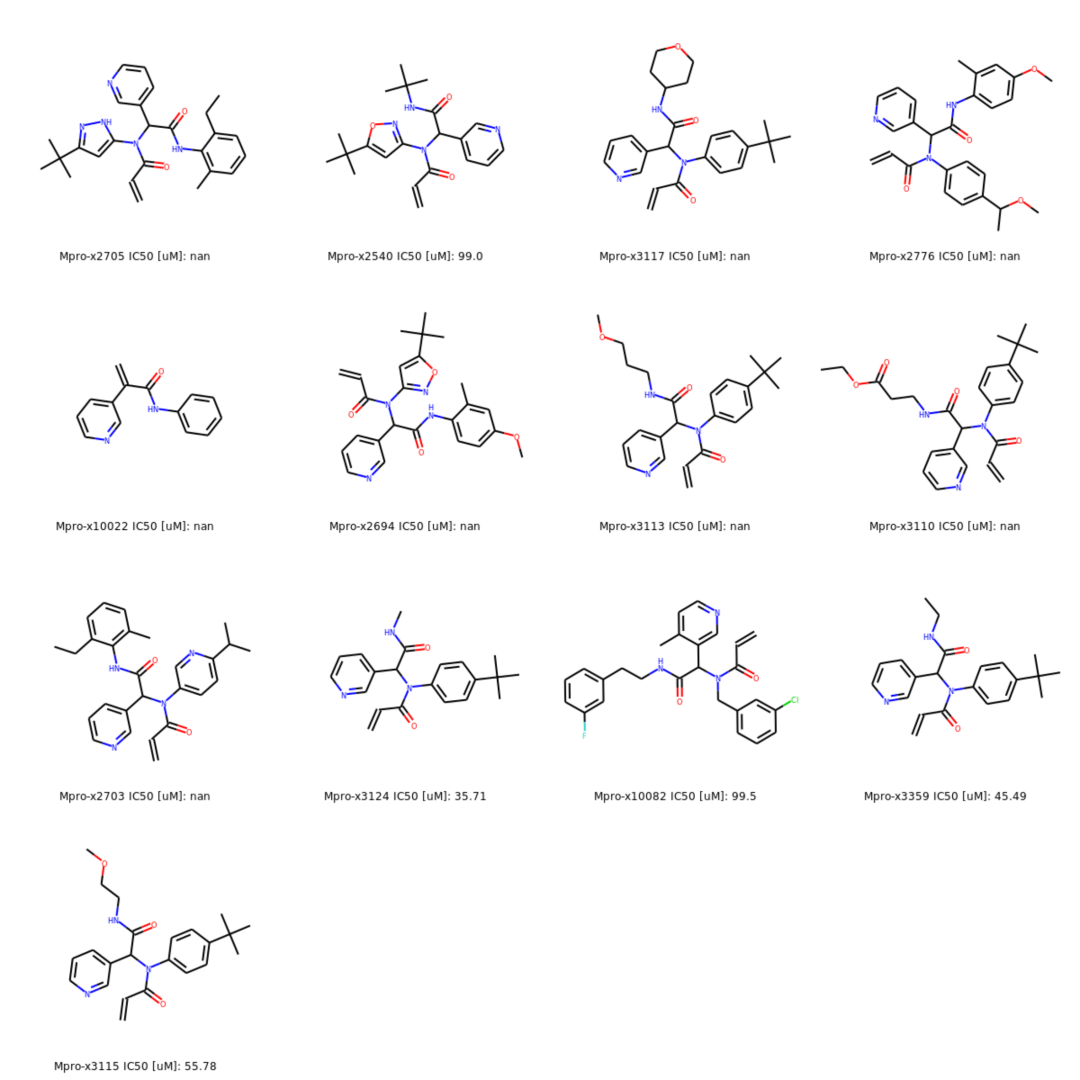
图改编自本作品的原始出版物。在Mpro氧阴离子空穴中结合的所有簇5化合物的选择。所有化合物都是共价抑制剂,通过丙烯酰胺弹头与Cys-145反应。IC50值从海报中获取。注:“nan”表示化合物尚未被检测。
图改编自本作品的原始版本,采用CCBY 3.0许可协议。focc – cas -e3a94da8-1(绿色)与MIH-UNI-e573136b-3(蓝色)的对接位叠加,以及晶体学观察到的x10789(三文鱼)与Mpro (PDB: 5RER)的结合模式。化合物focc – cas -e3a94da8-1上的黄色表示x10789扩展到氧阴离子空穴。
a) Nirmatrelvir(三文鱼)的晶体形态,共价附着于Mpro (7RFS;1。91˚A分辨率)。关键的Mpro残基用紫色表示,关键的氢键用黄色虚线表示,它们的距离用˚a表示。Nirmatrelvir的晶体结构与之前在簇5)和天然底物模型中发现的主要相互作用相同,即与glu166主链的双HB,与His-163的S1亚位的HB,以及S2亚位的一系列疏水相互作用。b) Nirmatrelvir的对接(青色)和晶体(鲑鱼色)姿态在Mpro (7RFS)表面的叠加。c) Nirmatrelvir的二维结构。








微信群 公众号





 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


