通过计算方法预测蛋白质-蛋白质相互作用(PPI)靶标和调节剂之间的相互作用可以加速PPI药物的筛选和设计。因此,上海交通大学魏冬青教授和熊毅副研究员团队开发了MultiPPIMI框架。该文章于2023年12月1日发表于《Journal of Chemical Information Modelling》。该框架基于生物和化学序列信息预测PPI和调节剂之间的相互作用,它可以推广到训练数据外的PPI靶标和小分子调节剂。

PPIs是各种生物过程和疾病的重要组成部分。小分子调节剂是一种能够调节PPI的化合物,它们被视为新型的抗癌、抗病毒和抗微生物的药物候选物。然而,大多数现有的筛选PPI调节剂的计算方法都需要靶标三维结构或参考调节剂作为输入,这限制了它们对新型PPI靶标的适用性。为了解决这个挑战,作者提出了一个基于序列的深度学习框架MultiPPIMI,它可以预测任意给定的PPI靶标和调节剂之间的相互作用。MultiPPIMI整合了PPI靶标和调节剂的多模态表示,并使用双线性注意力网络来捕捉分子间的相互作用。在benchmark数据集上的实验结果显示,MultiPPIMI在三种冷启动场景下达到了平均0.837的AUROC,在随机划分场景下达到了0.994的AUROC。
数据来源
作者通过DLiP数据库构建了一个benchmark数据集,该数据库包含了从公共数据库和文献中整理出来的PPI和与之对应的小分子调节剂;PPI的蛋白质序列是从UniProt数据库中获取的。经过严格的数据过滤后,9,817个调节剂和120个PPI靶标被保留下来。为了构建一个平衡的PPI-调节剂相互作用数据集,作者定义正样本为PPI靶标和与之对应的活性调节剂。为了生成负样本,对于每一个PPI家族,作者假设所有与其他PPI家族的靶标相互作用的调节剂都是非活性的。负样本随后被定义为PPI靶标和与之对应的假定的非活性调节剂。为了解决类别不平衡的问题,作者对负样本进行了采样,得到了一个由11,630个正样本和11,630个负样本组成的数据集。
模型设计
MultiPPIMI由以下四个模块组成(图1):首先通过SMILES序列计算小分子表示,该表示由GraphMVP编码器提取的structural embedding和物理化学性质向量构成;另外根据氨基酸序列计算PPI 表示,其包含由ESM2提取的structural embedding和物理化学性质向量;接下来,双线性注意力模块根据PPI和调节剂的embedding学习分子间的相互作用规则;最后,一个全连接层将注意力矩阵映射到一个连续的概率值,用来表示PPI-调节剂相互作用的可能性。
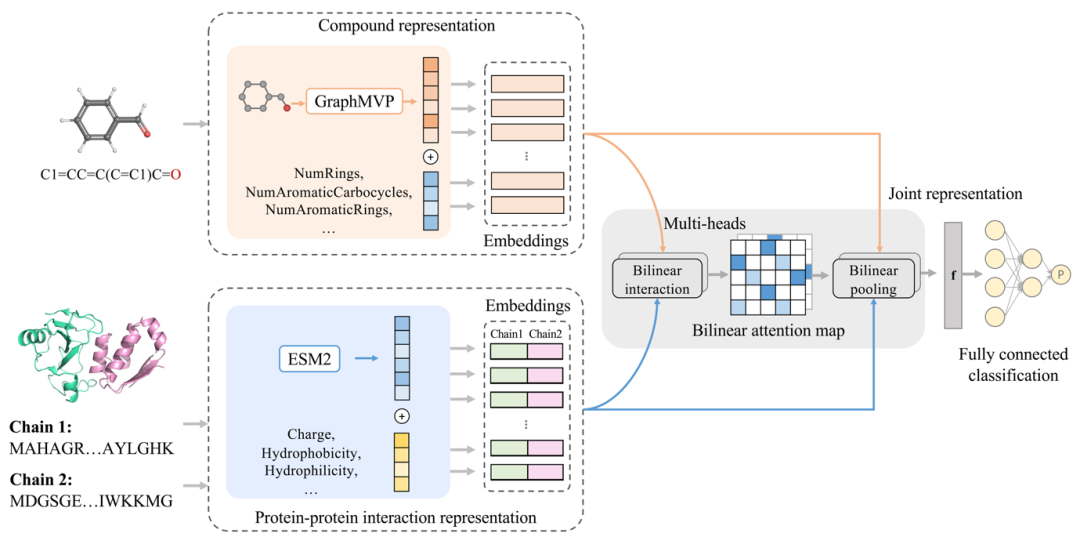
图1. MultiPPIMI模型图
实验结果
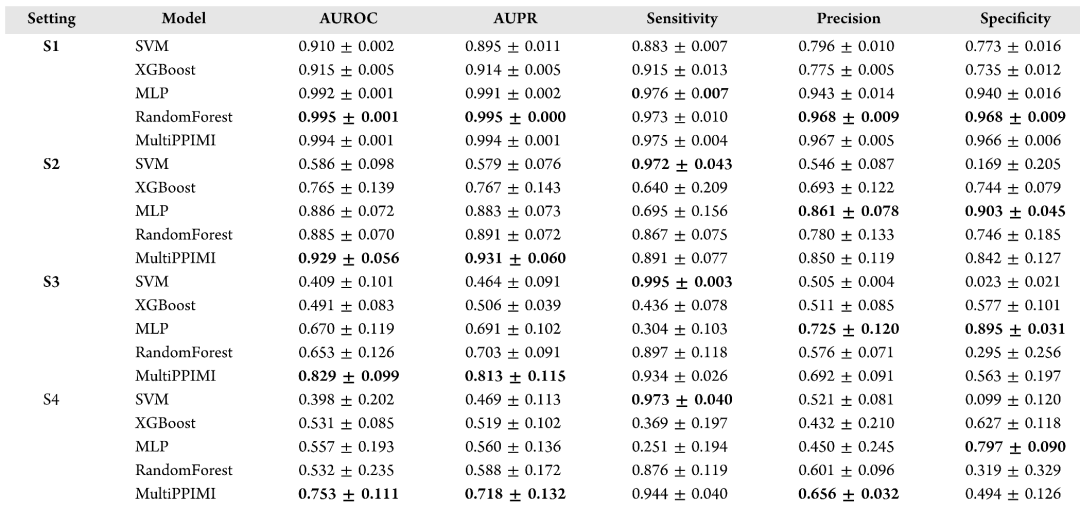
表 1
作者将benchmark数据集按照四种方法划分(S1-S4),其中S1为随机划分,S2为调节剂冷启动,S3为PPI靶标冷启动,S4为调节剂和PPI成对冷启动。作者在这四种场景下用5折交叉验证分别评估了MultiPPIMI以及四种基线模型(表1),包括SVM、XGBoost、MLP和Random Forest。在随机划分场景下,MultiPPIMI、MLP、RandomForest都达到了0.99以上的AUROC和AUPR。为了排除过拟合的可能性,作者又在三种冷启动场景中对模型评估,MultiPPPIMI所达到的AUROC和AUPR显著高于其它模型,表现出了更强的泛化能力。
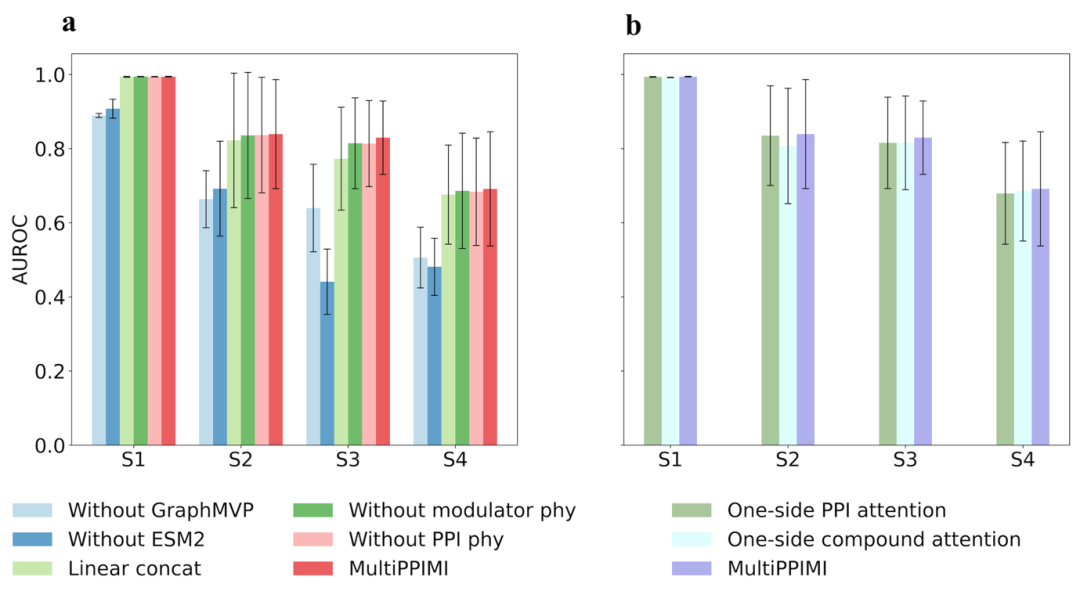
图2. 消融实验结果
作者通过消融实验进一步分析MultiPPIMI中各模块的贡献度。在四种数据划分的场景下,移除任何一种模态的特征或仅仅将特征简单拼接都会导致AUROC下降(图2a)。另外,双向注意力机制往往优于单向注意力机制(图2b),并且它们之间的差异在三种冷启动场景下更加显著,这说明了双向注意力机制对于学习广义的分子间相互作用规则的重要性。
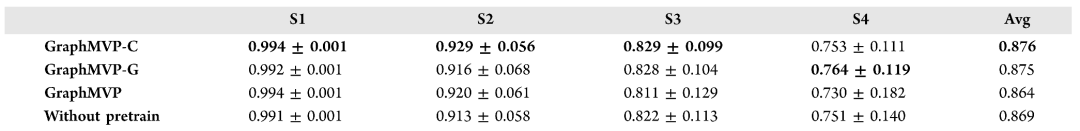
表2
作者通过使用不同的自监督学习(SSL)任务来预训练GraphMVP(小分子结构编码器),以研究不同预训练任务对PPI-调节剂相互作用预测的影响。表2展示了MultiPPIMI通过不同SSL任务预训练的GraphMVP变体在四种数据划分场景下的AUROC。这些变体包括:无预训练;GraphMVP原始预训练任务,及学习3D几何和2D拓扑之间的对应关系;GraphMVP-C,及在GraphMVP基础上添加一个2D对比学习任务;GraphMVP-G,及在GraphMVP基础上添加一个2D生成SSL任务。平均而言,GraphMVP-C和GraphMVP-G的表现优于无预训练的GraphMVP。这一观察表明,在预训练阶段,不同的模态可以相互作为补充,从而提升PPI-调节剂相互作用的预测准确率。然而,原始的SSL任务(GraphMVP)并不优于无预训练的变体,这表明预训练对PPI-调节剂相互作用预测的贡献取决于SSL任务的选择。
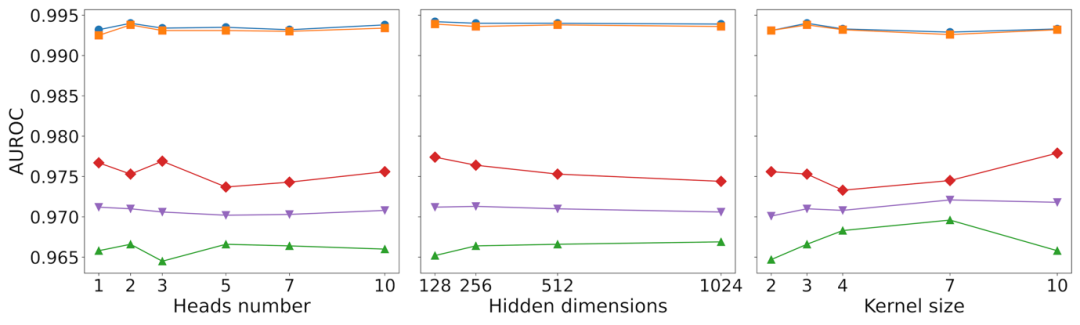
图3. 参数敏感性分析
作者还分析了三个关键参数对MultiPPIMI性能的影响:双线性注意力模块中的注意力头数、隐藏维度和核大小。图3显示了在五折交叉验证中MultiPPIMI对这些参数是鲁棒的,只展现出轻微的AUROC波动。此外,多头注意力始终优于单头注意力,这可以归因于多头注意力可以从多个特征空间建模调节剂和PPI靶标之间的相互作用,这与原子和氨基酸之间存在的多种非共价相互作用类型的观测一致。
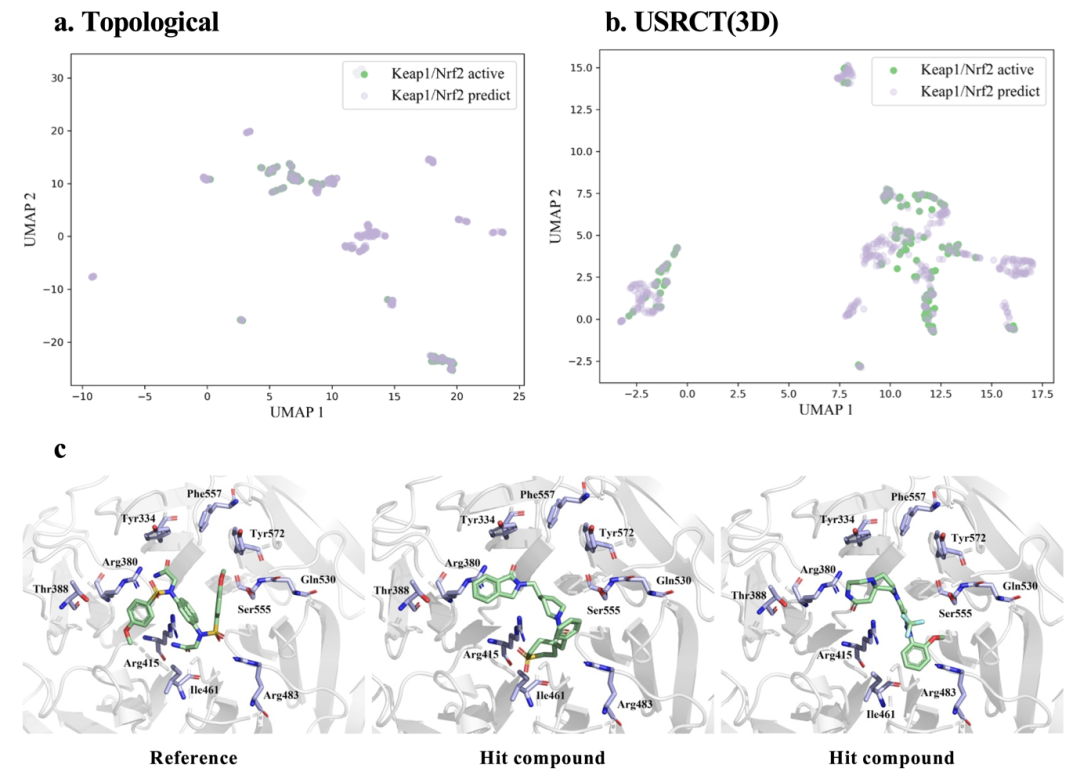
图4. 案例分析
由于针对PPI靶标的小分子库的初筛命中率通常较低,作者提出用MultiPPIMI的预测结果作为起点,在此基础上做基于结构的虚拟筛选,从而提升PPI调节剂筛选的效率和命中率。Keap1/Nrf2 是调节抗氧化防御的重要PPI靶标,与包括神经退行性、自身免疫和代谢紊乱在内的多种疾病有关。作者使用新合成的DLiP-PPI库作为虚拟筛选库,该库经过去重后包含15,074个化合物,它与MultiPPIMI的训练数据集不重叠。作者用MultiPPIMI预测虚拟库中的小分子与Keap1/Nrf2相互作用的概率,概率大于0.7被选为命中化合物。作者通过映射化合物的MACCS指纹和USRCAT指纹的UMAP图评估了Keap1/Nrf2的活性抑制剂和MultiPPIMI预测的抑制剂的化学空间(图4a,图4b)。MultiPPIMI预测的命中化合物和已知的活性抑制剂的化学空间重叠,并且命中化合物扩展了更多的化学空间。作者进一步对MultiPPIMI的命中化合物进行了基于分子对接的虚拟筛选,保留对接分数优于参考化合物的化合物。如图4c所示,命中化合物和参考化合物与由Tyr334、Arg380、Thr338、Arg415、Ile461、Arg483、Gln555、Ser555和Tyr572残基形成的结合口袋结合。命中化合物表现出比参考化合物更强的结合亲和力。结果表明,深度学习工具能够以数据驱动的方式挖掘PPI-调节剂相互作用的潜在规律,从而提供一个与基于物理规则的方法不同的视角,结合两种方式可以加速PPI调节剂的筛选。
结论
这项研究中,作者提出了MultiPPIMI,这是一种通用的多模态深度学习模型,通过预测PPI靶标与调节剂的相互作用,实现PPI调节剂的虚拟筛选。该模型整合了PPI靶标和小分子的预训练structural embedding和物理化学性质,通过双线性注意力机制学习广义的分子间相互作用规则。
参考资料
Sun, H.; Wang, J.; Wu, H.; Lin, S.; Chen, J.; Wei, J.; Lv, S.; Xiong, Y.; Wei, D.-Q. A Multimodal Deep Learning Framework for Predicting PPI-Modulator Interactions. J Chem Inf Model 2023. https://doi.org/10.1021/acs.jcim.3c01527.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


