为解决上述蛋白骨架设计问题,近日,瑞士洛桑联邦理工学院的Bruno E. Correia课题组提出了一种基于卷积变分自编码器(convolutional variational autoencoder, CVAE)的蛋白骨架生成方法Genesis [1],该方法能够基于给定的蛋白结构草图(protein sketch)生成全新的、可设计的蛋白骨架,基于这些骨架设计的序列经计算和实验验证能够很好地折叠成预期的结构。进一步地,作者通过系统的蛋白骨架设计流程生成了自然界中尚不存在的全新蛋白结构(”darkfolds”),并成功地进行了实验验证,从而向探索蛋白质宇宙中的未知结构迈出了重要的一步。
——方法——
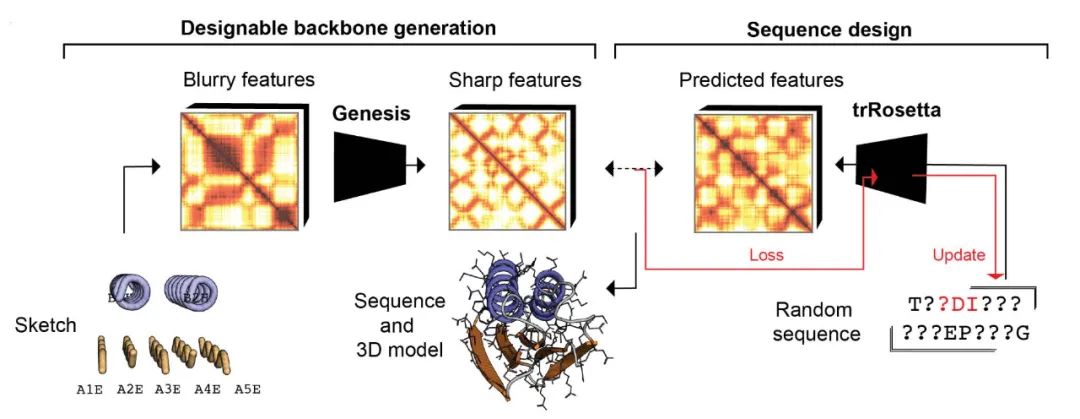
图1. 模型整体架构及蛋白设计流程
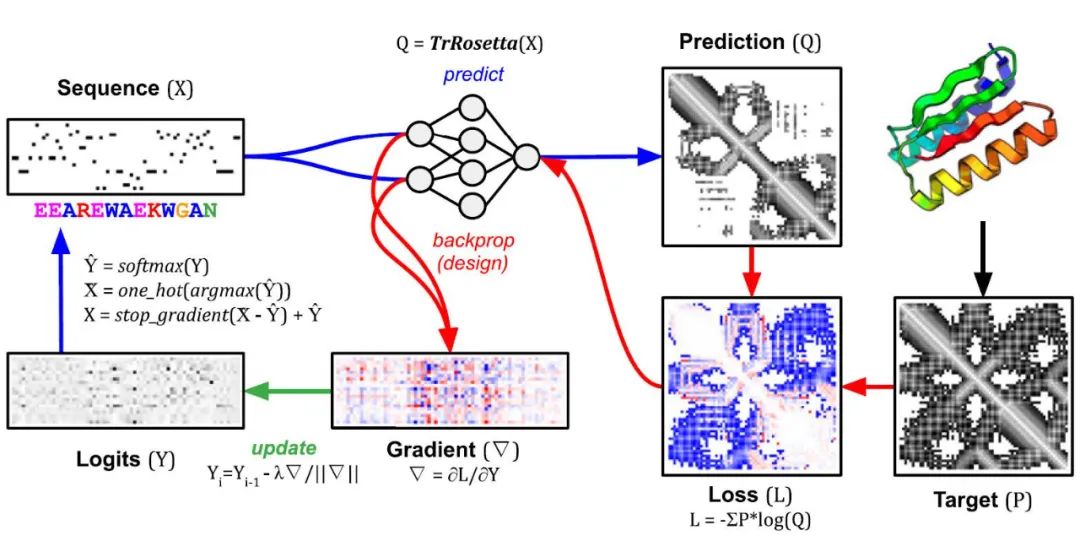
图2. trDesign进行蛋白序列设计的流程
作者提出的模型整体架构及蛋白设计流程如图1所示,整个蛋白设计流程分为蛋白骨架生成和固定骨架的蛋白序列设计两部分。在蛋白骨架生成过程中,作者首先根据先前的工作TopoBuilder [2] 所定义的方式将预期的SSE排列顺序(即蛋白结构草图,protein sketch)转化成初始的蛋白骨架结构,随后将初始结构转化为trRosetta [3] 的特征图(feature maps)输入Genesis模型,通过直接对基于成对距离和扭转角的特征图进行操作保证了模型的SE(3)等变性。Genesis模型对输入的模糊的特征图进行降噪,再还原为蛋白质主链的三维结构,从而生成可设计的蛋白骨架。在固定骨架的蛋白序列设计过程中,作者使用David Baker课题组发展的trDesign [4] 方法对生成的骨架进行序列设计,trDesign的具体流程如图2所示。
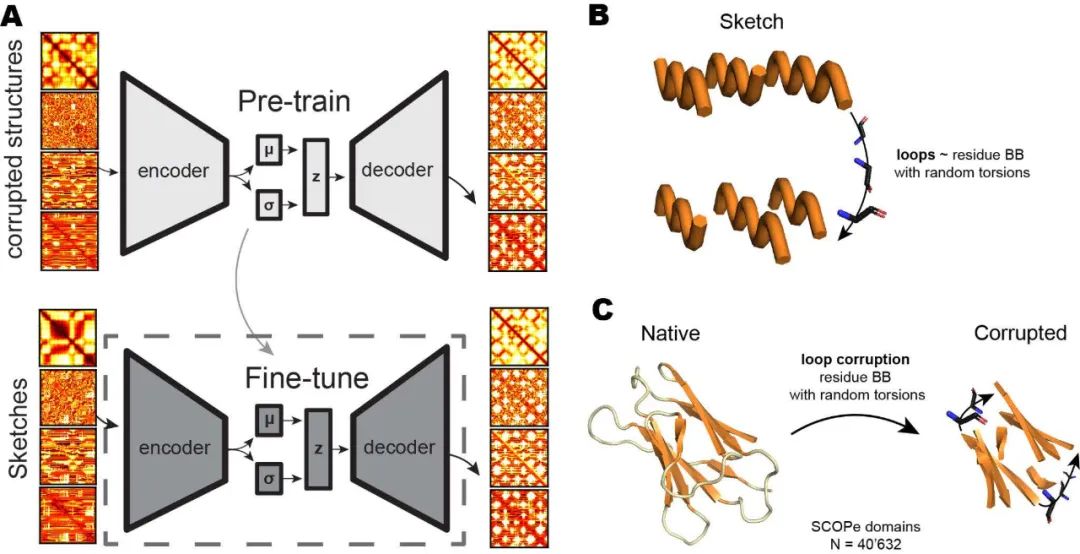
图3. Genesis模型的训练过程及训练数据
如图3A所示,Genesis模型以CVAE为主体架构,编码器为4层2D卷积层(2D-conv),解码器为3层2D解卷积层(2D-deconv),其训练过程分为预训练和微调两个阶段。在预训练阶段,如图3C所示,作者筛选SCOPe训练集中长度为40~128 AA的40726个结构域,移去所有loop结构,将loop区原有的残基平均分布在两端Cα原子的连线上,并且随机初始化所有loop区残基的扭转角,形成Genesis模型的输入结构,模型的任务是将loop区恢复成天然的构象。在微调阶段,如图3B所示,作者基于TopoBuilder所提出的方式遍历了不同SSE所组成的可能的蛋白结构草图,按照预训练中的方式初始化loop区构象,在SCOPe训练集中搜索与这些初始结构RMSD<3 Å的天然结构,共得到35435对结构,随后训练模型将这些初始结构降噪成真实的蛋白结构。在训练过程中,模型的损失函数为4类特征图对应的4个重建损失(reconstruction loss)和1个隐空间的KL散度损失(KL divergence loss),其中重建损失的形式是p=1的Wasserstein距离,如下式所示: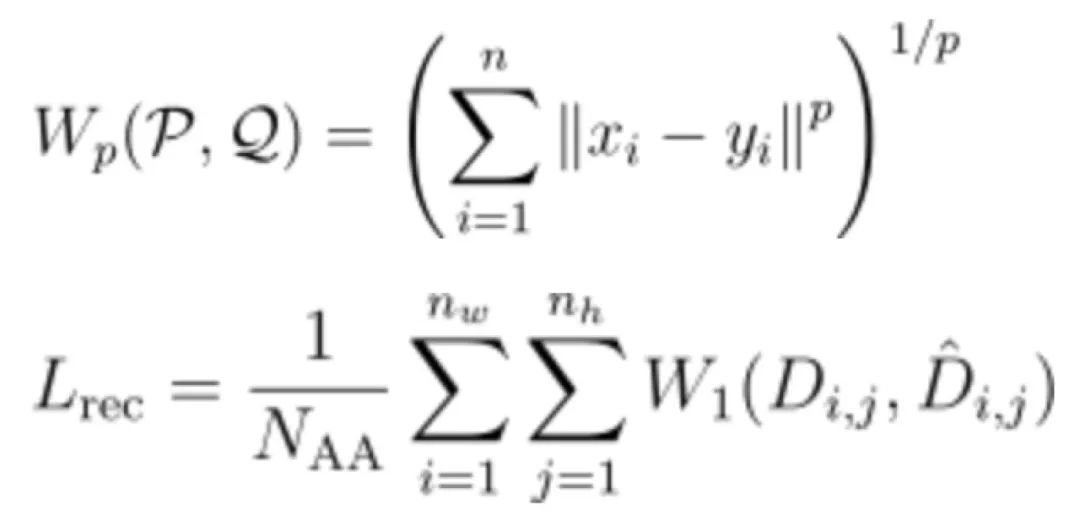
总的损失函数为:
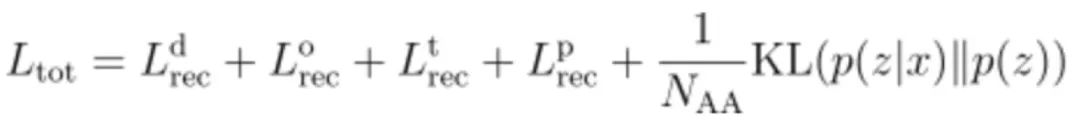
——结果——
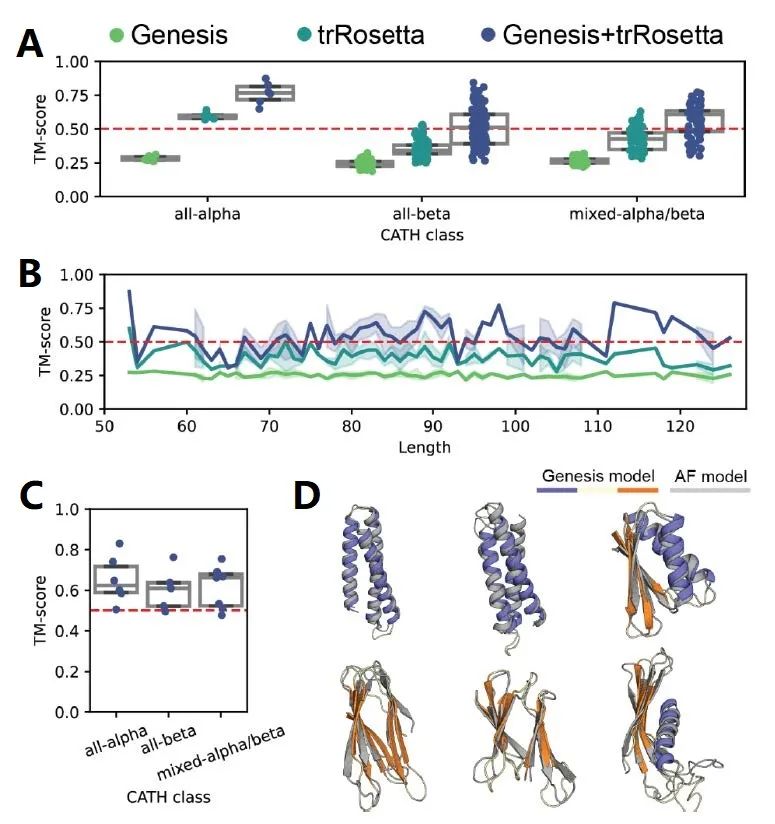
图4. Genesis-trDesign蛋白设计流程在SCOPe数据集上的表现
作者首先评估了模型在SCOPe数据集上的表现,如图4所示,其中图4A和图4B是基于训练集中随机挑选的273个结构进行重新设计的结果,图4C和图4D是在测试集上得到的结果。可见Genesis和trDesign两个模块在蛋白设计任务中都具有重要功能,而Genesis-trDesign设计流程在不同CATH类、不同序列长度下都获得了较好的设计效果,并且能够很好地泛化到训练集中未出现的结构上。
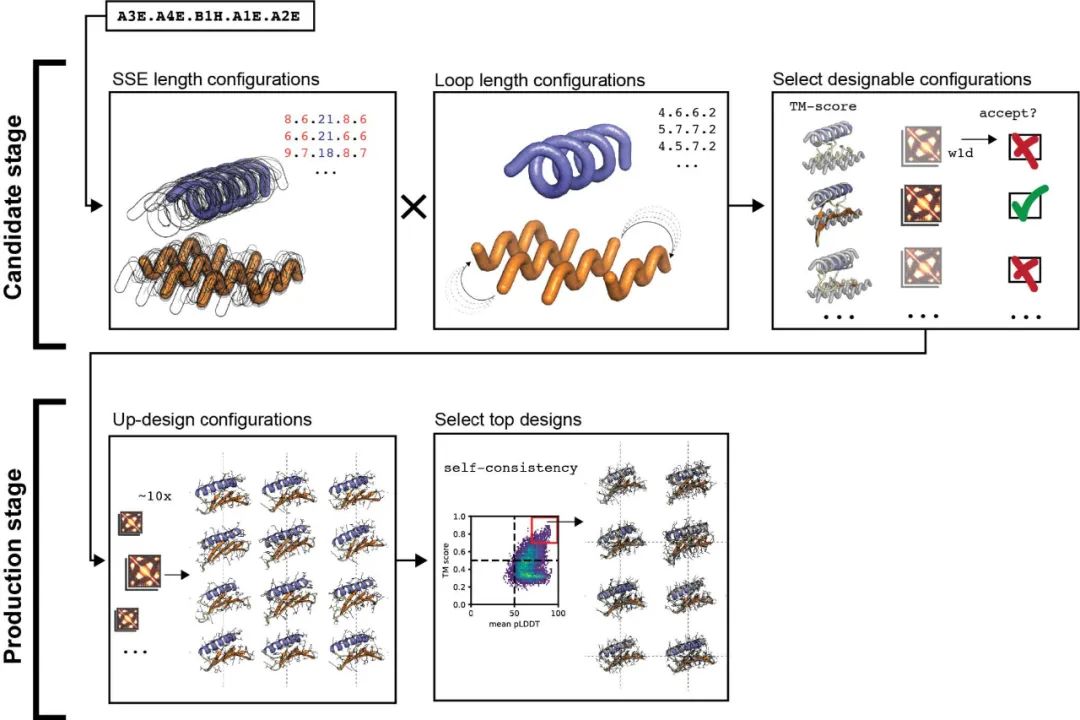
图5. 天然蛋白拓扑结构大规模从头设计的两阶段采样策略
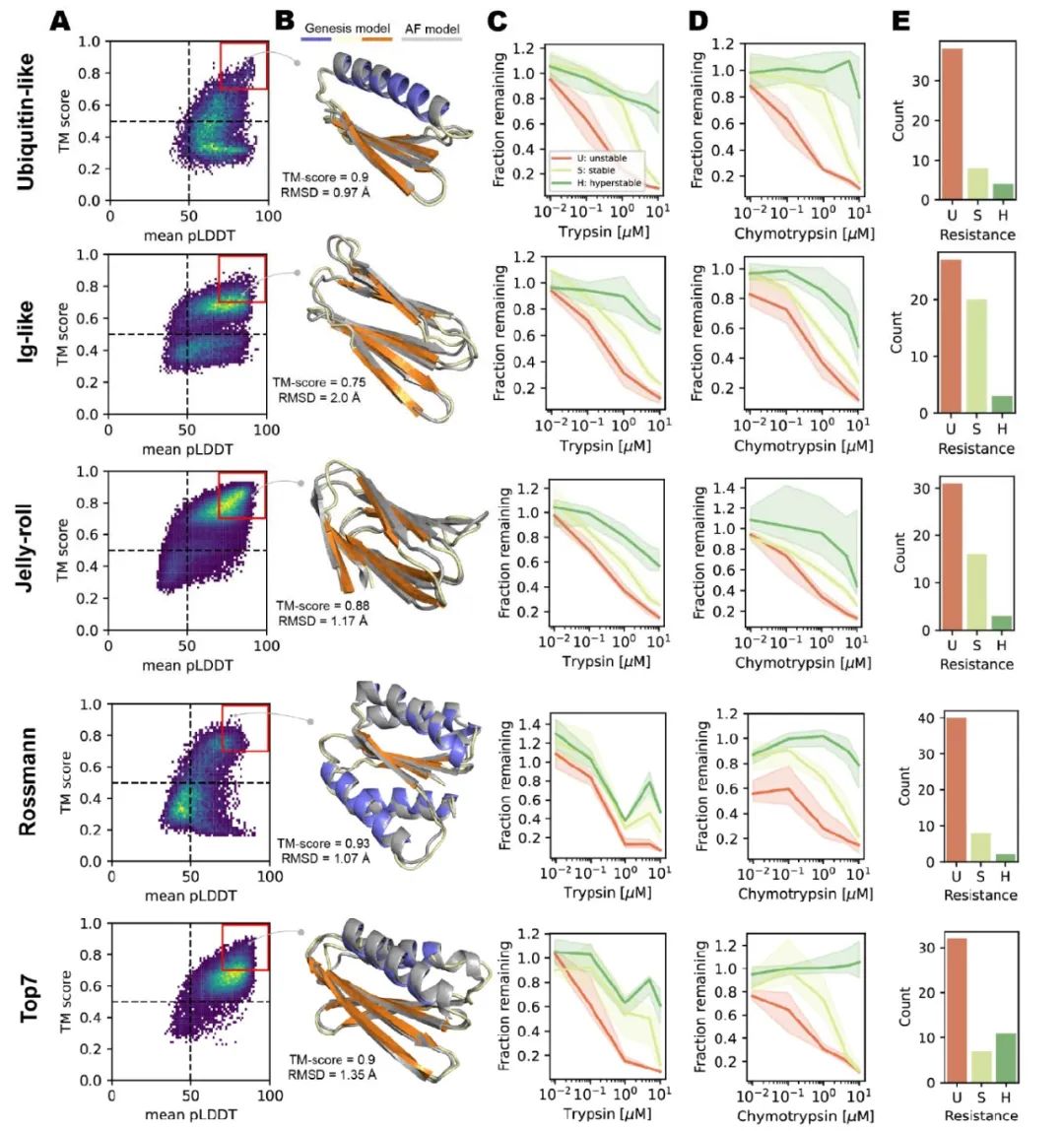
图6. 天然蛋白拓扑结构大规模从头设计的结果
作者随后使用两阶段采样策略对Ubiquitin-like、Ig-like、Jelly-roll和Rossmann 4种天然蛋白拓扑结构(native topologies/folds)及Top7 fold(第一个从头设计的蛋白结构 [5],表征模型的泛化性能)进行了大规模从头设计,方法如图5所示。具体地,作者在候选结构搜索阶段(candidate search stage)遍历天然拓扑结构对应的所有可能的SSE组成、长度及loop区长度,生成大量可能的初始结构并进行小规模初步设计,根据生成结构的TM-score和Wasserstein距离筛选出可行的初始结构。随后,作者在产出阶段(production stage)对上述初始结构进行大规模(每个结构约20000条)序列设计,根据TM-score和AF2 pLDDT进行第二轮筛选,每个fold挑选50条序列进行酵母文库(yeast library)的构建,通过实验验证所设计序列的稳定性,结果如图6所示。可见作者所提出的Genesis-trDesign蛋白设计流程对5个fold都成功设计出了多条稳定折叠成期望结构的序列,具有相当程度的实用价值。
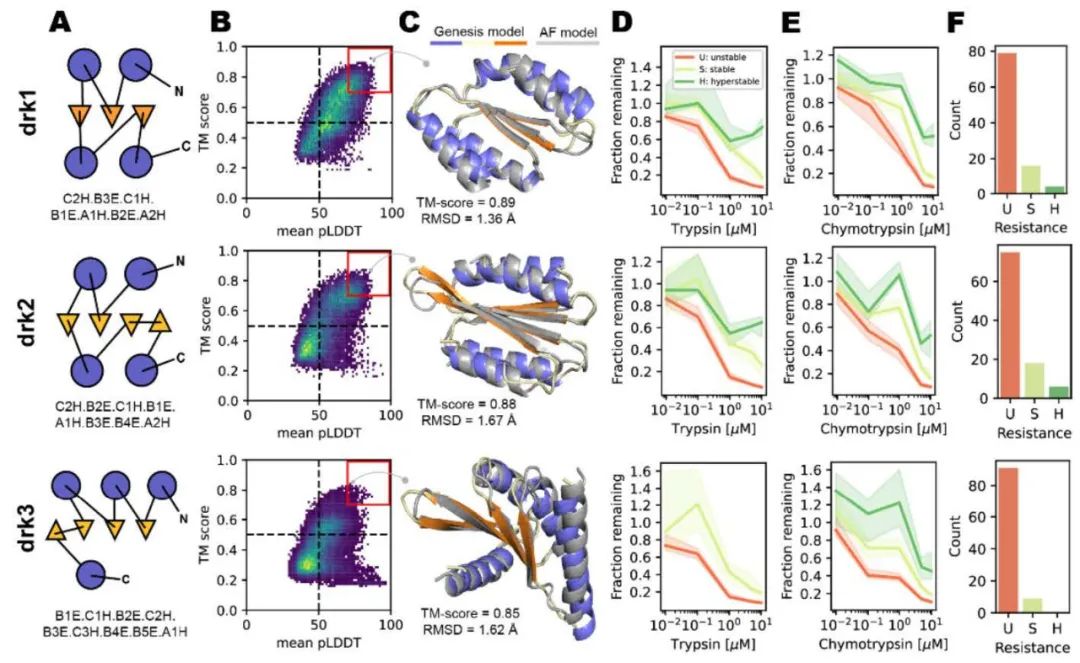
图7. 全新蛋白拓扑结构大规模从头设计的结果
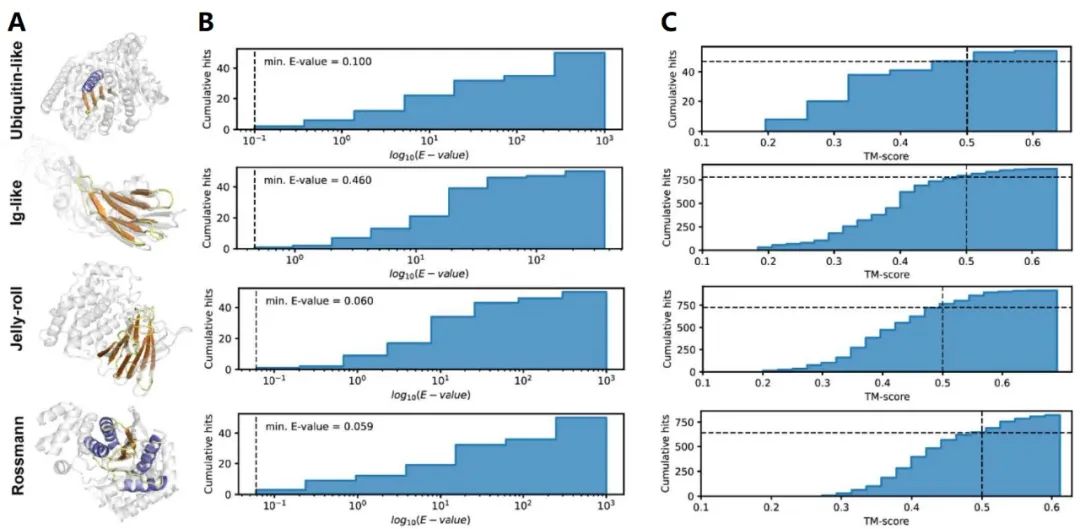 图8. 全新蛋白拓扑结构大规模从头设计的序列及结构比对
图8. 全新蛋白拓扑结构大规模从头设计的序列及结构比对
如图7所示,作者随后使用类似的方法对自然界中不存在的全新蛋白拓扑结构进行了大规模从头设计,选取其中的3种fold进行第二阶段的序列设计,经实验验证成功得到了能够稳定折叠成期望结构的蛋白序列。进一步地,如图8所示,作者对所设计的蛋白(图8A, 与最接近的结构进行了alignment)使用BLASTp [6] 在Non-Redundant Database(NR)中进行了序列比对(图8B)、使用FoldSeek [7] 在PDB中进行了结构比对(图8C),结果显示作者所设计的全新蛋白无论结构还是序列都具有显著的新颖性。
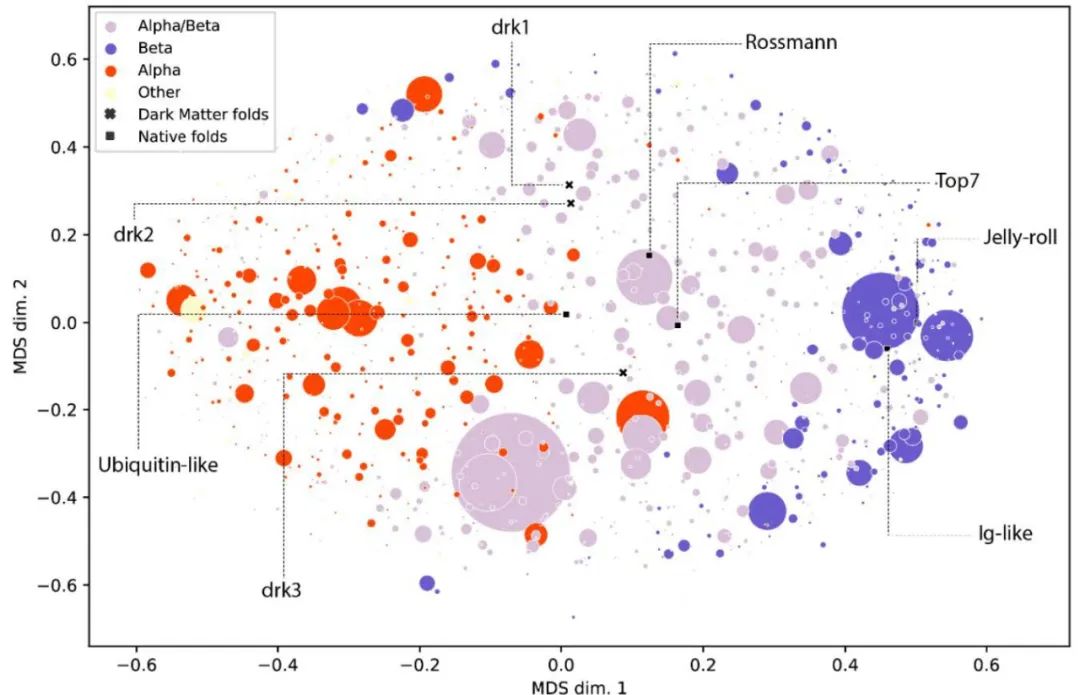 图9. 全新蛋白结构及已有蛋白结构的降维可视化作者最后使用多维标度(Multidimensional Scaling, MDS)方法对得到的全新蛋白结构以及CATH数据库中已有的蛋白结构进行了降维可视化,如图9所示。可见作者所设计的蛋白不属于任何一个已知的CATH fold,是蛋白宇宙中的“暗物质”。使用Genesis-trDesign进行的蛋白从头设计流程能够探索未知的、全新的蛋白结构,对于蛋白质理性设计领域有着重要的意义。
图9. 全新蛋白结构及已有蛋白结构的降维可视化作者最后使用多维标度(Multidimensional Scaling, MDS)方法对得到的全新蛋白结构以及CATH数据库中已有的蛋白结构进行了降维可视化,如图9所示。可见作者所设计的蛋白不属于任何一个已知的CATH fold,是蛋白宇宙中的“暗物质”。使用Genesis-trDesign进行的蛋白从头设计流程能够探索未知的、全新的蛋白结构,对于蛋白质理性设计领域有着重要的意义。
——总结——
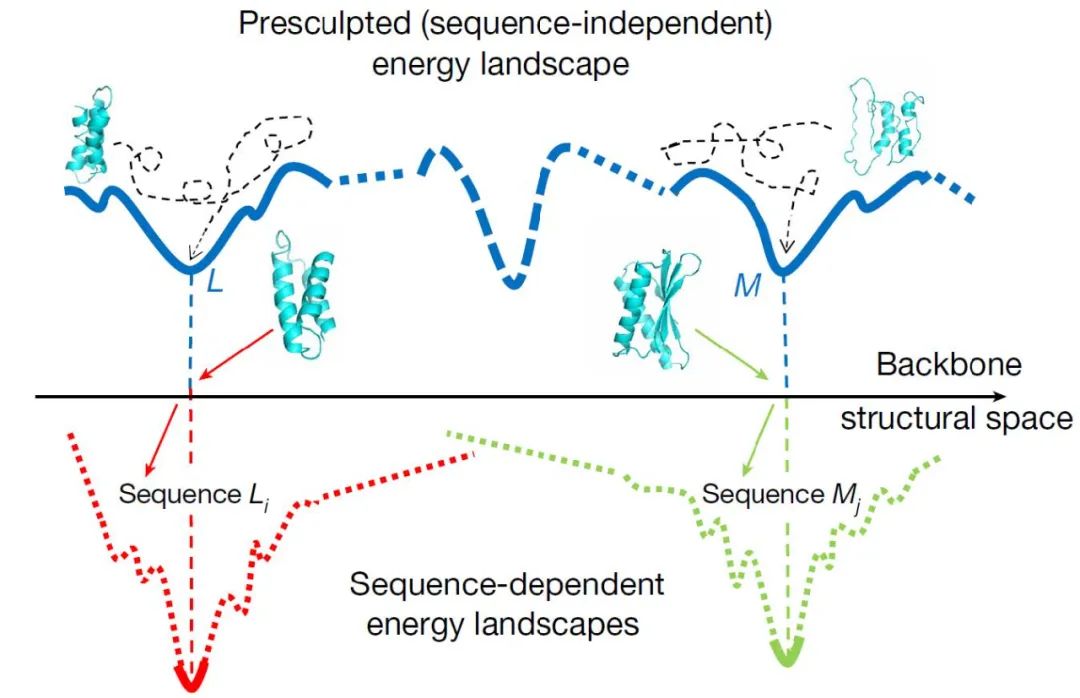
图10. 提升蛋白骨架可设计性对蛋白质从头设计的意义 [8]
1、本文介绍了一种基于CVAE的蛋白骨架生成方法Genesis,该方法能够基于给定的蛋白结构草图系统性地生成全新的、可设计的蛋白骨架,基于这些骨架设计的蛋白序列经计算和实验验证能够很好地折叠成预期的结构。作者认为,通过将蛋白骨架表示成trRosetta中的特征图,Genesis-trDesign流程不需要直接一步生成最终的3D蛋白骨架结构,而是可以在蛋白序列-结构空间中进行更为高效的优化。2、形象地,如图10所示,Genesis模块首先通过提升蛋白骨架的可设计性极大地限制了序列-结构空间中的搜索范围,而随后trDesign模块在有限的搜索空间中进行序列设计时对蛋白骨架可能的噪音具有很好的鲁棒性,上述流程相较一些序列-结构共同设计(sequence-structure co-design)方法可能能够更好地找到序列-结构空间的极小值点,也即能够稳定折叠成预期结构的序列。3、Genesis-trRosetta设计流程很好地解决了基于结构草图生成特定形状的全新蛋白的问题,对蛋白质从头设计领域具有相当程度的启发性,同时也证明了发掘自然界中尚不存在的全新蛋白结构的巨大潜力。4、当然,该方法目前仍存在一些问题,例如在给定结构草图进行全新蛋白质设计时仍需进行大规模遍历和筛选,整个过程的时间成本不应忽视,而如何将Genesis-trDesign流程与已有的基于扩散的蛋白生成模型相结合可能是有探索价值的方向。
参考文献:[1] Harteveld, Z., Van Hall-Beauvais, A., Morozova, I., Southern, J., Goverde, C. A., Georgeon, S., … & Correia, B. (2023). Exploring “dark matter” protein folds using deep learning. bioRxiv, 2023-08.[2] Harteveld, Z., Bonet, J., Rosset, S., Yang, C., Sesterhenn, F., & Correia, B. E. (2022). A generic framework for hierarchical de novo protein design. Proceedings of the National Academy of Sciences, 119(43), e2206111119.[3] Yang, J., Anishchenko, I., Park, H., Peng, Z., Ovchinnikov, S., & Baker, D. (2020). Improved protein structure prediction using predicted interresidue orientations. Proceedings of the National Academy of Sciences, 117(3), 1496-1503.[4] Norn, C., Wicky, B. I., Juergens, D., Liu, S., Kim, D., Tischer, D., … & Ovchinnikov, S. (2021). Protein sequence design by conformational landscape optimization. Proceedings of the National Academy of Sciences, 118(11), e2017228118.[5] Kuhlman, B., Dantas, G., Ireton, G. C., Varani, G., Stoddard, B. L., & Baker, D. (2003). Design of a novel globular protein fold with atomic-level accuracy. Science, 302(5649), 1364-1368.[6] Altschul, S. F., Gish, W., Miller, W., Myers, E. W., & Lipman, D. J. (1990). Basic local alignment search tool. Journal of molecular biology, 215(3), 403-410.[7] van Kempen, M., Kim, S. S., Tumescheit, C., Mirdita, M., Lee, J., Gilchrist, C. L., … & Steinegger, M. (2023). Fast and accurate protein structure search with Foldseek. Nature Biotechnology, 1-4.[8] Huang, B., Xu, Y., Hu, X., Liu, Y., Liao, S., Zhang, J., … & Liu, H. (2022). A backbone-centred energy function of neural networks for protein design. Nature, 602(7897), 523-528.
点击左下角的”阅读原文“即可查看原文章。
作者:王宇哲审稿:王丽莹编辑:王丽莹
GoDesignID:Molecular_Design_Lab( 扫描下方二维码可以订阅哦!)



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


