编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Angela Ruohao Wu 和Can Yang团队的一篇论文。空间转录组学(ST)技术正在革新探索组织空间结构的方式。目前,ST数据分析通常局限于单个二维(2D)组织切片,这限制了我们理解在三维(3D)空间中发生的生物过程的能力。在这里,作者介绍了STitch3D,这是一个统一的框架,它整合多个ST切片以重建3D细胞结构。通过联合建模多个切片并将其与单细胞RNA测序数据整合,STitch3D同时识别出具有一致基因表达水平的3D空间区域,并揭示了3D细胞类型分布。

每个单独的组织切片描述了一个二维的转录组景观,多个切片的联合建模则提供了描绘生物系统三维画面的机会。以下两个三维组织分析任务十分重要。第一个任务是识别生物学上可解释的具有相似基因表达的三维空间区域,以揭示组织结构。然后,结果可以进行如检测与区域相关的具有三维空间模式的基因等下游分析。第二个任务是通过整合多个ST切片和单细胞RNA测序(scRNA-seq)图谱来推断三维细粒度的细胞类型分布。现有的下一代测序ST技术可以检测空间点内的全基因组基因表达,但每个点通常包含多个细胞,导致分辨率相对较低。利用来自scRNA-seq图谱的信息,三维细胞类型解卷积任务解析空间点中的细胞类型混合物,从而实现三维重建的更高分辨率,允许洞察特定细胞类型富集区域的生物功能。在文中,作者介绍了STitch3D,用于使用多个切片描述三维组织结构。它以统一的框架解决上述两个三维分析任务,提供三维组织结构的补充视图。通过有效地模拟来自多个切片的基因表达和空间位置,STitch3D可以区分切片间的生物变异和批处理效应,并整合跨切片的信息来组装强大的三维组织模型。
模型部分
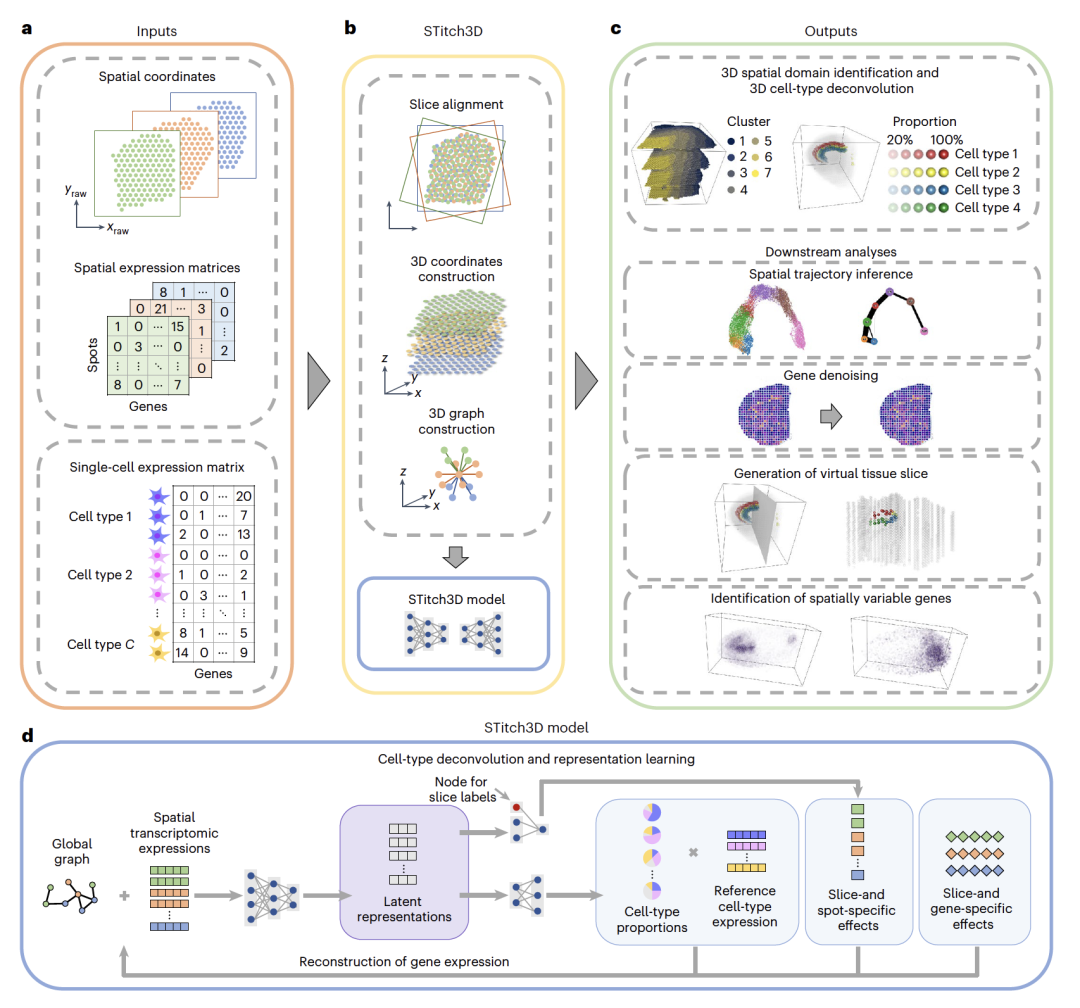
图 1
STitch3D是一种基于深度学习的方法,它使用多个二维切片来重建三维组织结构(见图1)。STitch3D的输入是多个切片和一个匹配的单细胞RNA测序(scRNA-seq)。预处理步骤包括通过对齐切片来建立三维空间点坐标,并构建全局三维邻域图。在这些步骤之后,STitch3D被训练以整合所有切片的信息。引入了一个共享的潜在空间来提取有意义的生物变异,并促进批处理效应的移除。在潜在空间中,每个点都有其表示,这用于共同执行空间域识别和细胞类型解卷积任务。STitch3D通过基于图注意力的网络将多个切片的点的基因表达和空间信息映射到共享的潜在空间,该网络利用了点的三维邻域图。此外还引入一个判别网络以从潜在表示中推断细胞类型比例。在整合多个切片的有效批处理效应建模下,STitch3D被训练以通过结合估计的细胞类型比例和细胞类型特异性基因表达谱来重建基因表达。
基准测试
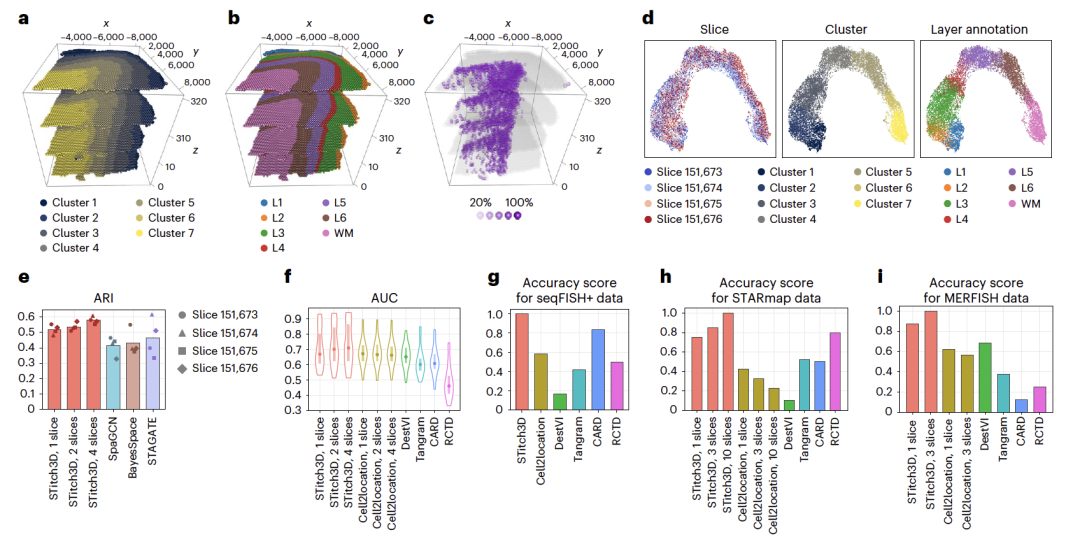
图 2
作者首先使用DLPFC数据集评估了STitch3D在空间域检测方面的表现。数据集标记了六层切片(L1-L6)和白质(WM)。当应用于每个单独的切片时,STitch3D稳定地恢复了层结构。在多切片分析中应用时,STitch3D产生了跨切片一致的结果,促进了三维重建。STitch3D的结果与原数据相比具有相似的模式,表明其可靠性(图2a,b)。为了定量评估,作者将数据标记视为真实情况。STitch3D的多切片结果与其单切片结果相比,获得了更高的ARI分数,表明STitch3D能够跨切片借用信息(图2e)。作者还将STitch3D与包括BayesSpace、SpaGCN和STAGATE在内的空间域识别方法进行了比较。与STitch3D不同,这些方法在检测皮质层方面显示出较差的能力(图2e)。STitch3D在三维空间区域识别方面的优势归因于其能在共享潜在空间中有效整合切片(图2d)。接下来,作者评估了STitch3D的细胞类型解卷积性能,STitch3D在多切片分析中比单切片结果获得了更高的AUC分数(图2f)。
重建鼠脑
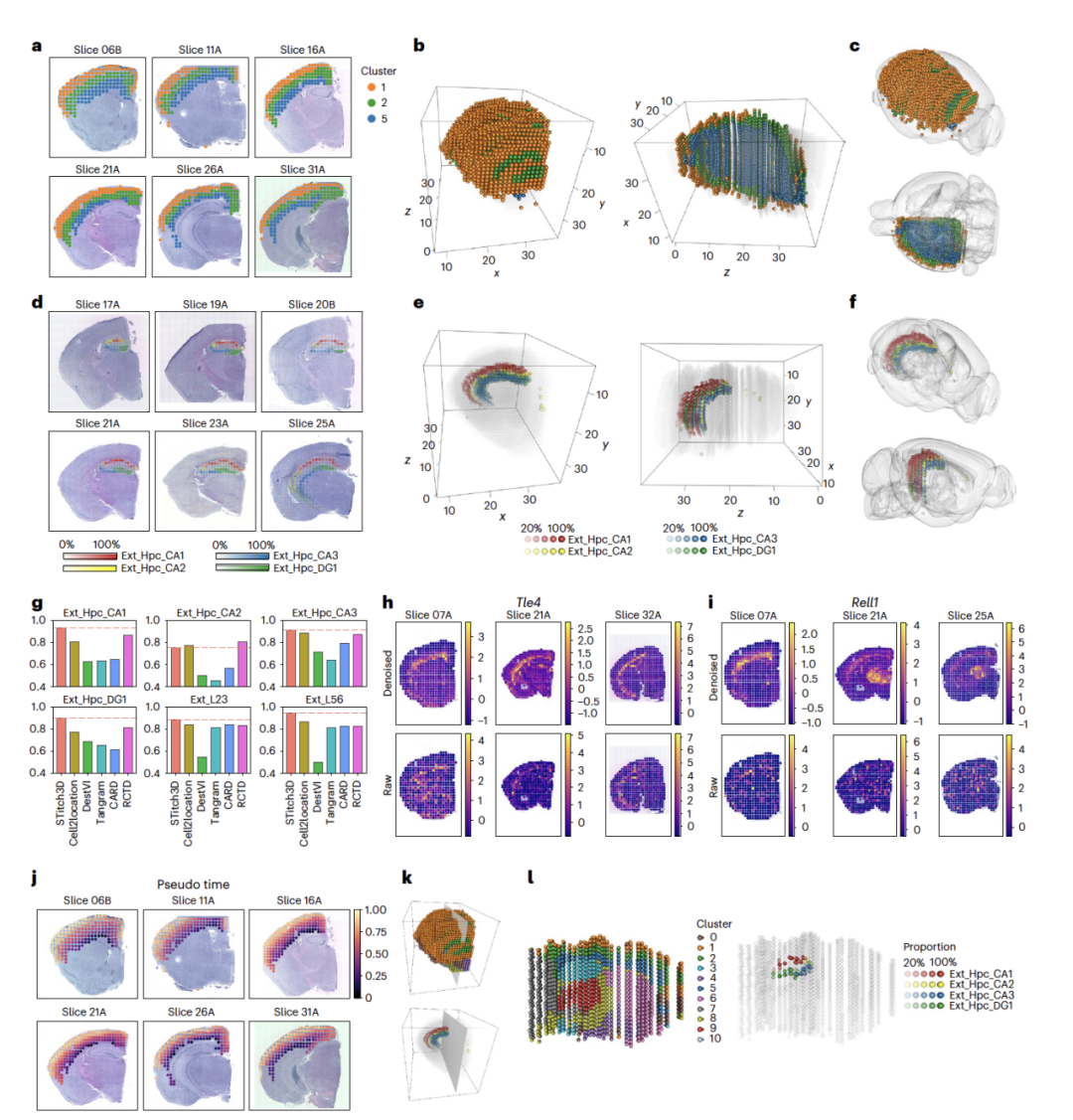
图 3
作者展示了STitch3D如何准确重建复杂的成年小鼠大脑三维结构。这里的三维重建任务具有挑战性,因为它要求方法考虑数十个切片中的批处理效应,并区分细微的细胞亚型差异。STitch3D基于综合点表示将大脑划分为有序的三维域。例如被标记为簇1、2和5的三个层状结构域形成了等皮层区域(图3a-c)。这些簇在轨迹推断中显示出强连通性。然后,在这些簇中进行的假时分析显示了所有切片中与皮层生成一致的模式(图3j)。利用参考中的细粒度细胞类型特征,STitch3D揭示了三维细胞类型分布。例如,它准确重建了在角回(CA)和齿状回(DG)的四种海马神经元类型的分布(图3e,f)。这些分布正确地与参考图谱中标注的海马区域CA1、CA2、CA3和DG相匹配(图3d)。STitch3D还捕获了皮层层中兴奋性神经元的分布,以及其他主要区域亚型。对于方法之间的定量比较,作者使用了四种海马神经元类型的估计比例来恢复CA1、CA2、CA3和DG区域,并使用ROC分析与区域注释进行了比较。类似的分析也针对2-3层和5-6层中的两种皮层神经元类型进行(图3g)。STitch3D的单切片分析已经显示出与其他方法相比整体更好的AUC分数。通过跨切片借用信息,STitch3D在其多切片分析中实现了更好的准确度。
HER2阳性乳腺癌数据
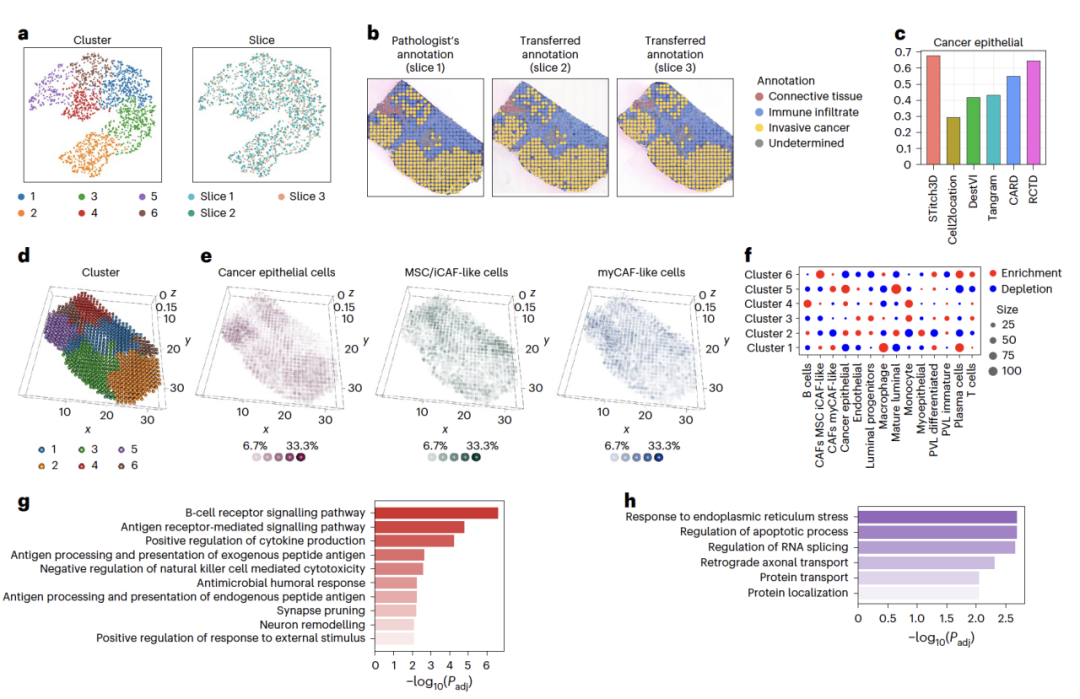
图 4
作者分析了HER2阳性乳腺癌数据,展示了STitch3D在分析异常组织中生物学发现方面的能力。除了正常组织数据外,作者还将STitch3D应用于在结核分枝杆菌诱导后的小鼠淋巴结数据和小鼠皮肤伤口愈合期间的数据,以展示其捕捉微观层面对干扰的反应的能力。在将STitch3D应用于来自八名患者的肿瘤样本后,计算了全局细胞类型共定位情况。作者在所有样本中发现了一些一致的结果,例如,两种与癌症相关的成纤维细胞(CAFs)之间的负空间相关性。此外,还发现间充质干细胞和炎症样CAFs(MSC/iCAF-like)与包括B细胞、T细胞和浆细胞在内的免疫细胞共定位,与肌成纤维样CAFs(myCAF-like)的模式不同。然后,作者重点关注了E患者的结果。在三个切片中,第一个切片由病理学家注释。使用STitch3D,三个切片被很好地整合(图4a),这说明模型能够将病理学家的注释转移到未标记的切片上(图4b)。STitch3D还识别了六个空间区域(图4a,d)。具体来说,簇2-5恢复了侵袭性癌症区域,STitch3D确实发现癌上皮细胞在这些簇中与其他区域相比富集(图4e,f)。癌上皮细胞比例与乳腺癌标记物ERBB2表达之间的高相关性支持了STitch3D的结果(图4c)。此外,两种CAF亚型的分布模式显示出不同(图4e),这与它们的细胞类型比例负相关性一致。接下来,作者研究了癌区域的异质性。簇4和5都显示出癌上皮细胞的富集。然而,簇4显示出B细胞和T细胞的更高富集,而簇5显示出癌上皮细胞的更高富集(图4f)。作者对这些簇进行了差异表达分析,随后进行了GO分析(图4g,h)。这些富集的生物学过程之间的区别表明这两个区域的差异,凸显了它们各自不同的微环境。
参考资料
Wang, G., Zhao, J., Yan, Y. et al. Construction of a 3D whole organism spatial atlas by joint modelling of multiple slices with deep neural networks. Nat Mach Intell 5, 1200–1213 (2023).
https://doi.org/10.1038/s42256-023-00734-1



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


