structure prediction,PSSP)模型的论文。本文通过将CondConv与GCNN模型相结合提出了CondGCNN模型,并使用集成编码器来融合LSTM和CondGCNN的特征提取结果,能够更好地捕获蛋白质序列特征进而编码蛋白质序列。此外,本文探讨了蛋白质二级结构预测问题和图像分割问题之间的相似性,并基于空洞空间金字塔池化模型提出ASP网络,以捕捉精细的蛋白质二级结构边界细节。本文通过在多种数据集上进行广泛实验表明,本文提出的模型在蛋白质二级结构预测任务上的性能优于现有方法。
1 介绍:
蛋白质的结构决定其具体功能,如果蛋白质的结构发生改变,则蛋白质可能变性并且不能如预期地发挥相应功能,因此对于蛋白质结构的研究具有重要意义。蛋白质是由肽键连接而成的氨基酸链,从氨基酸序列直接预测蛋白质的三维结构是一项具有挑战性的任务,因此对于蛋白质二级结构的预测是这一任务的重要组成部分。虽然基于氨基酸序列编码器的深度学习方法已经取得了巨大的成功,但对于蛋白质二级结构之间关系的研究仍比较少。比如DeepCNF方法采用条件随机场(CRF)作为输出层,以学习相邻二级结构标签之间的相互依赖性。然而这一模型没有考虑到蛋白质二级结构分布的特殊性,因此对于Q8精度的提高是有限的。本文提出蛋白质序列中相邻的氨基酸残基通常具有相同的二级结构,因此,蛋白质二级结构预测(PSSP)任务与图像语义分割(ISS)任务非常相似。然而,这两个任务之间存在两个不同点:①PSSP任务的输入数据是一维序列,而ISS任务的输入一般是二维数据。②对于ISS任务,由于相邻像素的池化可以有效地减小输入图像的大小,因此可以广泛地使用池化层,在保留大部分图像信息的同时减少网络参数。然而对于PSSP任务,每个位置的氨基酸信息对于蛋白质序列是至关重要的,因此池化层不适用于氨基酸序列。
本文基于PSSP任务与ISS任务的相关性提出了一种蛋白质二级结构预测模型,本文具体贡献如下:
(1)首次将图像分割处理技术应用于蛋白质二级结构预测任务,充分利用了图像分割领域的模型优势,基于ASPP模型捕获二级结构的精细边缘细节;
(2)首次将CondConv网络应用于序列处理问题,并将其嵌入GCNN中,形成一种新型的氨基酸序列编码器,在模型通道维度使用门控网络进行特征过滤,并实现了对于样本的注意力机制;
(3)通过构建集成编码器,从氨基酸序列中提取到多样的特征信息;
(4)通过广泛的消融实验,详细分析了本文模型不同组成部分对于蛋白质二级结构预测效果的重要性。
2. 方法
本文提出的模型:
本文提出的模型如图1所示,主要分为集成编码模块和蛋白质二级结构生成模块两部分。其中集成编码模块中分别使用CondGCNN模型与BLSTM模型对输入的序列信息进行特征提取,并将提取到的特征进行拼接,然后将拼接后的特征输入到蛋白质二级结构生成模块。蛋白质二级结构生成模块由本文提出的ASP网络和输出层组成,其中输出层将一般的全连接网络改为1*1卷积网络,在保证模型预测效果的同时减少了模型的参数规模。
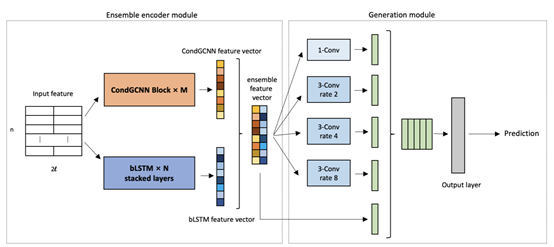
图1 本文提出模型的架构
CondGCNN模型:
CondGCNN模型及其组成部分的示意图如图2所示。图2(a)是CondGCNN模型,其由M个CondGCNN块组成,CondGCNN块的结构如图2(b)所示。每个CondGCNN块由两个CondGCNN层组成,并将输入特征与输出特征进行相加,以构建残差连接。
每层CondGCNN网络的具体架构如图2(c)所示,图2(c)左侧体现了CondCNN模型的优势,模型将输入数据通过一个Route网络获取到多个卷积核的权重,通过将多个卷积核带权相加来确定卷积核的参数值,并使用多个卷积核带权相加得到的卷积核对输入数据进行卷积操作。这样可以为每个序列样本构造不同的卷积核参数,体现了每个样本的独特性,实现了对于样本的注意力机制。图2(c)右侧体现了GCNN的优势,通过设置门控网络,在通道维度上对卷积后的特征信息进行过滤,以突出重要的特征。
CondGCNN网络的输出特征计算公式如下:
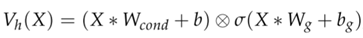
其中,Vh(X)表示CondGCNN网络的输出结果,X表示输入的序列特征,Wcond表示多个卷积核带权相加后得到的卷积核,b为卷积层的偏置,Wg表示门控网络的卷积核,bg表示门控网络的偏置,⊕表示矩阵之间进行逐点相加。
Wcond的计算公式如下:
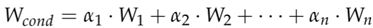
其中αi表示第i个卷积核的权重,Wi表示第i个卷积核的具体参数。
Route网络的具体计算公式如下:
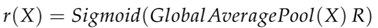
其中r(X)表示Route网络的输出结果,结果中包含每个卷积核所占的权重,X表示输入特征,R表示一个矩阵,负责将输入特征进行全局平均池化后的结果映射到每个卷积核所占的权重上。
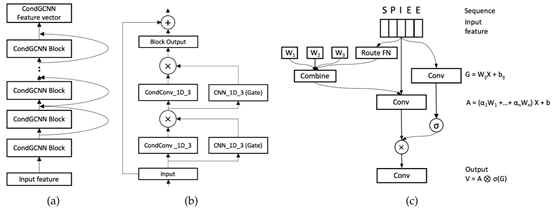
图2 CondGCNN模块及组成部分示意图
ASP模型:
本文基于ASPP模型进行改进,提出了ASP模型。由于PSSP任务中序列中每个位置上的氨基酸残基的特征都非常重要,因此本文将原始ASPP模型中的全剧平均池化去掉并直接将输入特征与卷积后的特征进行拼接,作为ASP模型的特征提取结果。
图3为一个膨胀率为2的空洞一维卷积的示例,从涂总可以看出膨胀率为2的时大小为3的卷积核与大小为5的卷积核具有相同的感受野,说明相同感受野大小的情况下,膨胀卷积需要的参数规模更小。
图3 空洞一维卷积示例
3. 结果
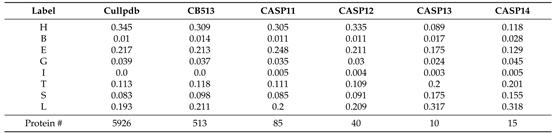
本文使用两组数据集进行实验分析。第一组数据集从CullPDB数据集中随机抽取501条序列作为验证集,其余序列作为训练集,并使用CASP11、CASP12、CASP13、CASP14和CB513数据集作为测试集,第一组数据集每种蛋白质二级结构的数量占比如表1所示。第二组测试集是SPOT-1D基准数据集,其中的10200条序列组成训练集,剩余的1000条序列作为验证集,并使用Test2016和Test2018对模型进行测试,这两个独立测试集中分别包含1213和250条蛋白质序列。
表1 第一组数据集每种蛋白质二级结构的数量占比
本文使用One-hot编码和PSSM矩阵分别对每条蛋白质虚了进行编码,其中One-hot方法将长为L的蛋白质序列编码为21*L维的特征矩阵,每个氨基酸残基由21维的向量表示,21维中包括20种标准氨基酸和非标准氨基酸的维度。PSSM矩阵包含丰富的生物进化信息,一条长为L的蛋白质序列的PSSM矩阵维度为21*L,本文使用Uniref50计算每条序列的PSSM矩阵。
本文通过将每条序列的One-hot特征矩阵与PSSM矩阵进行拼接后得到42*L维的特征矩阵,并将拼接后的矩阵作为模型的输入。
在模型评估方面,本文除了使用传统的准确率指标之外,还定义了一种蛋白质二级结构边界预测准确率指标,即只计算蛋白质二级结构边界处的预测准确率,蛋白质二级结构边界处的判定标准如图4所示,其中上方的seq为蛋白质的氨基酸序列,下方的ss为此序列对应的二级结构标签,用红色框起来的部分为蛋白质二级结构的边界处。

图4 蛋白质二级结构边界处判定标准
对每个模型组件的消融实验:
(1)探究CondCNN、GCNN、CondGCNN模型的影响
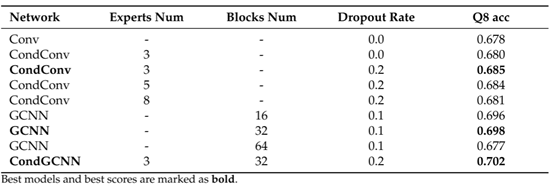
本文分别使用传统的卷积神经网络(Conv)、门控卷积神经网络(GCNN)、条件参数化卷积(CondCNN)以及将后两种模型相结合的CondGCNN模型进行实验,在CB513测试集上的实验结果如表2所示。
从表2中可以看出,当直接将Conv模型换为CondConv模型时模型预测准确率的提升幅度很小,只有0.002,本文分析这是因为CondConv模型的参数量太大所造成的,因此,本文调整了CondConv模型的Dropout率,并达到了更好的效果。其次也可以看出当使用GCNN模型时,预测准确率普遍较高,并且设置块数为32、Dropout率为0.1时,GCNN模型的预测效果最好。本文通过将CondCNN模型和GCNN模型进行结合,提出了CondGCNN模型,并在这一模型上进行实验,发现CondGCNN模型的预测准确率达到最高。
表2 不同卷积架构下模型的预测准确率
(2)探究BLSTM模型层数的影响
本文通过调整BLSTM模型的层数,探究模型层数对预测性能的影响,在CB513数据集上进行测试的实验结果如图5所示,从图5中可以看出当堆叠两层BLSTM网络时,BLSTM模型的预测准确率达到最高。
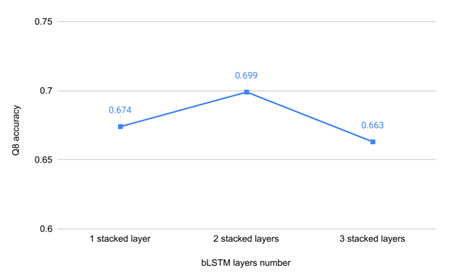
图5 不同BLSTM模型层数下模型的预测准确率变化
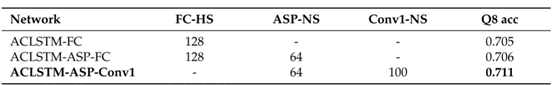
(3)探究ASP和1*1Conv的影响
本文分别基于BLSTM模型和ACLSTM模型进行实验,实验结果如表3-4所示。从两个表中可以看出,当直接在BLSTM和ACLSTM后面添加ASP层并连接全连接层时,模型性能并没有提升,但当把全连接层换成1*1卷积层后,模型的性能有了较大提升。
表3 ASP网络和1*1卷积对BLSTM模型预测结果的影响

表4 ASP网络和1*1卷积对ACLSTM模型预测结果的影响
与先进模型进行对比:
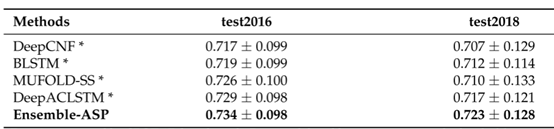
本文分别在两组数据集上与多种先进模型进行实验对比,对比结果如表5-6所示。其中Ensemble(ours)为本文模型中不添加ASP网络的模型,Ensemble-ASP 和Ensemble-ASP(ours)为本文模型中添加ASP网络的模型。从表5-6中可以看出,本文模型在所有数据集上的预测效果都优于其他先进模型,并且添加ASP网络的模型优于不添加ASP网络的模型的预测性能,体现了ASP网络的重要性。
表5 在第一组数据集上与先进模型对比结果
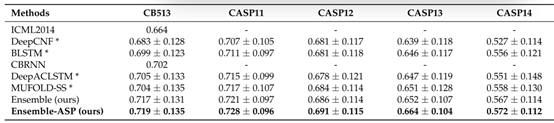
表6 在第二组数据集上与先进模型对比结果
蛋白质二级结构边界预测对比实验:
本文进行了蛋白质二级结构边界预测的对比实验,由于整个氨基酸序列中边界残基的数量较少,因此本文选择一个相对较大的测试集CB513来进行蛋白质二级结构边界预测的对比实验,实验结果如表7所示。从表7中可以看出边界预测的准确率远低于整体预测的准确率,但本文提出模型可以显著提高蛋白质二级结构边界预测的准确率。
表7 蛋白质二级结构边界预测对比实验结果

4. 结论
本文提出了一个集成编码器与空洞空间金字塔网络相结合深度学习模型(Ensemble-ASP)用于蛋白质二级结构预测。本文通过大量实验证实,所提出的模型优于最先进的蛋白质二级结构8态预测模型。此外,本文通过边界残基预测实验证实本文模型能够更好地预测氨基酸序列边界残基的二级结构类型。
本文通过使用ISS领域的模型表明,特定的网络设计可以帮助解决特定的生物信息学问题,除了开发新的深度学习算法外,学习和适应其他领域的跨学科研究思想也同样重要。此外,本文提出的模型将适用于任何蛋白质相关的预测任务,如二面角和溶剂的可及性,而非仅限于蛋白质二级结构预测任务。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


