今天为大家介绍的是来自Dan Zhao和Jianyang Zeng团队的一篇论文。目前为了克服分子特性预测中数据稀缺的挑战,人们对通过自监督学习技术预训练图神经网络(GNNs)表现出了浓厚的兴趣。然而现有的自监督学习方法面临两大障碍:缺乏明确的自监督学习策略,以及GNNs的有限学习能力。为此,作者提出KPGT模型,该模型有效地捕捉了分子的结构和语义知识。通过在63个数据集上的广泛计算测试,KPGT在预测多个领域的分子属性方面展现了卓越的性能。

近年来,基于人工智能(AI)的方法在预测分子属性方面发挥了越来越关键的作用。AI方法在分子属性预测中的一个主要挑战是如何表示分子。早期基于机器学习的方法尝试使用基本的人工特征来表示分子,这些基于这些表示的预测方法高度依赖复杂的特征工程策略,因此限制了它们的泛化能力和灵活性。近年来,深度学习方法作为预测分子属性的工具出现,主要是因为它们能够自动从简单的输入数据中提取有效特征。然而,数据集的有限性和化学空间的广阔性限制了它们的预测性能,特别是在处理分布外数据样本时。随着自监督学习方法在自然语言处理和计算机视觉领域取得显著成就,这些技术已被用于预训练图模型,以改进分子的表示学习,从而在下游的分子属性预测任务中取得了显著的效果。
当前的自监督学习方法通常涉及对分子图的修改,例如节点或子图的掩蔽,然后预测这些被掩蔽的部分,或利用对比学习目标来在潜在空间中将修改后的图与其原始图对齐。分子固有的特性与其结构紧密相连,这意味着即使是对分子图的微小修改也可能导致其语义信息的丢失。分子的这种自然特性可能限制了当前基于自监督学习的分子图方法在捕捉分子之间的结构相似性和丰富的分子属性语义信息上的能力。此外,如果没有分子图的语义信息,被掩蔽节点与其相邻节点之间的唯一联系就是共价键规则,这往往无法引导模型准确预测被掩蔽的节点。因此,这种限制可能导致模型仅仅是记忆数据集。作者提出假设,将量化描述分子特性的额外知识引入自监督学习框架可以有效解决这些挑战。分子的许多量化特征,如前述的分子描述符和指纹,可以通过当前已建立的计算工具轻松获取。将这些额外的知识整合进来可以向自监督学习引入大量有关分子的语义信息,从而显著增强语义丰富的分子表征的获取。在这项研究中,作者介绍了KPGT,这是一个旨在增强分子表示学习的自监督学习框架,进而提高下游分子属性预测任务的效果。
KPGT结构
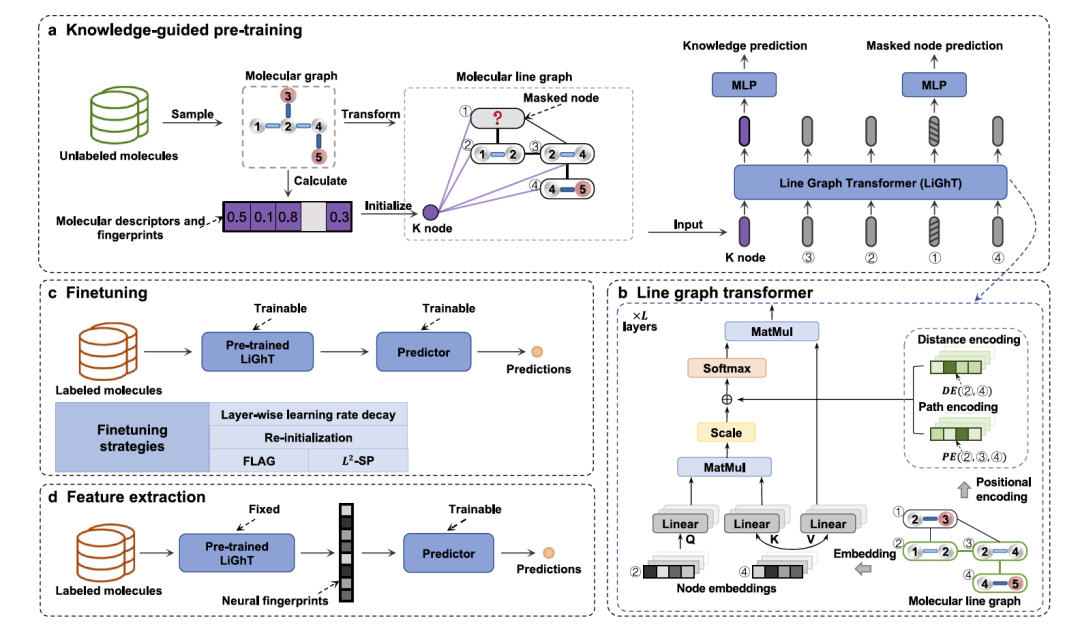
图 1
KPGT框架包括两个主要部分:一个名为线图变换器(LiGhT)的模型和一个知识引导的预训练策略。LiGhT专门设计用于全面捕捉分子图结构中的复杂模式。该模型基于经典的transformer编码器。LiGhT将分子线图作为输入,这些图表示原始分子图的边之间的邻接关系。将分子表示为线图使LiGhT能够充分利用化学键的内在特性,这在之前定义的变换器架构中通常被忽视。作者提出的知识引导预训练策略基于一个遮蔽图模型目标,先随机遮蔽分子图中的一部分节点,然后学习预测这些遮蔽节点。该策略最显著的特点是额外知识的结合。每个分子图都通过与图中原始节点相连的知识节点(K节点)进行增强。
实验部分
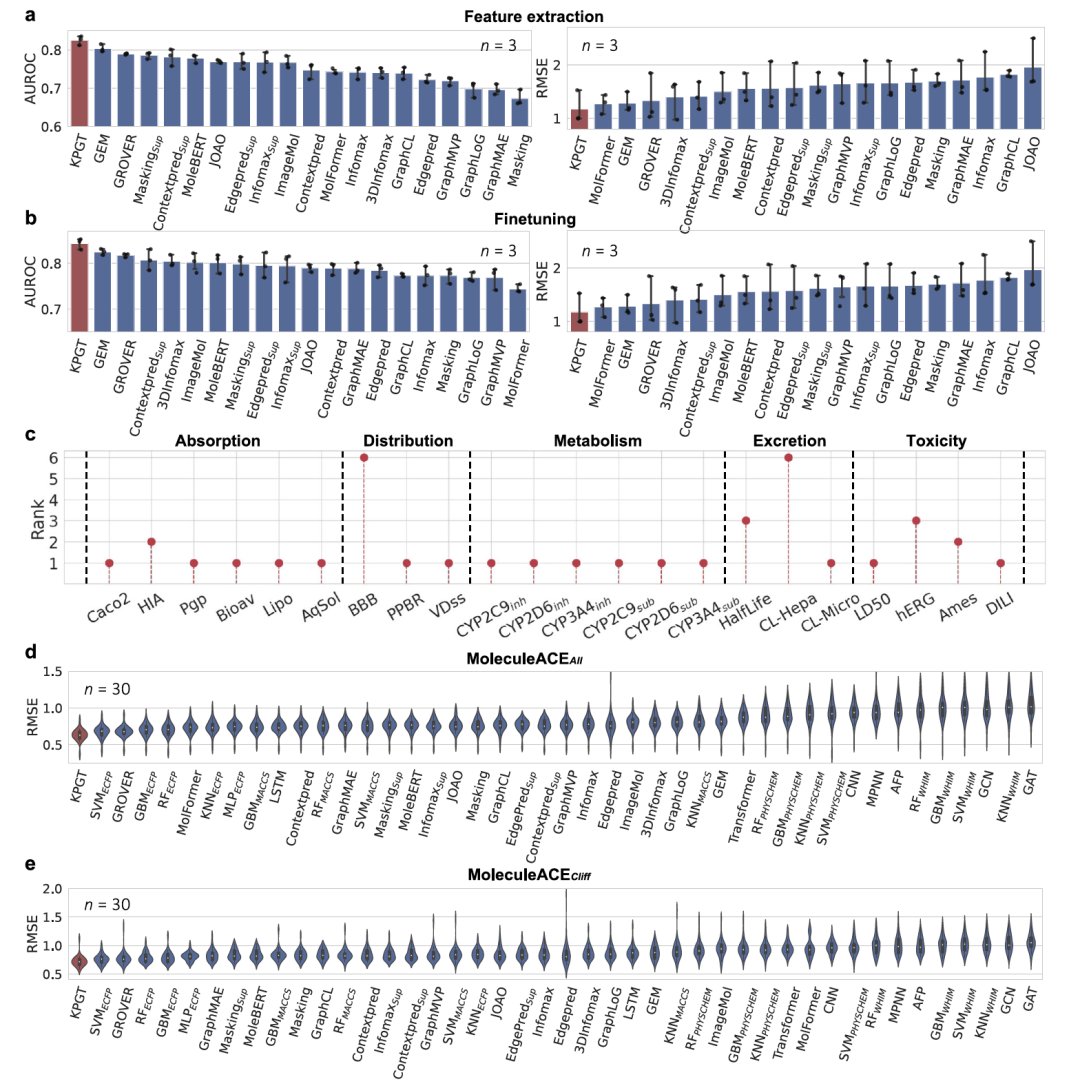
图 2
研究团队首先将KPGT与19种基于自监督学习的方法进行了比较。这次比较涉及11个分子属性数据集,其中八个设计用于分类任务,另外三个用于回归任务,覆盖了从生物物理、生理学到物理化学等多种分子属性的广泛范围。比较在两种设置下进行:特征提取和微调。在特征提取设置中,KPGT在八个分类数据集中的七个和三个回归数据集中的两个上展现了优于基线方法的性能,相对于分类任务提高了2.0%,对于回归任务提高了4.5%。在微调设置中,KPGT在八个分类数据集中的七个和所有三个回归数据集上优于基线方法,分类任务相对提高了1.6%,回归任务提高了4.2%。这些结果表明(图2),与之前的方法相比,KPGT呈现出更强大的自监督学习框架。
接下来,研究团队将KPGT与基于机器学习和监督深度学习的方法进行了比较,使用的数据集来自TDC和MoleculeACE平台。TDC是一个包含22个分子属性预测任务的基准测试平台,涵盖了从吸收、分布、代谢、排泄到毒性(ADMET)等药物发现和开发领域的关键分子属性。KPGT在与TDC排行榜上的28种基线方法比较时,表现优于基线方法的22个数据集中的16个。在预测活性崖(活性显著不同但结构高度相似的分子对)的生物活性这一更具挑战性的任务上,KPGT也表现出色。MoleculeACE提供了来自30个大分子靶标的30个涉及活性崖的生物活性数据集。在这项评估中,KPGT在MoleculeACE基准测试平台的24种基线方法中表现出色,在30个数据集中的26个上超越了基线方法,整体相对提升了3.9%。在专门针对每个测试集中的活性崖进行评估时,KPGT在30个数据集中的22个上优于基线方法,整体相对提升了1.2%。
探究KPGT模型
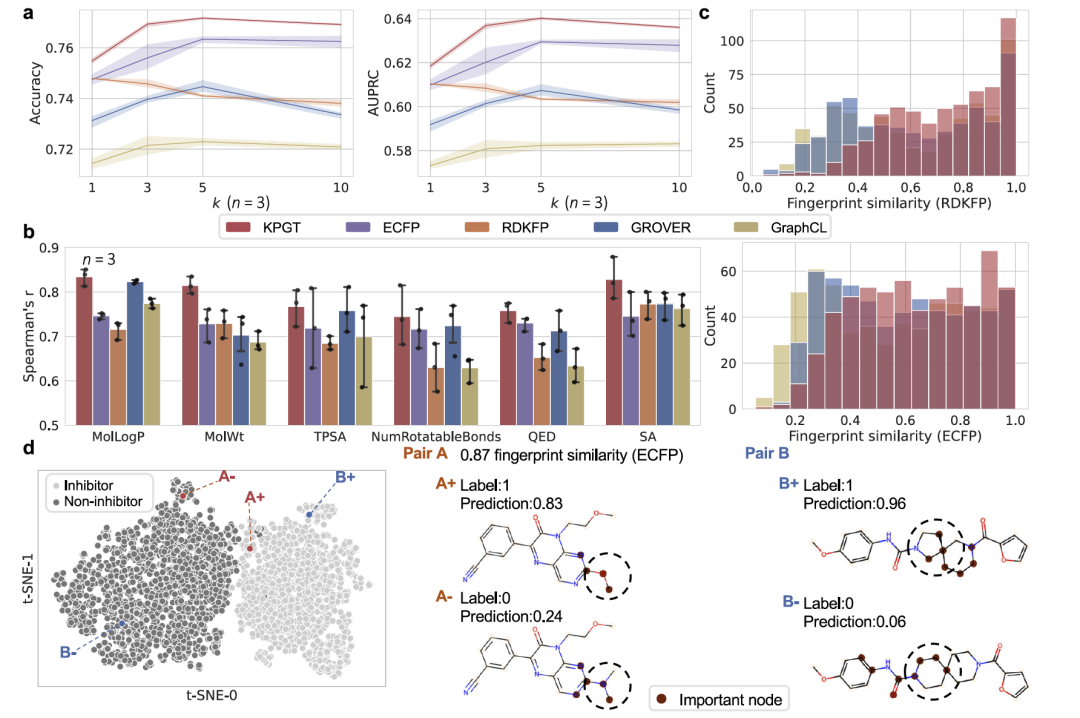
图 3
为了探究KPGT在预测分子属性方面优越性能的原因,研究团队对其预训练和微调过程中获取的知识进行了进一步调查。首先,他们分析了KPGT预训练后构建的潜在空间。他们使用了一个包含12,328个分子的数据集,这些分子针对CYP3A4酶的生物活性(即抑制或不抑制),CYP3A4在药物代谢中扮演关键角色。研究团队首先使用预训练的KPGT生成了来自CYP3A4数据集的分子的神经指纹(即分子特征表示),然后从该数据集中随机抽取200个分子作为测试集,其余分子构成训练集。接着,他们使用基于神经指纹的k近邻分类(kNN)来对测试集中的分子进行预测,并将KPGT的性能与两种经典指纹(ECFP和RDKit指纹RDKFP)以及两种来自监督学习方法(GROVER和GraphCL)的神经指纹进行了比较。KPGT在AUPRC方面相对于基线方法提高了1.3%-2.7%,这表明KPGT预训练后构建的潜在空间中的相邻分子可能倾向于展现更相似的特征(图3a)。
此外,研究团队进行了额外的测试来进一步验证他们的发现。具体来说,他们首先检索了潜在空间中每个测试集分子的最近数据点,得到200对分子。接下来,他们计算了这些抽样分子和查询分子的五个在药物发现中扮演重要角色的分子性质,包括分子LogP(MolLogP)、分子重量(MolWt)、拓扑极性表面积(TPSA)、可旋转键数量(NumRotatableBonds)、药物类似性定量估计(QED)和合成可达性(SA),然后测量了每个分子性质的抽样和查询分子对之间的相关性,结果表明,与基线方法相比,KPGT在这五个描述符上实现了更高的相关性。此外,他们通过计算它们的RDKFP/ECFP指纹之间的Tanimoto相似度来测量抽样分子和相应查询分子之间的结构相似性。结果显示,KPGT查询的分子在结构相似性方面优于基于自监督学习的基线方法。这些结果综合表明,KPGT学习到的潜在空间中,分子的邻近性不仅意味着它们结构上的相似性,而且还表示了相似的语义(图3b 图3c)。
接下来,研究团队在CYP3A4数据集上微调了KPGT,并采用-SNE技术来可视化测试集中的分子表示。结果显示,KPGT在抑制剂和非抑制剂的表示之间提供了明显的区分,表明在微调过程中,KPGT能够学习到具有不同属性分子的可区分特征(图3d)。
将模型用于药物发现
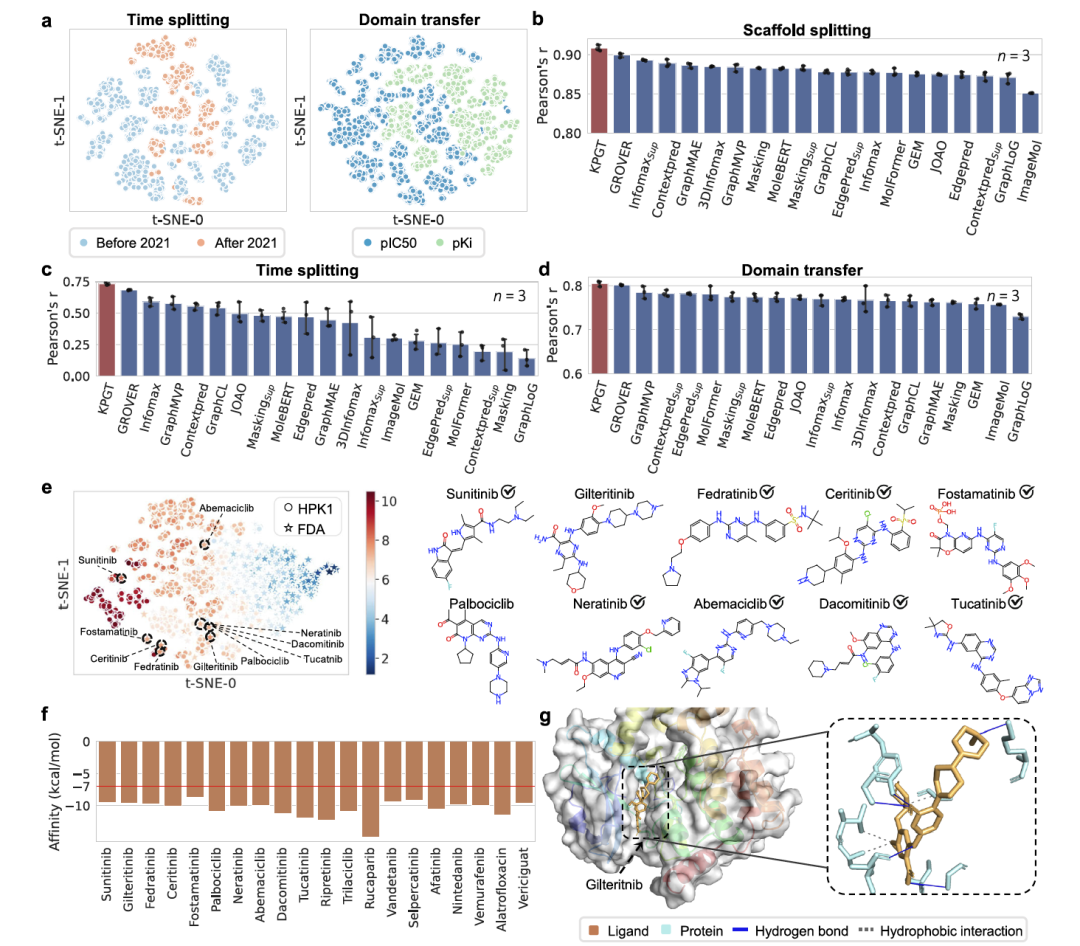
图 4
在这一部分研究中,研究团队使用KPGT来发现可能有效的抗肿瘤靶点抑制剂。造血祖细胞激酶1(HPK1)和成纤维细胞生长因子受体1(FGFR1)在多种癌症类型中具有重要作用,因此成为抗肿瘤疗法的研究重点。为了识别有效的HPK1抑制剂,研究团队收集了4442种经实验确定的HPK1抑制剂,并使用三种不同的数据划分方法对KPGT的预测性能进行了全面评估。结果显示,KPGT相关性方面显著优于19种自监督学习基线方法。值得注意的是,即使在时间分割和领域转移场景下,训练集和测试集中的分子在结构上存在显著差异,KPGT仍然保持了较高的相关性得分。这些观察结果验证了KPGT在预测HPK1抑制剂方面的卓越泛化性和可靠性(图4)。接下来,研究团队通过药物再定位来使用KPGT识别潜在的HPK1抑制剂。他们首先获取了来自DrugBank的2718种FDA批准的药物,然后对KPGT在HPK1抑制剂的pIC50数据集上进行微调,并对FDA数据集中的分子进行预测。结果显示,前20个KPGT预测中的12种药物已被之前的实验验证为HPK1的潜在抑制剂。
随后研究团队对FGFR1行了测试,这是一个与肿瘤进展和侵袭相关的有前景的药物靶标。首先,他们从专利和以往研究中收集了12,461种已有的具有实验pIC50值的FGFR1分子。在脚手架分割和时间分割的设置下评估了KPGT在FGFR1数据集上的预测性能。KPGT在脚手架分割和时间分割场景下均取得了高相关性值。接下来,研究团队对FDA数据集进行了药物再定位研究。结果显示,前20名预测的小分子中有13种在之前的研究中被实验证明是高亲和力或有效的FGFR1抑制剂。
总体而言,这些观察结果进一步强调了KPGT在加速潜在药物候选物的识别方面的能力,从而确立了它在药物发现中作为一种有价值工具的地位。
编译 | 曾全晨
审稿 | 王建民
参考资料
Li, H., Zhang, R., Min, Y. et al. A knowledge-guided pre-training framework for improving molecular representation learning. Nat Commun 14, 7568 (2023).
https://doi.org/10.1038/s41467-023-43214-1



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


