 作者提出了一种在生物医学网络中进行药物多任务预测的通用超图学习算法(A general HyperGraph learning algorithm for Drug multi-task predictions in micro-to-macro biomedical networks, HGDrug)。该模型将药物-子结构关系引入到分子相互作用网络中,集成了药物子结构和分子相互作用信息,用于药物多任务预测。
作者提出了一种在生物医学网络中进行药物多任务预测的通用超图学习算法(A general HyperGraph learning algorithm for Drug multi-task predictions in micro-to-macro biomedical networks, HGDrug)。该模型将药物-子结构关系引入到分子相互作用网络中,集成了药物子结构和分子相互作用信息,用于药物多任务预测。
HGDrug在4个基准任务(药物-药物、药物-靶点、药物-疾病和药物副作用相互作用)上实现了高度准确和稳健的预测。实验分析验证了HGDrug模型架构和多分支设置的有效性和合理性,并证明了HGDrug能够有效识别潜在的药物相关的相互作用,帮助提高其他GNN模型的性能。总之,HGDrug可以这些结论为如何在生物医学网络上引入药物的子结构信息以完成多种药物相关的相互作用任务提供了重要的见解,通过构建微观到宏观的以药物为中心的异构网络,为识别药物相关的相互作用提供了一个通用而强大的工具。
背景
大规模药物相关的相互作用网络和深度学习的强大结合,为加速药物发现过程提供了新的机会。生物医学网络包含领域知识,可用于药物发现和药物相互作用等各种任务的预测,但目前的生物医学网络忽略了对药物性质起重要作用的化学物质。以往的药物相互作用预测方法大多依赖于成对连接,而没有关注复杂生物网络中的局部强连接,无法将药物与化学结构进行关联,使得在药物性质和涉及更多节点的高阶关系中起重要作用的化学结构的数据挖掘,在当前的生物医学网络研究中尚未得到解决。
方法
HGDrug对药物进行分解,构建药物与子结构的相互作用网络,通过具有相同子结构的药物数量和子结构之间的包涵关系来挖掘药物的特征信息,捕捉药物之间的潜在关联。
为了获取药物之间的高阶信息,首先将这些异构网络和分子相互作用网络对齐,以完成特定的预测任务,并基于异构网络上的网络基序构建超图(图1(a))。作者重点研究了代表药物之间高阶关系的三角形和四边形图案(motif),并精心设计了一套图案来指导超图的构建。除了药物子结构网络,作者还建立了基于分子相互作用网络的超图,只关注这些异质网络中的三角形图案(图1(b))。作者还考虑到药物是否直接相关,并细分为:DRSS(与药物相关且具有相同亚结构)、DISS(与药物无关且具有相同亚结构)、DRSM(与药物相关且具有相同分子相互作用)、DISM(与药物无关且具有相同分子相互作用),其中每一类又包括三种图案,所以共有12种图案。图1(a)显示了所有使用的图案(M1-M12)。
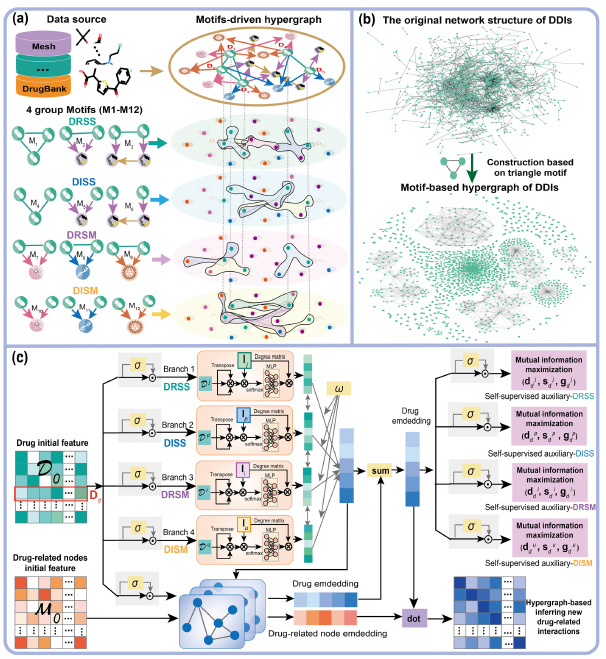 图1 HGDrug结构图
图1 HGDrug结构图
如图1(c)所示,在上述得到的四组图案驱动超图的基础上,使用四个分支提取给定异构网络的多尺度药物特征,每个分支捕获基元驱动超图中的高阶药物关系模式。为了区分不同超图中的高阶药物-药物关系对最终药物相关相互作用预测性能的影响,在药物初始特征向量矩阵D0作为每个分支的输入时,采用滤波后的自门控单元进行预处理。在非线性门通过维度重加权以特征粒度调制药物的初始特征后,获得了分支特异性药物特征。药物嵌入通过消息传播后,对每一层得到的嵌入特征进行平均以避免过度平滑。此外,由于从不同分支获得的药物特征并不是同等重要,因此采用注意力机制对从每个分支获得的药物特征信息进行聚合。对于药物多任务相互作用预测的下游任务,使用图卷积从特定下游任务的已知相互作用数据M0中获取药物和药物相关节点信息。
该模型采用贝叶斯个性化排名(BPR)损失进行优化。BPR是一种两两损失,它促进观察到的相互作用的排名高于未观察到的相互作用。设药物d为模态的输入。假设存在与药物d相关的随机正样本i和与药物d无关的随机负样本j,则模型优化的目标是使样本i在药物d的预测列表中的排名高于样本j。
不同分支可以学习不同超图上不同分布的药物特征。为了避免聚合操作可能导致的高阶信息丢失,采用自监督辅助任务来提高关联预测任务的性能。受深度图信息(Deep Graph Infomax, DGI)方法的启发,模型计算节点表示和全图表示之间的互信息(mutual information)。在自监督辅助任务中,通过层次化地最大化药物表征与对应的以药物为中心的子超图以及各分支的超图之间的互信息,可以得到反映不同超图中药物节点局部和全局高阶连接模式的综合药物表征。对于每个分支的超图,构建邻接矩阵来捕获药物的高阶关联信息,每一行通过相应超图的行索引表示一个以药物为中心的子超图。子超图表示可以通过一个读出函数得到。这样得到的药物嵌入与药物相关节点嵌入进行点乘,得到基于超图的药物相关的互作关系。
结果
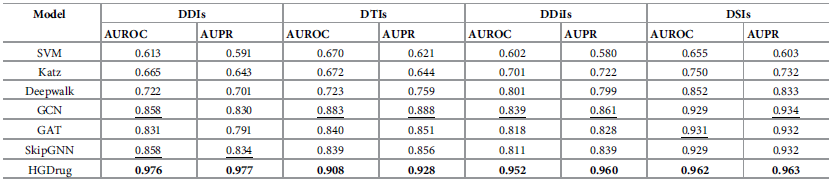
作者将HGDrug与一些具有代表性的方法进行了比较。在药物-药物相互作用(DDI),药物-靶标相互作用(DTI),药物-疾病相互作用(DDiI),药物-副作用相互作用(DSI)这四种任务上,比较各个方法的AUROC和AUPR。如表1所示,HGDrug在所有四个任务的AUROC和AUPR上都取得了最佳结果(加粗数字),且显著高于第二名(下划线数字)。
表1 与其他方法对比
作者设计了模型消融实验。实现了HGDrug的5个简化变体,以检验HGDrug中每个成分在4种不同类型的预测任务中的作用。结果如图2所示。HGDrug的5个简化变体记为:(1)HGDrug_0:移除第1 ~ 4个超图分支。(2)HGDrug_2f:去除第3和第4个分子相互作用超图分支,保留第1和第2个药物亚结构超图分支。(3)HGDrug_2m:去除第1和第2药物亚结构超图分支,保留第3和第4分子相互作用超图分支。(4)HGDrug_ac:通过超图卷积网络获得药物表示。(5)HGDrug_self:移除自监督辅助任务。
图2显示了单独使用药物-子结构超图分支和分子相互作用超图分支所获得的差异很小,这证明了药物-子结构网络捕获的药物信息可以与药物相关相互作用网络获得的药物特征信息相媲美。对于已知相互作用较少的DDiI任务,是否利用所有超图分支来获取特征(HGDrug_ac, HGDrug_self, HGDrug) (HGDrug_0, HGDrug_2m, HGDrug_2f)可以观察到很大的差异。它说明了在已知相互作用不足时获得更可靠的侧信息对分子相互作用关联预测的重要性。HGDrug采用超图关注网络架构来关注超图中相邻特征的差异,并使用自监督辅助任务来权衡不同类型的高阶信息在交互预测中的贡献。因此,HGDrug可以从超图中学习到更高质量的药物特征信息,优于通过超图卷积网络获得药物表示的HGDrug_ac和去除了自监督辅助任务的HGDrug_self。这些研究还表明,HGDrug可以捕获额外的特征信息,以增强药物相关相互作用的预测,这可能为理解药物、药物亚结构和分子之间的相互作用机制提供新的见解。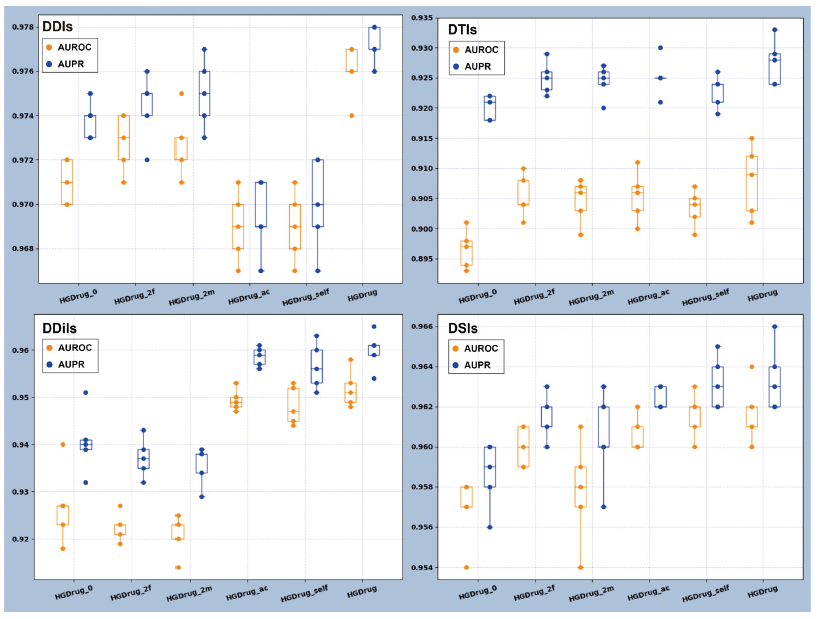 图2 消融实验作者还进行了案例分析。删除了预测模型中使用的已知DDI,并保留了DDI任务中预测的前20个新交互。表3显示了DrugBank数据库中记录的前20个新的预测DDI和显示两种药物之间关联的信息。由表2可以看出,在前20个新的预测DDI中,有18个可以在DrugBank的药物相互作用记录中找到(标签为1)。表2 案例分析
图2 消融实验作者还进行了案例分析。删除了预测模型中使用的已知DDI,并保留了DDI任务中预测的前20个新交互。表3显示了DrugBank数据库中记录的前20个新的预测DDI和显示两种药物之间关联的信息。由表2可以看出,在前20个新的预测DDI中,有18个可以在DrugBank的药物相互作用记录中找到(标签为1)。表2 案例分析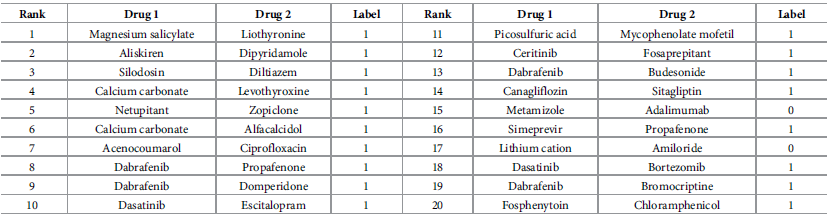
结果表明,HGDrug模型可以有效识别潜在的新型相互作用。这表明,即使缺乏标签信息,从超图分支中学习到的表示也遵循某种与药物类别密切相关的模式。
总结
在这项工作中,作者提出了以药物为中心的异构网络方法,并开发了一个称为HGDrug的通用框架,这是一个用于预测药物相关相互作用的新型超图注意力网络。
HGDrug可以作为预测药物相关相互作用的有效方法,为药物相关的相互作用的计算提供新的思路,并为药物发现提供计算机辅助指导。基于超图的方法不受网络数据大小的限制。它利用现有的网络数据提取药物之间的强相关性,从而提高药物相关任务的预测准确性。这种方法允许将计算扩展到更大的网络数据集,而不会增加计算复杂性。此外,HGDrug擅长处理大多数与药物相关的交互任务预测,显著减少了计算资源的消耗,使相关任务的执行更加方便和高效。
参考文献
[1] Jin et al. A general hypergraph learning algorithm for drug multi-task predictions in micro-to-macro biomedical networks. PLoS Comput Biol. 2023
——— End ———



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


