近日, Protein Science期刊在线发表了上海交通大学生命科学技术学院魏冬青教授与熊毅副研究员团队题为TEPCAM: prediction of T cell receptor-epitope binding specificity via interpretable deep learning的研究论文。上海交通大学硕士研究生陈俊炜是论文的第一作者。上海交通大学熊毅副研究员和中南大学李敏教授是论文共同通讯作者。

介绍
T细胞在人体免疫系统中起着至关重要的作用。其表面的T细胞受体(TCR)能够识别由主要组织相容性复合体(MHC)呈递的特异性抗原表位(epitope),该过程是触发下游免疫应答的关键。探究TCR-epitope的结合模式对开发新抗原疫苗、药物等免疫疗法至关重要。通过深度学习方法准确预测TCR-epitope的结合特异性仍然具有挑战性,特别是在待预测的新序列未出现在训练集等不可见的外推情景下。为了提高模型的泛化性和可解释性,作者提出了一种融合了自注意、交叉注意机制和多通道卷积的深度学习模型TEPCAM。结果表明,该模型在两个具有挑战性的任务(数据集严格分割的Zero-shot和外部数据集的Zero-shot任务)上优于几个最先进的模型。此外,通过交叉注意力模块提取可解释性矩阵,并将其映射到相应三维结构中,证明了该模型可以学习TCR与表位之间的相互作用模式。
模型与方法
作者提出了名为TEPCAM的TCR-epitope的结合特异性预测框架。TEPCAM主要包含四个模块:序列编码模块、注意力模块、卷积模块和前馈神经网络模块。
-
序列编码模块:首先,TCR序列在输入编码模块前使用IMGT编号进行对齐,考虑到不同氨基酸在序列不同位置出现的含义不同,因此在应用Embedding层编码每个氨基酸时额外加入位置编码信息。
-
注意力模块:自注意力模块的作用是进一步处理Embedding后的序列向量,而交叉注意力层则是TCR与Epitope的核心信息交换模块。多头注意机制具有捕捉长程依赖关系的能力,在交叉注意力模块中,Q(Query)与K (key)-V (value)对来自于两个不同实体,从而捕获它们之间的相互作用。从自注意力层提取的注意力分数可以解释为每个位置对整个模型的贡献。交叉注意力层得到的矩阵则更直接地反映了TCR与epitope各位置之间的相互作用。
-
卷积模块:从注意力矩阵与像素图的可类比性出发,交叉注意力模块的输出矩阵使用卷积模块处理,该模块由三层卷积神经网络组成,输出通道数逐渐增加,使用批量归一化处理每层的输出。
-
前馈神经网络模块:卷积模块的输出与交叉注意力模块的输出(残差连接)合并后输入由三层全连接层组成的前馈神经网络。该模块使用GeLU作为激活函数,最终使用Softmax将输出表征为TCR与Epitope相互识别的概率。
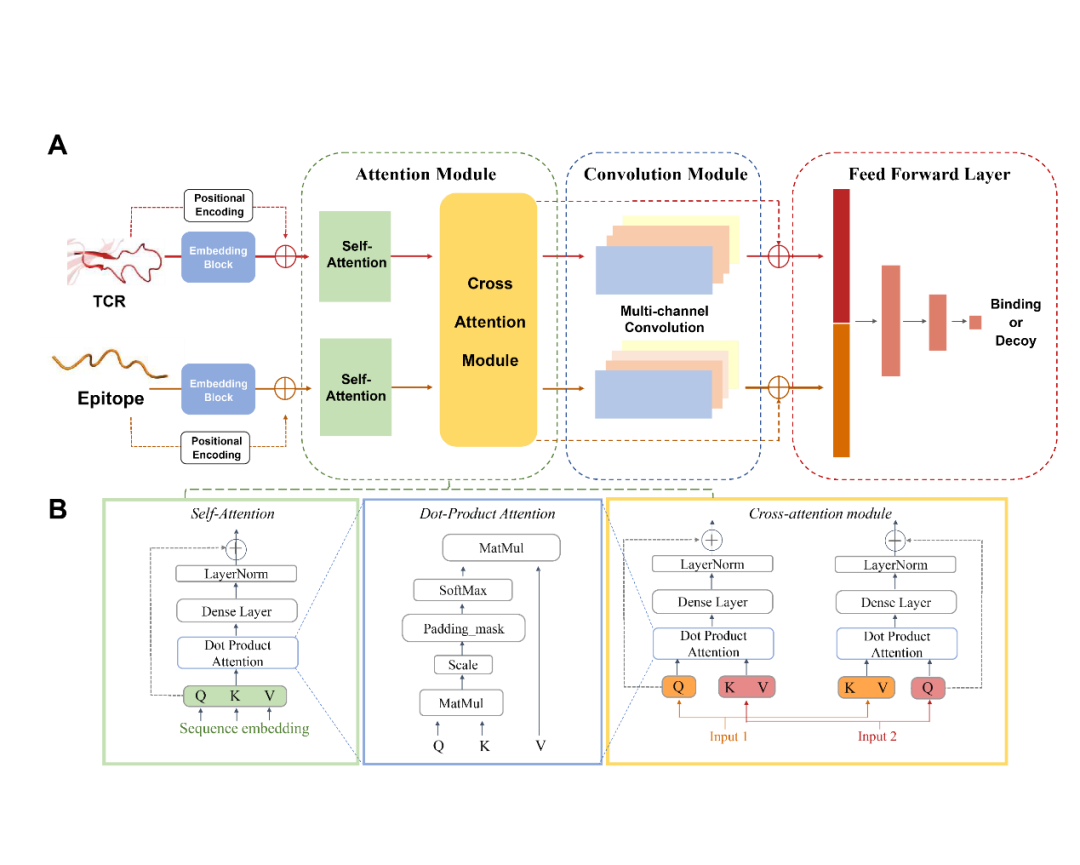
图1. TEPCAM模型总体框架图
结果
TEPCAM在两个TCR Zero-shot任务中的表现优于多个现有模型
作者清洗并整合了VDJdb, McPAS和IEDB三个公共数据集的数据,为了避免负样本生成过程中的外部偏差,采用随机Shuffle正样本对的策略来生成平衡的负样本,得到了一个包含13万个样本对的数据集TEP-Merge。为了验证模型的泛化性,首先在TEP-Merge上进行TCR端的严格分割,将TEPCAM与其他四个TCR-epitope结合特异性预测模型进行了比较。四个基线模型均为基于有监督方式训练的深度学习方法,分别为TITAN、ERGO-AE、ERGO-LSTM和ATM-TCR。训练数据和测试数据按4:1的比例严格分割数据集。为了尽量减少随机性的影响,在数据分割和模型训练时均使用了不同的随机种子。TEPCAM比其他四种方法表现出更好的性能(图2A)。
接下来,作者将TEPCAM应用于外部数据集ImmuneCODE,以测试模型在涉及另一个可能具有不同分布的数据集时的性能。经过同样的过滤方法,最终的ImmuneCODE数据集包含28303个TCR-epitope对,分配给大约1000个COVID-19相关个体,其中TEP-Merge中出现的TCR均被删除。TEPCAM在该外部数据集的所有指标优于其他模型(图2B)。值得注意的是,与同一个数据集内部分割的任务相比,外部测试集更具挑战性,因为外部数据集的分布可能不同,显著的分布变化会使模型在不可见数据上的泛化变得更加困难。总之,实验结果表明,与其他最近发表的框架相比,TEPCAM可以更好地泛化到未知的TCR序列。
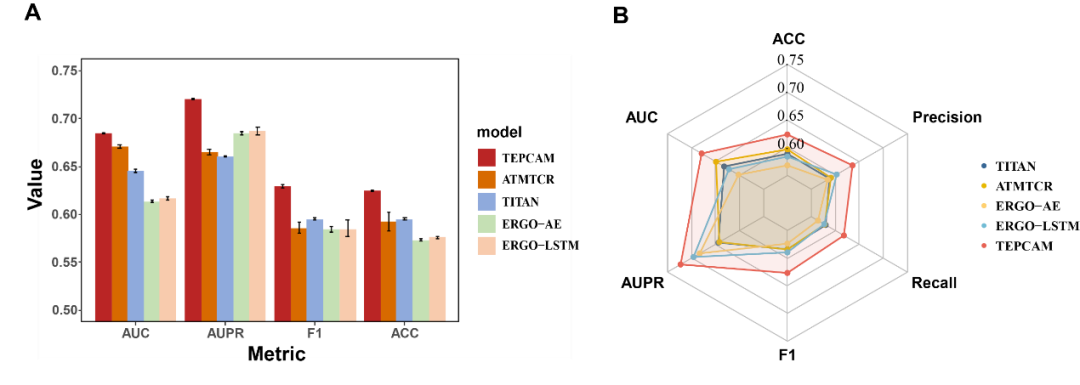
图2. 模型性能的对比
模型的消融分析和全面测试
在该部分,作者进一步评估了整个模型中各个模块的预测能力。一系列消融分析的实验结果表明:当单独去除自注意力模块、交叉注意力模块或卷积模块时,TEPCAM模型的性能都有所下降,但相应指标仍然高于基线模型。这可能是由于嵌入模块后的框架虽然去掉了一部分特征,但仍然可以从其他模块中提取出有用的特征进行预测。然后,通过模块次序交换将交叉注意力模块定位在卷积模块之后,这种重新排列也会导致模型的性能下降。
作者还测试了不同的Embedding策略的效果,包括BLOSUM62矩阵和两个最新发布的预训练框架TCR-Bert和TCR2Vec。TCR-BERT和TCR2Vec将TCR序列进行编码,输出为包含丰富进化信息的高维向量,下游任务性能比TAPE和ESM系列等一般蛋白质语言模型更好。然而,TCR-BERT和TCR2Vec在在该框架中的引入并未使模型表现提高 (图3A)。
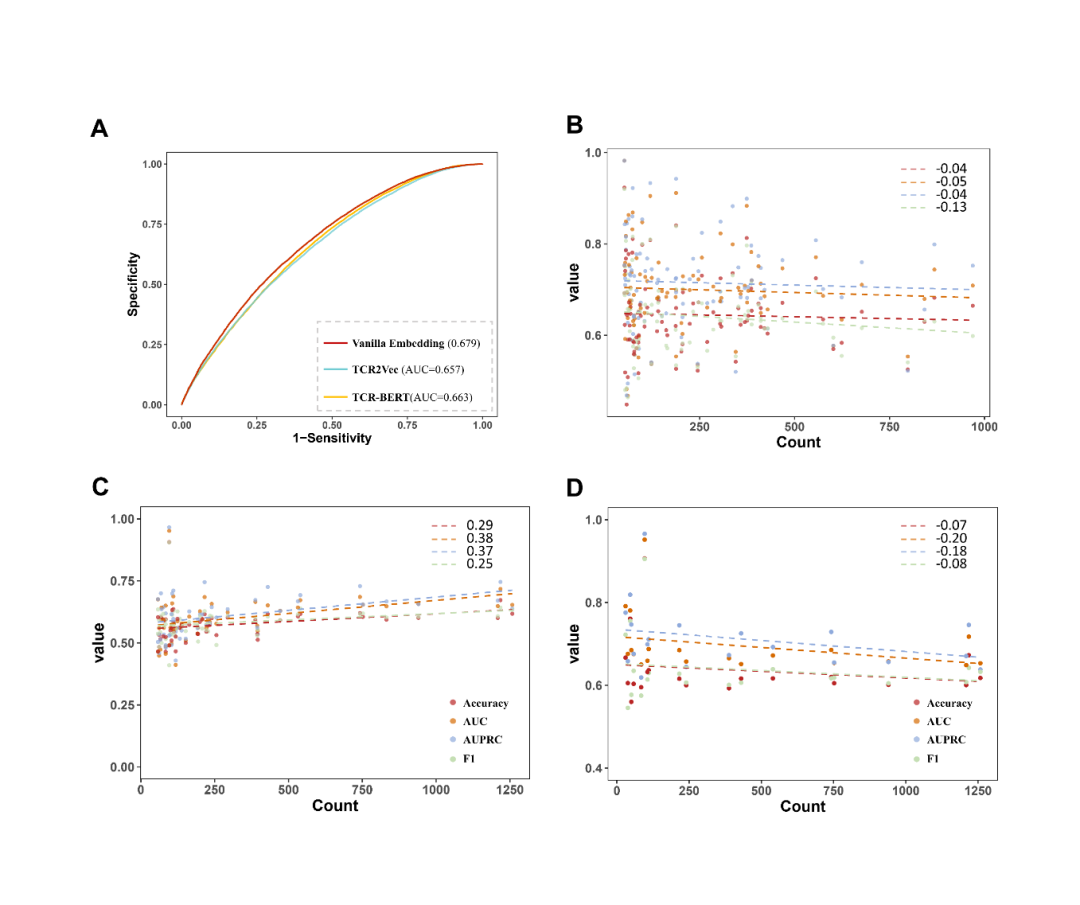
图3. 模型性能分析
深度学习模型有时倾向于在数量较多的样本上获得更好的性能。对于TCR-epitope相关数据集,TCR的多样性远远大于表位,并且表位的分布严重不平衡。对于在TEP-Merge进行交叉验证的任务,表位的数目与评价指标呈现非常弱的负相关,这表明此模型对该数据集中的表位分布不敏感(图3B)。在ImmuneCODE测试集上,观察到指标与计数的正相关。然而,当进一步搜索AUC预测性能最好的前20个表位时,表位计数和指标又呈现出负相关,说明对于预测较准确的表位,数目不影响模型性能。(图3C-D)。这些发现支持模型没有受到训练数据内部偏差的太大影响,提示TEPCAM模型可以学习到一些TCR-epitope结合模式。
以AUROC为指标评价模型在单个表位上的性能,即计算每个模型中获得AUROC排名第1的表位个数。TEPCAM在两个任务上都取得了最高的Top 1 AUROC (图4A)。其次,根据表位在训练集中的数量选出数目最多的20个与最少的20个,比较TEPCAM和ATM-TCR(4个基线模型中表现最好的)的AUROC。TEPCAM在大多数表位上表现更好,特别是在一些ATMTCR几乎无法预测的表位下,TEPCAM获得了较好的AUROC(图4)。
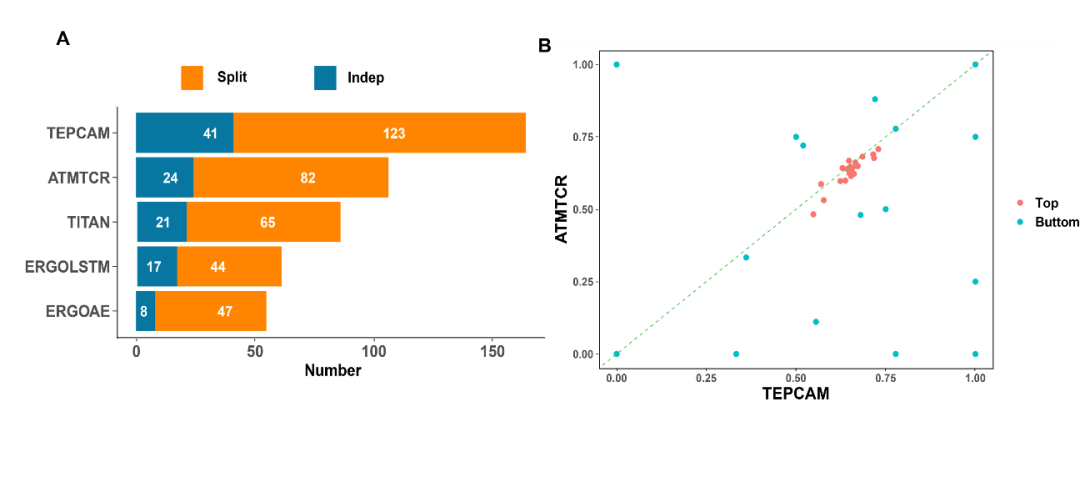
图4. 每个表位上AUROC的比较
注意力分数提示了TCR与表位的结合模式
TEPCAM在模型可解释性方面具有优势,可以在残基水平上更仔细地观察相互作用模式,这对于解析特定的生物规则至关重要。位置间的交互强度可以用注意力分数来表示,作者从ImmuneCODE测试数据集中提取了自注意力层和交叉注意力层的注意力分数,验证模型是否学习到特定的相互作用模式。结果显示,TCR序列中间部分的注意力分数较高 (图5A)。汇总所有样本的结果,从交叉注意力中提取的二维注意力矩阵揭示了TCR与表位之间相互作用模式的更精细的信息 (图5B),可以为具体案例提供更多的生物学见解。
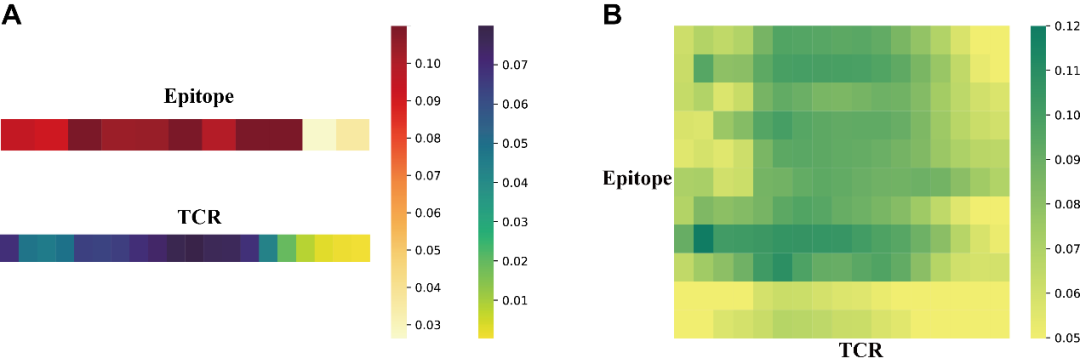
图5. TEPCAM提取的注意力分数
在案例分析部分,作者从STCRdab中取了PDB ID分别为2BNQ和5EU6的TCR-pMHC复合物。2BNQ:位于结合表面的表位上的甘氨酸残基(G*5)位于TCRβ CDR3区的G*99-T*101附近。在交叉注意力矩阵中,G*5 ~ G*99和G*5 ~ G*100对应的注意分数最高(图5A),表明模型捕获了这些有可能决定TCR与表位结合的残基对。5EU6:位于表位第5位的色氨酸残基(W*5)比其他位置的注意力分数更高。从晶体结构上可以看出,色氨酸残基中的芳香环使该位置与TCR序列中几个残基的距离更近。芳香环与酪氨酸(Y94)、缬氨酸(V95)和甘氨酸(G96)的距离在5Å之间。三个相互作用残基对中的两个被TEPCAM成功捕获。这些案例表明TEPCAM成功学习到一些关于潜在结合机制的知识。
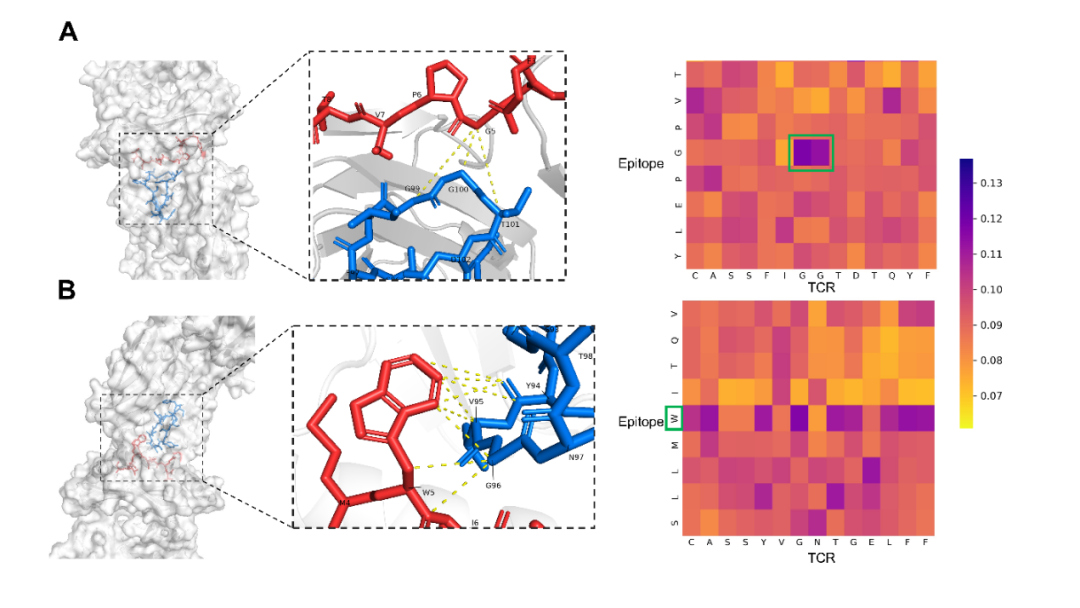
图6. 案例分析。(A) 2BNQ;(B) 5EU6。
总结
该工作提出了一种融合了自注意力、交叉注意力和多通道卷积的模型TEPCAM,在TCR端的Zero-shot任务上优于现有模型,TEPCAM能够学习更多的TCR-表位识别模式。此外,交叉注意力层提取的注意力矩阵增加了模型的可解释性,证明了该模型识别TCR和表位之间关键结合区域的有效性。
参考资料
Chen, J. et al. TEPCAM : prediction of T cell receptor‐epitope binding specificity via interpretable deep learning. Protein Science. 2024; 33(1):e4841.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


