今天介绍一篇2023年11月发表在《Briefings in Bioinformatics》期刊上的论文,题为“From Intuition to AI: Evolution of Small Molecule Representations in Drug Discovery”,文章的第一作者为英国爱丁堡大学的Miles McGibbon研究员和 Steven Shave研究员,以及中南大学的董界副教授,通讯作者为爱丁堡大学的Vincent Blay博士。该综述总结了药物发现领域中分子表示(表征)的演变历程,从最初的人类可读格式,逐步发展到现代的数字描述符、指纹,以及基于序列和图的学习表示。作者强调了各种表示方法在通用性、计算成本、不可逆性和可解释性等方面的优缺点。文章还讨论了药物发现领域的创新机会,包括为高价值、低数据制度创建分子表示,提炼更广泛的生物和化学知识成为新颖的学习表示,以及对新兴治疗方式进行建模。总体而言,文章聚焦于数字化分子表示在药物研发中的关键作用,同时探讨了所面临的挑战和机遇。

摘要
在药物发现领域,人工智能科学家和化学信息学家的目标是帮助识别分子起始点,从而开发出安全有效的药物,同时降低成本、时间和失败率。为了实现这一目标,以数字格式表示分子至关重要,这种格式使它们具有机器可读性,并有助于准确预测驱动决策的特性。多年来,分子表示已经从直观和人类可读的格式发展到定制的数字描述符和指纹,现在又发展到在广阔的化学空间中捕捉模式和显著特征的学习表示。其中,基于序列和基于图的小分子表示已变得非常流行。然而,每种方法在通用性、计算成本、生成式应用的不可逆性和可解释性等各个方面都有优缺点,这些在指导从业者决策方面可能至关重要。随着药物发现领域的发展,创新机会不断涌现。其中包括为高价值、低数据制度创建分子表示,将更广泛的生物和化学知识提炼成新的学习表示,以及对新兴治疗方式进行建模。
小分子的数字化表征
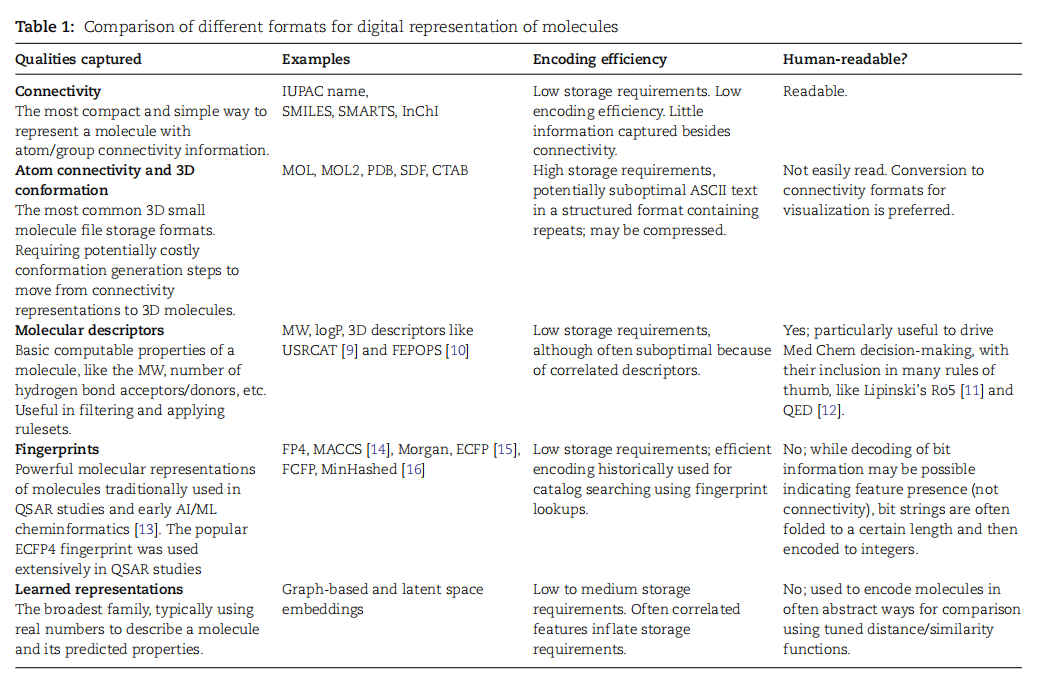
在分子表示的演变历程中,最初使用通用名称对分子进行命名,但随着化学领域的发展,1919年IUPAC++的成立标志着对化学命名法和术语的规范化。然而,这种命名法繁琐难懂,推动了更适合计算机的数字表示的发展,其中包括基于分子图的SMILES格式和3D分子格式。SMILES通过字符串简洁而可读地表示分子,但同一分子可有多个等效的SMILES表示,引发了其他格式的发展,如SMARTS、SMIRKS、SELFIES等。此外,IUPAC创建的InChI格式提供了机器可读的分子表示,包含电荷、立体化学和同位素信息。另一方面, 3D分子格式如MOL和MOL2包含了原子在三维空间中的位置,适用于包含更多信息的领域,如晶体学和分子动力学。随着计算药物发现领域的成熟,人们开始探索将分子输入数学模型的方法(表1),包括手工设计的分子描述符和指纹,以及近年来兴起的基于深度学习的学习表示。目前,SMILES和图形编码已成为神经网络模型中常用的小分子表示方法。然而,随着制药领域的不断发展,仍有许多改进和创新的空间。
表1.不同形式的分子数字表示比较
定制化表征
分子描述符是使用预定义规则计算的小分子的数值表示,旨在建立化学结构与性质之间的定量关系。发展历程自20世纪60年代始,最初采用物理化学描述符,如分子量和logP,以及拓扑描述符,为药物化学规则集的形成做出贡献。随着时间推移,出现了更复杂的E态电描述符和拓扑自相关描述符,全面捕捉分子结构和物理化学性质。计算机技术的进步推动了更多描述符的提出,包括原子对描述符和分子静电势。到2000年,已有数千种分子描述符,为化学领域提供了丰富的工具。
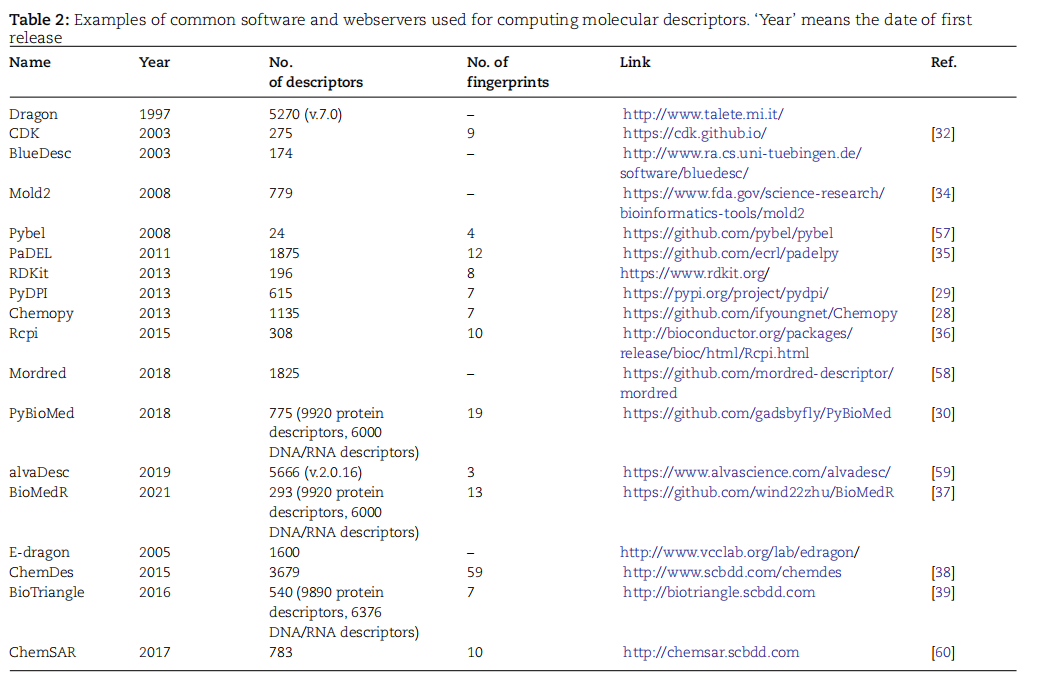
分子描述符的计算主要依赖于专业软件包。这些软件包通常源自三种流行的编程语言之一:C++、Java和R。RDKit是基于C++的流行包,提供Python接口,可计算208个描述符和5个指纹。CDK是用Java开发的另一种软件,可计算275个描述符和9个指纹图谱。以R编程语言为基础的Rcpi可生成300多个小分子描述符。这些软件被广泛应用,还有一些扩展软件如Chemopy、PyDPI和PyBioMed。此外,商业软件如Alvascience alvaDesc、CCG MOE、BIOVIA Discovery Studio等也提供描述符生成功能。网络服务器和数据库如E-Dragon、ChemDes和BioTriangle也是获取描述符的途径。这些描述符的计算对于理解分子结构与性质关系,尤其在药物发现领域,具有重要作用。
表2.用于计算分子描述符的常用软件和网络服务器示例。
在实践中,不同工具各有优势和劣势。商业软件提供高稳定性但需购买许可,独立软件包灵活但对特定环境依赖,而网络服务器无需编程知识但计算过程不透明。开源软件和FAIR原则的采用显示了对过程完全控制的需求。
分子描述符在研究化合物相互作用和药物设计中发挥关键作用。通过“分子相似性原理”,它们检索潜在活性物质,进一步进行命中扩展、SAR探索和骨架跳跃,为替换核心骨架、获取新的化学结构、改变药代动力学、毒性概况和化学空间提供支持。同时,它们用于数据库检索、QSAR、分子对接和结构/药效图可视化,用于理性药物设计。指纹可视为描述符的子类型,其计算快速、信息丰富,可用于加速建模。虽然存在成千上万的描述符,但指纹作为即插即用的选择,通过固定大小的向量编码分子,在描述符选择方面更为便捷。
量子机器学习(QML)模型在预测化学性质方面备受青睐,其速度不断提高且能从第一性原理生成详细数据。基于性质的模型如AlphaQ使用理论计算分子静电势的三维分布,而Schütt等采用深度神经网络构建化学环境表示,预测HF能量、电荷布居等分子性质。与基于性质和波函数的模型不同,一些delta-QML方法如DelFTa使用计算成本较低的GFN3-xTB替代昂贵的DFT计算,实现了高速度和DFT精度。这些方法有望在药物发现中开辟新的分子表示和性质建模可能性,特别是在早期捕获静电和场信息以更准确地编码分子特性方面。
习得表征
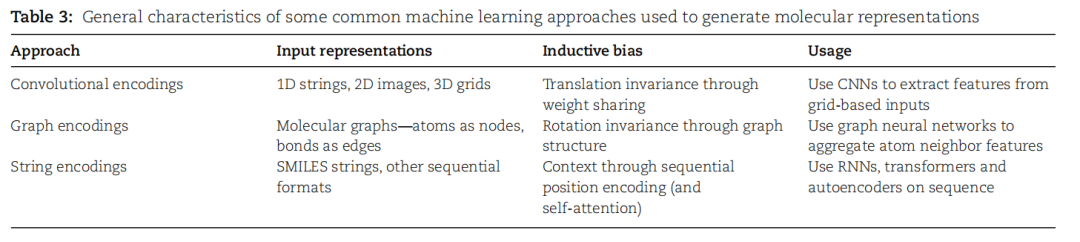
机器学习在药物发现领域的应用迅速增长,涵盖了基于结构的虚拟筛选、分子性质预测和分子相似性等多个方面。深度学习方法致力于将复杂的分子映射为适用于特定任务的丰富嵌入,与传统的分子描述符或指纹不同,习得的嵌入可能是无损的,能够提取母体分子中的任何信息,而不仅仅是预定义的特征,更适用于下游预测任务中推断所需属性。表3总结了一些讨论的体系结构。这些算法具有不同的归纳偏差,而图和字符串编码结合图神经网络和transformer体系结构变得非常受欢迎。为缓解数据需求,预训练的嵌入器可用于任何编码方法,通过在大规模未标记的分子数据上进行预训练,学得可泛化的表示,有助于在微调下游任务时缓解过拟合问题,特别是在标记数据稀缺的情况下。
作者在该节中详细讨论了卷积编码,图形编码(表4),字符串编码,预训练嵌入器(表5),迁移学习以及大型语言模型等体系结构。
表3. 用于生成分子表示的一些常见机器学习方法的一般特征。
卷积编码
卷积神经网络(CNNs)因在图像识别中的成功而广受赞誉,LeNet是早期高性能架构之一。在小分子领域,CNN应用于分子图像表示。通过卷积,模型可高效提取分子中的高级特征。卷积嵌入广泛应用于不同分子表示形式,如SMILES字符串的独热编码、基于体素网格的电子密度表示以及3D点云表示。然而,基于CNN的嵌入存在一些局限性,如缺乏旋转不变性,可能导致对同一分子的不同预测。为解决这问题,一些方法采用数据增强,对同一训练分子进行多次旋转或枚举有效SMILES字符串。然而,这增加了训练时间和计算成本。因此,人们更倾向于使用图编码进行分子表示,而等变神经网络是一个有前途的研究方向,它纳入欧几里得对称性,克服了对数据增强的需求。
图形编码
在几何或基于图的深度学习中,分子的表示采用图结构,其中原子被视为图中的节点,而原子之间的键则表示为图中的边。这些节点和边缘具有相关的特征,节点特征通常描述原子的性质(如化学元素和芳香性),而边缘特征表示键的类型(如单键、双键和三键)。整个分子因此被表达为一个图,而图神经网络能够提供几乎完美的旋转不变表示,适用于深度学习方法。这种几何深度学习在分子性质预测方面得到广泛应用,涵盖了溶解度、毒性和口服生物利用度等多个方面。
表4.将小分子编码为分子图时常用的节点和边缘特征列表
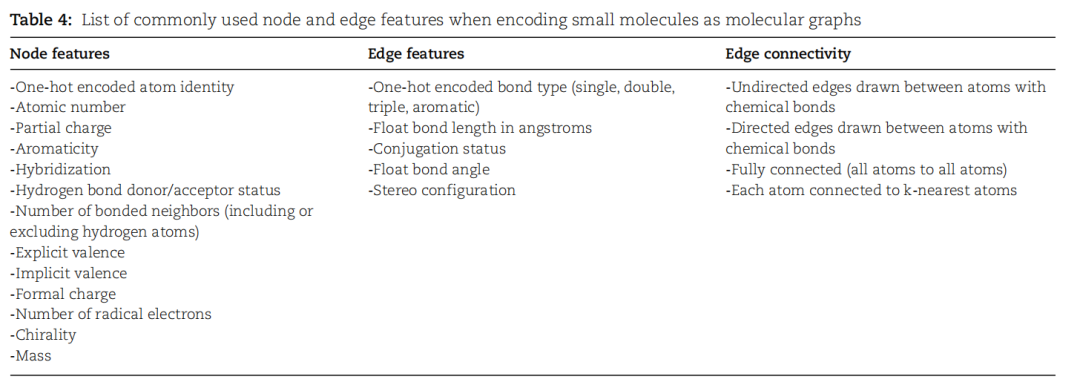
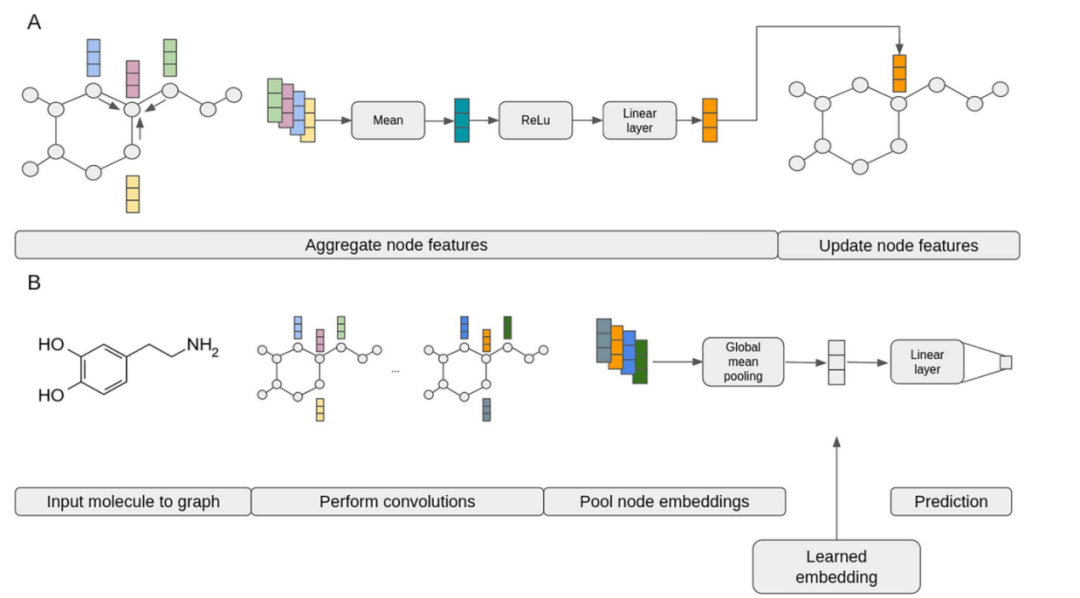
图1. 作为神经网络模型一部分的小分子图上的卷积和池化的图示。(A)在单个节点上执行的单个卷积更新节点特征的详细示意图。(B) 全图嵌入的高级视图,包括分子图、多个卷积层、全局池化和用于预测的线性层。
字符串编码
小分子的字符串编码通常包括两个步骤。首先,将小分子的字符串表示转换为数字表示,通常是整数标记或2D二进制独热编码矩阵。使用SMILES字符串来表示小分子在深度神经网络中的能力被证明在捕捉分子图方面具有优势。通过技术如独热编码,Gómez-Bombarelli等人使用自动编码器深度神经网络架构将小分子的SMILES嵌入潜在空间,实现了在连续化学空间上的生成方法。尽管这种表示相对于操作分子图像而言具有更低的噪声和效率,但潜在空间中存在的问题包括生成无效SMILES字符串,其中包含未闭合括号和不正确的键序。为了解决这个问题,出现了基于线的额外表示,如DeepSMILES和SELFIES,以减轻学习SMILES语法细节的需求。Transformer架构最近在字符串编码和解码任务中展现出先进性能,对于多种序列数据建模方面变得越来越受欢迎,包括小分子字符串、蛋白质序列和DNA序列。
预训练嵌入器
最近,为了克服小数据集和复杂模型参数的挑战,人们在分子表示中越来越多地采用预训练嵌入器。这些嵌入器的无监督训练使其能够在潜在空间中生成丰富的初始嵌入,全面捕获分子的表示,而不受过度拟合于具体化学空间的限制。预训练的嵌入器通常通过在潜在空间中编码输入分子进行训练,然后将其解码回实际输入。这种方法为模型提供更多数据,有望产生强大且具有推广性的新输入分子嵌入。在无监督学习中,常见的架构包括自动编码器和转换器。这种趋势的例子包括Chemformer和用于虚拟筛选抗SARS-CoV-2分子的高性能分子编码器。
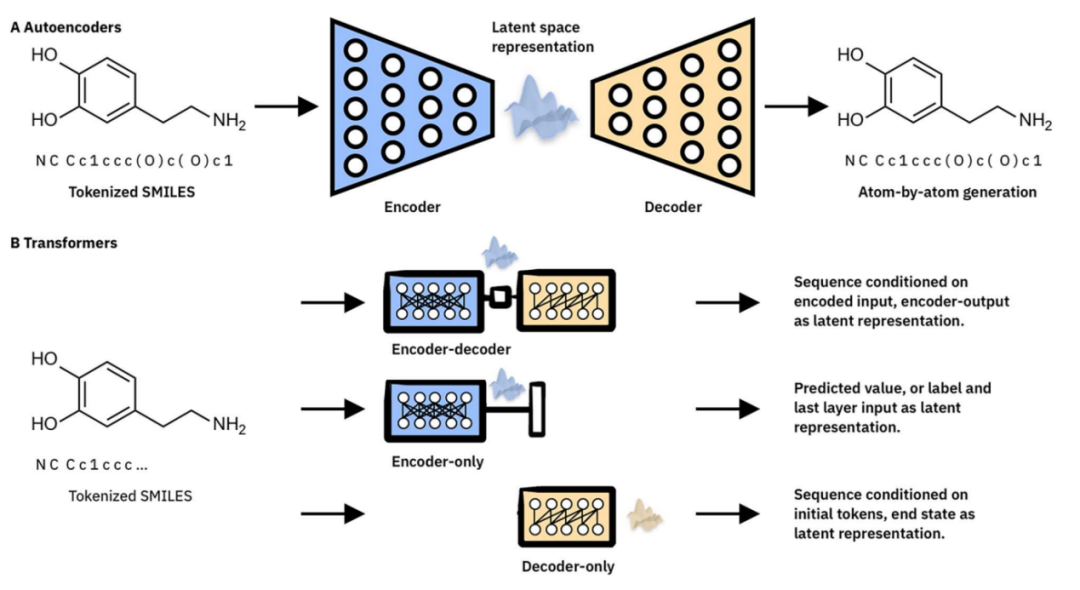
图2. 用于分子表示的无监督学习的机器学习架构的高层次比较。(A) 自动编码器架构由编码器、解码器和瓶颈层元素组成,后者是学习嵌入表示的位置。(B) 转换器架构,全部由预处理和位置嵌入步骤组成,然后是多个顺序编码器和/或解码器模块。上图:序列到序列的原始或 BART 样式的 Transformer,其中编码器输出可以聚合为学习嵌入表示。中间:仅编码器的 BERT 式 Transformer,通常根据分子分类/回归进行微调。编码器模块的输出可用作学习的嵌入表示。底部:仅解码器的 GPT 风格的 Transformer,其中最终状态可以用作学习的嵌入表示。
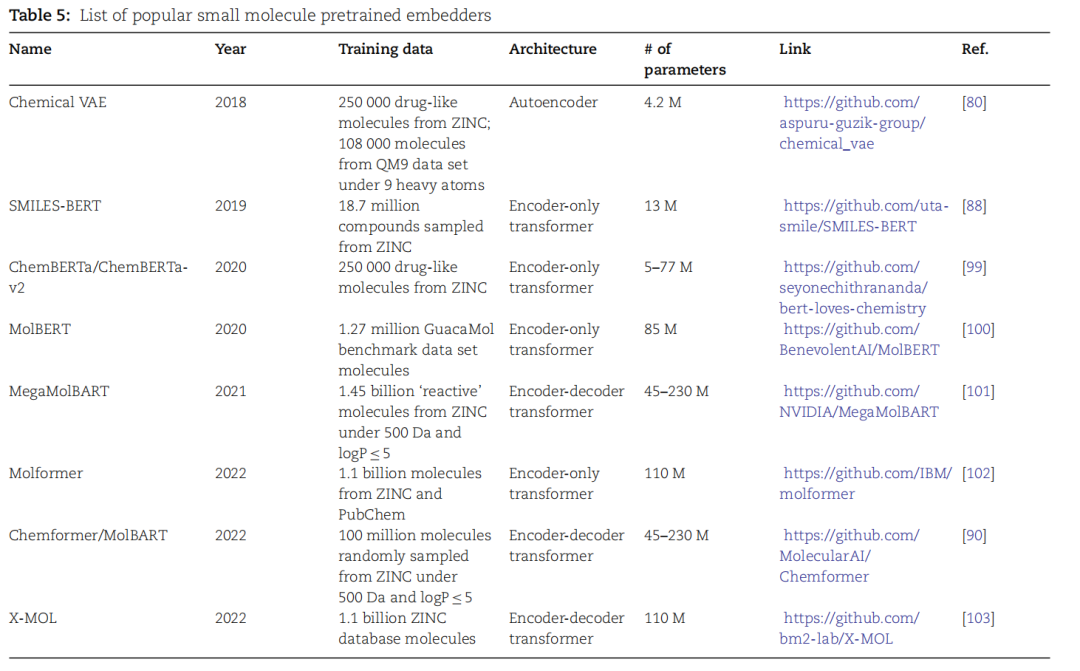
表5.常用小分子预训练嵌入器列表
迁移学习
在面对小规模数据集难以为大型模型找到最佳权重且大型模型难以收敛的挑战时,简单的机器学习模型如基于树的方法通常表现更好。为了克服预训练嵌入器可能缺乏任务特定细节的问题,以及端到端模型嵌入可能无法在化学空间中泛化的挑战,迁移学习成为一个有效的解决方案。迁移学习结合了无监督的预训练步骤和随后的监督微调,通过在大范围无监督数据上训练模型,然后在与下游任务相关的小数据集上微调模型,实现了在任务上的高性能。这种方法的优势在于更多的初始数据允许嵌入器在预训练期间收敛到合理的权重,简化了微调步骤,使模型更容易收敛到高性能的权重组合。尽管预训练过程可能昂贵且复杂,迁移学习仍然是一种有效的策略,已在多个小分子任务上取得成功。
大型语言模型
最近的大型语言模型(LLM),如GPT-4,通过自然语言文本跨领域训练展现了强大的潜在应用。在化学文献等领域,这些模型提取信息的能力尤为有趣。然而,它们在处理化学问题时可能存在准确性不足的问题,例如对分子结构和IUPAC名称的处理。一些项目,如ChemCrow,通过提供专门设计的化学工具,使LLM能够结合外部工具响应,从而解决一些更复杂的化学任务。这种LLM代理在负责任的使用下有助于降低非专家访问信息的障碍,加速研究过程。然而,对于LLM在药物发现等领域的潜在双重用途,需要审慎考虑,并与社区共同讨论设置保障以减轻潜在风险。
总结
药物发现中人工智能科学家和化学信息学家的关键目标是降低成本、时间和失败率,尽管当前面临高成本、低批准率和难以重现性等挑战。然而,生物技术和制药正朝着新的模式扩展,如邻近诱导的新生物学、天然产物、生物制剂等,并利用DNA编码文库、高内涵成像、多组学和人工智能屏幕等强大工具。这为新型分子表示提供了需求和机会。
面临可用数据集规模有限的挑战,一种方法是迁移学习,通过在大型数据集上预训练模型提取一般特征,然后微调以适应特定任务。知识图谱(KG)的整合为分子表征学习提供了强大手段,帮助捕捉分子之间的复杂关系。此外,结合超级计算机的物理仿真为人工智能模型提供了宝贵的领域知识,形成了人工智能在药物发现中的闭环。
可解释性是另一个挑战,尽管深度学习方法在分子表示学习中具有表达能力,但缺乏可解释性。定制的描述符易于解释和分析,对小型数据集具有竞争力。最后,随着更大的治疗方式的重要性增加,需要更适合新治疗方式的分子表示,如核酸、肽和蛋白水解靶向嵌合体(PROTAC)等。整合自然语言处理的进步表明,利用未标记的背景信息可以在药物发现中取得卓越性能。将所有化学知识整合到一个模型中,有望实现更高通量的数据生成,为药物发现提供更好的模型和更准确的预测。
参考资料
Miles McGibbon, Steven Shave, Jie Dong, Yumiao Gao, Douglas R Houston, Jiancong Xie, Yuedong Yang, Philippe Schwaller, Vincent Blay, From intuition to AI: evolution of small molecule representations in drug discovery, Briefings in Bioinformatics, Volume 25, Issue 1, January 2024, bbad422, https://doi.org/10.1093/bib/bbad422



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


