structure prediction,PSSP)模型的论文。蛋白质二级结构预测(PSSP)是预测蛋白质三级结构的关键,近年来其在药物设计和开发领域的需求激增。此外,它还可以用来学习不同蛋白质的功能。本文提出了一种深度学习模型——级联特征学习模型(CFLM),该模型使用基于残差密集网络(RDN)的多阶段迁移学习方法来预测蛋白质的二级结构。该模型在不同级别的迁移学习模型中使用不同的蛋白质数据集进行训练,并接受特定的蛋白质特征,如溶剂可及性、氨基酸残基的物理化学性质、PSSM(位置特定评分矩阵)和PSFM(位置特定频率矩阵)。

1 介绍
蛋白质是负责生物体中所有生物活动的大分子。由于蛋白质可以评估生物体中发生的功能和其他分子机制,因此了解蛋白质的结构非常重要。随着先进技术的应用,新的蛋白质序列被发现并保存在蛋白质数据库(PDB)中。尽管如此,这些序列的许多结构尚未被发现。一级、二级、三级和四级是蛋白质结构的四个不同水平,其中蛋白质的二级结构作为蛋白质一级和三级结构之间的联系,在氨基酸之间的长程相互作用中具有显着特征。蛋白质二级结构的形成是由于多肽链中两个相邻氨基酸残基的酰胺氢和羰基氧之间的氢键连接,能够决定蛋白质折叠的速度。
蛋白质结构可以使用X射线晶体学,核磁共振光谱(NMR)和Cryo-EM进行实验测定。但是这些实验方法中的每一种都有其自身的局限性,这些方法耗时,成本高,并且需要专家进行实验。本文的主要贡献是提出了用于蛋白质二级结构的级联特征学习模型(CFLM),能够快速预测未知结构蛋白质序列的二级结构信息,并使用迁移学习的方法提高了蛋白质二级结构预测的准确率。
本文的工作流程如图1所示,首先使用DeepMSA算法从蛋白质数据库中匹配出每个查询序列的同源序列,进而计算查询序列的PSSM矩阵和PSFM矩阵,然后使用相应方法获取查询序列的溶剂可及性,七种理化性质特征以及二级结构标签。特征获取完毕后,对PSSM矩阵,PSFM矩阵以及七种理化性质特征进行初步特征提取,然后将每个数据样本的特征及标签存入数据库中。最后使用数据库中的数据样本对CFLM模型训练完毕后即可预测未知结构序列的蛋白质二级结构。
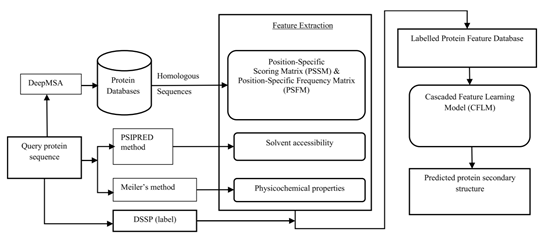
图1 本文工作流程
本文使用的数据集:
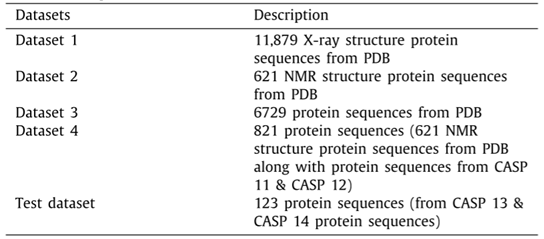
如表1所示,本文使用的所有数据集均是按照一定标准从PDB数据库中筛选得到的。Dataset1中的序列同源性均小于25%,其结构的最小分辨率为2.5埃米,其中所有序列的结构特征均来自X射线实验。Dataset2中的所有序列的结构特征均来自核磁共振波谱实验。Dataset3中的所有序列同源性小于25%且序列长度在26至700之间,其中没有冗余序列,且此数据集中的所有序列存入PDB数据库的时间早于CASP13实验。Dataset4中的序列来自CASP11,CASP12以及核磁共振波谱实验。最后一个数据集作为本文的测试集,其中的序列来自于CASP13和CASP14实验,且所有序列与其他数据集不重复。
此外,本文使用四种特征作为模型的输入,分别是PSSM矩阵、PSFM矩阵、氨基酸残基的溶剂可及性(SEA)以及七种理化性质。PSSM矩阵中的数值表示序列每个位置的氨基酸残基突变为各种氨基酸残基的评分,一条长为L的蛋白质序列的PSSM矩阵维度为21*L;PSFM矩阵中的数值表示序列每个位置的氨基酸残基突变为各种氨基酸残基的概率,一条长为L的蛋白质序列的PSFM矩阵维度为21*L;本文使用PSIPRED程序计算每个氨基酸残基的SEA特征,每个残基的SEA特征为一个3维向量,每个维度分别表示SEA在(30,+∞),(0,10)和(10,30)区间内的可能性;氨基酸残基的七种理化性质特征包括极化率、疏水性、螺旋几率、等电点、归一化范德华体积、空间参数和折叠几率。
表1 本文使用的数据集
本文提出的模型:
如图2所示,本文使用残差密集网络RDN来提取蛋白质的序列特征,每个RDN由多个RDB组成,下图中间部分为一个RDB的内部结构。每个RDB中含有多个卷积层,每个卷积层的输出特征不仅作为其后面卷积层的输入特征,也会和这一RDB后续的所有卷积层的输出特征进行拼接并作为后面卷积层的输入特征。最后这一RDB的输入特征与其中所有卷积层的输出特征进行拼接后再次进行卷积操作,将得到的特征与这一RDB的输入特征进行相加,作为这一个RDB的输出特征。本文的残差密集网络由20个RDB组成,首先依次通过20个RDB提取查询序列的特征,然后将这20个RDB提取到的特征进行拼接,在进行卷积操作,将得到的特征与模型最开始的输入特征进行相加,然后通过一个1*1卷积和Softmax操作得到二级结构的预测结果。其次,需要注意的是,在查询序列特征输入到RDN之前,还经过了行列归一化,ReLU激活层,卷积层和Maxout激活层的前期处理。
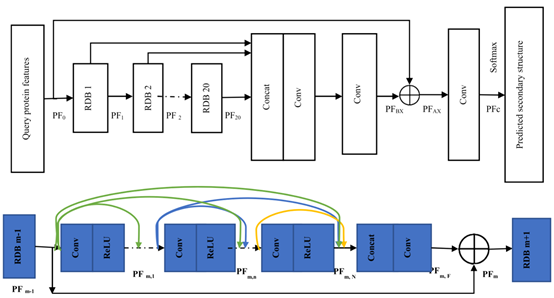
图2 RDN、RDBs模型架构
如图3所示,本文使用迁移学习的方法对模型进行训练。具体方法为:首先使用Dataset1直接对RDN模型进行训练,然后进行第一级迁移学习,通过阻塞训练好的RDN的前11个RDB,使用其他数据集对后9个RDB进行训练,最后进行第二级迁移学习,本文通过继续阻塞第12至第15个RDB,使用Dataset4对最后五个RDB进行训练。
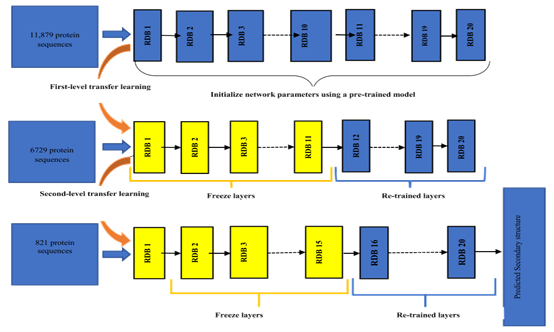
图3 迁移学习策略
2 结果
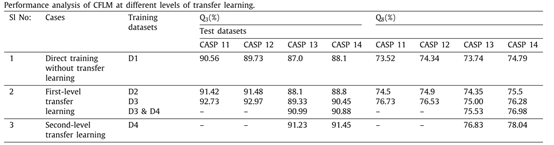
如表2所示,本文首先探究了迁移学习策略对模型性能的影响:表格第一行表示只使用Dataset1对模型进行训练的情况,第二行表示使用不同数据集对模型进行一级迁移学习的情况,第三行表示使用Dataset3对模型进行一即迁移学习并使用Dataset4对模型进行二级迁移学习的情况。Q3和Q8分别表示蛋白质二级结构三态预测和八态预测的准确率。从表2中可以看出,使用Dataset2对模型进行一级迁移学习时,相较于不进行迁移学习的模型,预测准确率提高了约1%,而使用Dataset3对模型进行一级迁移学习时,相较于不进行迁移学习的模型,预测准确率提高了约2%,而当使用Dataset3和Dataset4组合数据集对模型进行一级迁移学习时,模型的预测性能进一步提升。当使用Dataset3进行第一级迁移学习,使用Dataset4进行第二级迁移学习时,相较于其他模型,在CASP13和CASP14上的Q3和Q8指标均有所提升。
表2 迁移学习策略对模型性能的影响
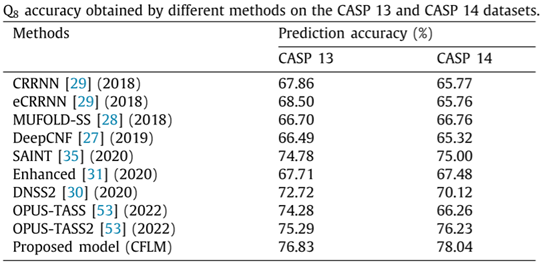
本文使用CFLM模型与其他先进的蛋白质二级结构预测模型进行了对比,表3和表4分别为模型的三态和八态预测的结果对比,可以看出,无论是三态预测还是八态预测,本文提出的CFLM模型均达到了最佳性能。
表3 蛋白质二级结构三态预测结果对比
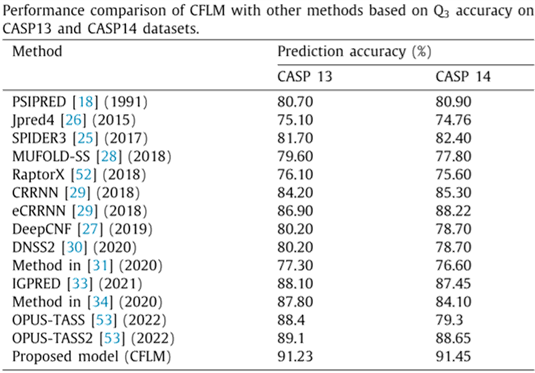
表4 蛋白质二级结构八态预测结果对比
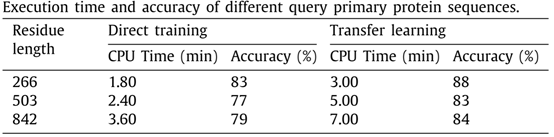
此外,本文也对CFLM模型的计算时间复杂度进行了分析,分析结果如表5所示。表5中展示了对于不同长度的查询序列,CFLM所花费的计算时间,经分析得出模型的时间复杂度为k*O(n)d,其中k为模型中ReLU函数的数量,d为一个RDB模型中的特征维度。
表5 CFLM模型计算时间复杂度分析
3 结论
本文提出了一种新的蛋白质二级结构预测模型——级联特征学习模型(CFLM),该模型使用基于残差密集网络(RDNs)的多阶段迁移学习方法来预测蛋白质二级结构。它使用蛋白质序列的PSSM矩阵、PSFM矩阵、氨基酸残基的溶剂可及性以及七种物理化学性质特征作为输入。这些特征来源于已知的蛋白质序列及其相应的同源序列。
使用DeepMSA算法检索每个初级蛋白查询序列对应的同源序列。结果表明,使用Q3和Q8指标进行评估时,在CASP13和CASP14基准数据集上,CFLM比其他比较方法的准确性更高。其次,本文在迁移学习过程的不同层次上分析了CFLM的有效性,并发现基于级联特征的迁移学习方法能够显著提高预测精度。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


