点击蓝字 关注我们
AItellU
编者 | 刹那芳华
制版 | 戈戈

第一作者:Zijun Zhang
通讯作者:Olga Troyanskaya
第一单位:美国西达-西奈医疗中心
DOI:10.1038/s43588-023-00569-1
1
全文一句话速览
本文可解释网络Elektrum预测CRISPR-Cas9脱靶酶促反应速度与结果,证明Elektrum在保证强大预测能力的同时保持了物理可解释性。
2
研究背景
酶促反应在细胞过程中至关重要。然而,由于反应途径的复杂性和细胞环境的干扰因素,发现和测试其生化动力学模型非常困难。研究者提出了一种新的分析工具——可解释的神经网络(INN),以更好地理解细胞的化学动力学。然而,现有的可解释神经网络结构存在一些限制,例如需要在训练前提供候选动力学模型,且训练数据集受到体外实验数据的限制。这些限制影响了这些模型在理解体内场景时的性能。因此,作者开发Elektrum在保证物理可解释性的同时提高对反应的预测能力。
3
全文亮点
Elektrum在保证物理可解释性的同时提高对CRISPR-Cas9脱靶酶促反应的预测能力
4
图文解析
Elektrum框架概述
Elektrum旨在模拟体外和体内序列依赖的酶动力学(图1)。Elektrum利用神经体系结构搜索(NAS)和迁移学习技术,自主探索KINN(动态可解释神经网络)模型的体系结构空间,旨在揭示系统的潜在动力学。该框架最初通过概率建模遗传算法生成一组候选KINN,预测体外可测量的运动速度。随后,第二个NAS步骤将KINN与卷积神经网络(CNN)相结合,以捕获体内数据中细微差别的序列依赖信号。预先训练的KINN模型充当中间层,优化体内模型的性能。
Elektrum基于数据驱动的方法高效地构建了多个候选动力学模型,超过了人类派生的模型。KINN体系结构包括对DNA引导RNA(GRNA)子序列进行采样,计算动力学速率,并利用三层King-Altman神经网络来估计稳态生产率。该框架引入了一个可区分的特征分解层来模拟非稳态动力学系统动力学,这对于理解蛋白质结合和切割等实验设置至关重要。贝叶斯概率遗传算法根据KINN性能更新模型结构分布,在识别模拟动力学系统的准确模型方面显示出优于随机抽样的优势。为了评估体内反应,Elektrum创新性地将预先训练的KINN作为特殊的神经网络层,通过考虑体内序列背景下对动力学速率的扰动影响来优化体内模型的性能。转移学习方案增强了整体模型的性能,展示了Elektrum在揭示复杂酶动力学方面的有效性。
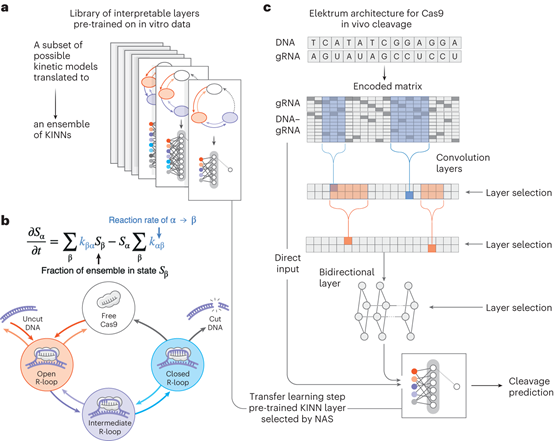
图1 Elektrum框架概述
用KINN搜索模拟Cas9体外脱靶切割
利用Elektrum对Cas9蛋白在DNA序列结合和切割过程中的酶途径进行建模,解决了切割速度依赖于引导RNA(GRNA)与DNA匹配所带来的动力学学习问题。Elektrum利用已发表Cas9解离的体外大规模平行动力学图谱数据集,优化了KINN,以准确预测Cas9解离率。Elektrum基于专家策划的动力学模型创建KINN模型空间,使用遗传算法从以专家知识为中心的模型空间中对KINN进行采样。优化的KINN在测试皮尔逊相关性方面比基准随机采样CNN提高了25.2%。此外,优化的KINN需要显著更少的参数,转化为更快的训练时间,并探索具有不同数量的酶状态的复杂自由能场景,为基于序列和细胞背景的体内转移学习提供可解释性和高预测性(图2)。
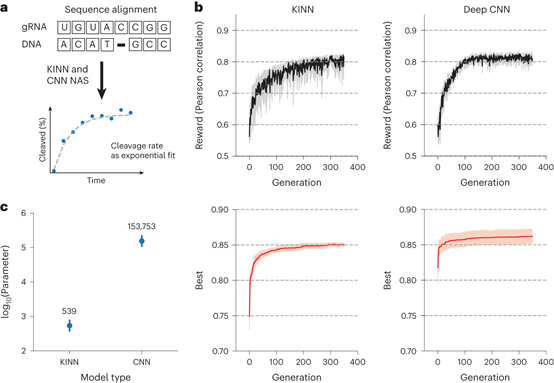
图2 寻找体外Cas9解离动力学的KINN结构
利用体外KINNs捕获体内脱靶编辑
准确的靶外切割预测对于Cas9在研究和治疗应用中的有效和安全使用至关重要。实验方法,如GUIDE-SEQ、CIRCLE-SEQ和SITE-SEQ,提供了关于活细胞中非靶点位置的有价值的数据,但缺乏一个全面的框架来预测非靶点切割频率和时间依赖性。本研究使用体内数据集作为抵抗,以评估基于体外数据训练的KINN在预测Cas9脱靶解离率方面的性能。KINN模型显示,预测的解离率和观察到的非目标编辑水平之间存在很强的正相关。KINN在体内预测中的表现优于传统的Cas9非目标预测值。此外,KINN具有学习的基础机制模型,提供了推广到训练数据参数之外的系统的潜力,使改进的全基因组编辑协议的设计成为可能。
整合NAS与训练的KINNs以改善体内预测
通过Elektrum的迁移学习策略实现的,该策略将训练有素的KINN与卷积骨干相结合(图3)。这个由CNN架构搜索算法创建的卷积骨干,解释了在体外学习的动力学速率中没有捕捉到的序列-上下文效应。探索了多个候选KINN,以自动确定体内数据集上最具预测力的最优动力学模型。KINN的完全可解释性允许卷积骨干在保持体外KINN冰冻重量的同时仅学习体内特定效应的修饰性术语。与仅基于体外数据训练的基础KINN相比,通过随机NAS应用的迁移学习策略改善了体内的性能。由此产生的Elektrum模型集成在验证和外部数据集上表现出了卓越的测试性能,在某些情况下甚至超过了无法解释的最先进的预测值。
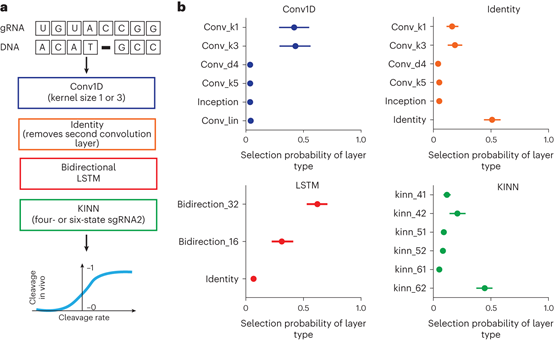
图3 NAS中迁移学习的集成用于体内解离预测
序列背景对体内Cas9解离的影响
Elektrum在预测体内动力学速率方面具有独特的优势,通过比较Elektrum的体内模型预测和KINN的体外预测,可以评估对Cas9体内编辑至关重要的序列背景。尽管体外解离率和体内预测的解离率之间存在很强的正相关性,但在不同的数据集中,体内解离率始终高于体外解离率。这种对体外解离率的低估在外部数据集中一直被观察到,表明特定的序列元件参与了体内切割激活。使用Shapley加性解释来区分体内预测的和体外预测的Cas9解离率差异的序列决定因素。Shap分析强调了Cas9靶区的鸟嘌呤对于增加体内切割的重要性,与靶区的其他核苷酸相比,鸟嘌呤表现出明显更高的Shap值(图4)。这表明富含鸟嘌呤的非靶区更有可能在体内发生切割,这与之前的报道一致。
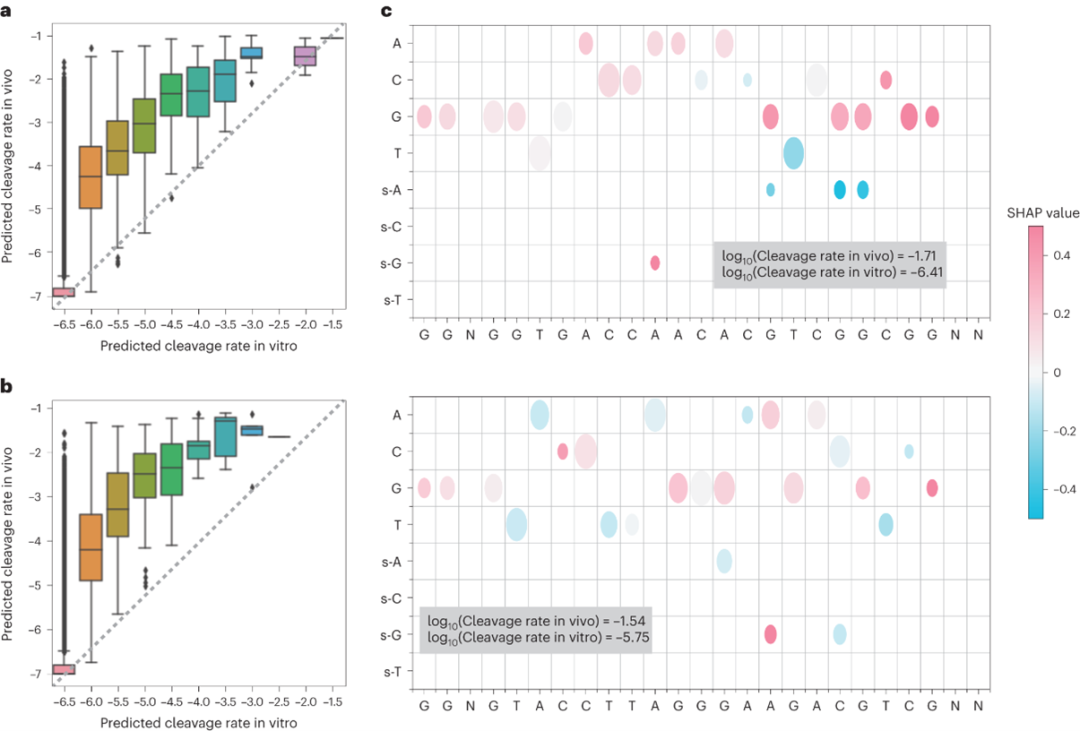
图4 体内Cas9裂解率预测的序列背景分析
5
通讯作者介绍
Olga Troyanskaya,普林斯顿大学教授。研究方向:1.机器学习方法与应用;2.人类疾病与精准医学;3. 交互式公共系统和数据可视化。
6
编者有话说
Elektrum的设计目的是在几乎没有先验知识的情况下有效地生成和应用于体内过程的动力学模型,主要关注DNA切割的动力学作为预测Cas9编辑的主导过程。Elektrum的可解释神经体系结构搜索(NAS)利用KINN,通过支持较低复杂性的CNN来展示效率,而不是假设需要更复杂的模型来封装额外的过程。尽管Elektrum在安全基因组编辑的gRNA目标优先顺序方面很有效,但当以实验测量的非目标为基准时,它确实会做出假阳性和假阴性预测。KINN模型的可解释性提供了生物物理见解,揭示了Cas9/gRNA“搜索空间”的可变性和非靶标浓度依赖过程对Cas9切割效率的影响等因素。Elektrum在利用序列类输入方面的多功能性使其能够适应各种动力学模型,有可能在未来将其应用于其他蛋白质和不可逆转的生化反应。
转载须知
【原创文章】AItellU原创文章,欢迎个人转发分享,未经允许禁止转载,所刊登的所有作品的著作权均为AItellU所有。AItellU保留所有法定权利,违者必究。

投稿、转载及合作邮箱:
zjhuangjunjie@163.com
原文链接:
https://www.nature.com/articles/s43588-023-00569-1
点击下方蓝字阅读原文



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


