点击蓝字 关注我们
AItellU
编者 | 刹那芳华
制版 | 戈戈

第一作者:Felix Wong
通讯作者:James J. Collins
第一单位:Broad研究所
DOI:10.1038/s41586-023-06887-8
1
全文一句话速览
本文开发可解释的基于化学子结构的深度网络Chemprop,测定了39,312个化合物的抗生素活性和人类细胞毒性图谱,并应用图神经网络集成预测了12,076,365个化合物的抗生素活性和细胞毒性,用于新抗生素挖掘。
2
研究背景
持续的抗生素耐药性危机有可能使目前的抗生素失效,并增加细菌感染的发病率。如果没有新的抗生素,到2050全球因耐药感染而死亡的人数预计将达到每年1,000万人。在过去的十年里,通过基于天然产物挖掘,高通量筛选,进化和系统发育分析,结构指导和合理设计的各种方法,以及使用机器学习的虚拟筛选,已经发现了抗生素候选。然而,开发有效的抗生素发现方法仍然是一个挑战,迫切需要新的方法来发现抗生素。
3
全文亮点
1.利用深度学习挖掘新类别抗生素
2.用蒙特卡罗树方法分析了分子结构对最终预测值的贡献
4
图文解析
抗生素活性模型
研究首先对一组初始化合物进行了筛选,包括大多数已知的抗生素、天然产物和结构多样的分子,以评估它们对甲氧西林敏感菌株S. aureus RN4220的生长抑制活性,共获得512个活性化合物(占所有化合物的1.3%)。使用Chemprop在筛选数据上训练了一组图神经网络,以根据化合物的化学结构对其是否抑制细菌生长进行二分类预测。每个图神经网络通过执行卷积步骤来操作,这些卷积步骤依赖于每个输入化合物结构的原子和键,该结构被视为一个数学图,具有顶点(原子)和边(键)(图1a)。为了提供可能改善模型性能的额外数据,为每个输入提供了由RDKit计算的分子特征列表。然后,对同一训练数据集的80% -20%拆分进行了每个模型的训练和验证,然后在测试集上进行测试。对应用于预留的测试数据的十个模型的集合,精度-召回曲线下面积(AUPRC)为0.364,表明在考虑训练数据中活性化合物的不平衡性时,性能良好(图1c)。
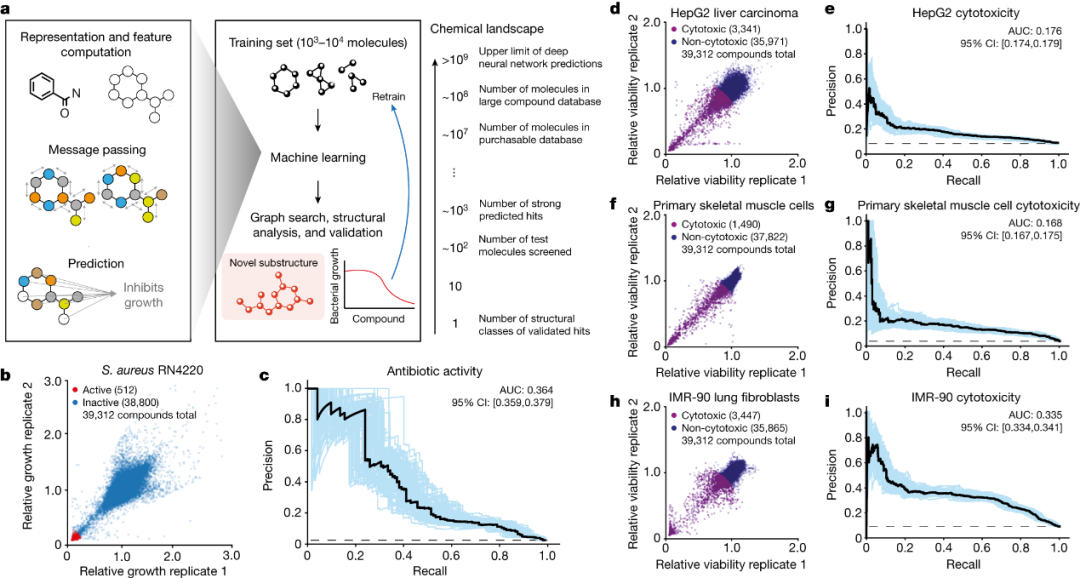
图1 用于预测抗生素活性和人类细胞毒性的深度学习模型集合
人类细胞毒性模型
对39,312个化合物的训练集进行了反筛选,以确定其对人肝癌细胞(HepG2)、人原代骨骼肌细胞(HSkMCs)和人肺成纤维细胞(IMR-90)的细胞毒性。使用严格的90%细胞活性截断将化合物活性二值化,结果分别有3,341(8.5%)、1,490(3.8%)和3,447(8.8%)化合物被归类为对HepG2细胞、HSkMCs和IMR-90细胞具有细胞毒性,而在512种活性抗菌化合物中,306对所有三种细胞类型都不具有细胞毒性(图1d、f、h)。用这些数据训练二分类模型,该模型根据化合物的化学结构预测新化合物是否对HepG2细胞、HSkMCs或IMR-90细胞具有细胞毒性的概率。对于训练和验证的10个Chemprop模型,然后在相同的80%-20%的数据分割上进行测试,HepG2、HSkMC和IMR-90模型的AUPCC值分别为0.176、0.168和0.335(图1E,g,i)。
过滤和可视化化学空间
用每个训练数据集的完整数据重新训练了20个Chemprop模型的集成,产生了四个预测抗生素活性、HepG2细胞毒性、HSkMC细胞毒性和IMR-90细胞毒性的集成。应用这些系综来预测12,076,365种化合物的抗生素活性和细胞毒性谱。
根据预测的抗生素活性和细胞毒性筛选了感兴趣的化合物,首先保留了Mcule数据库中抗生素预测分数大于0.4的3,004种化合物,以及来自布罗德研究所数据库(图2a,b)的7,306种抗生素预测分数大于0.2的化合物。然后,保留了那些HepG2、HSkMC和IMR-90细胞毒性预测分数低于0.2的化合物,经过严格的筛选,得到了3,646种化合物–来自Mcule数据库的1,210种化合物和来自其他数据库的2,436种化合物–占所有评估化合物的0.03%(图2a,c-e)。
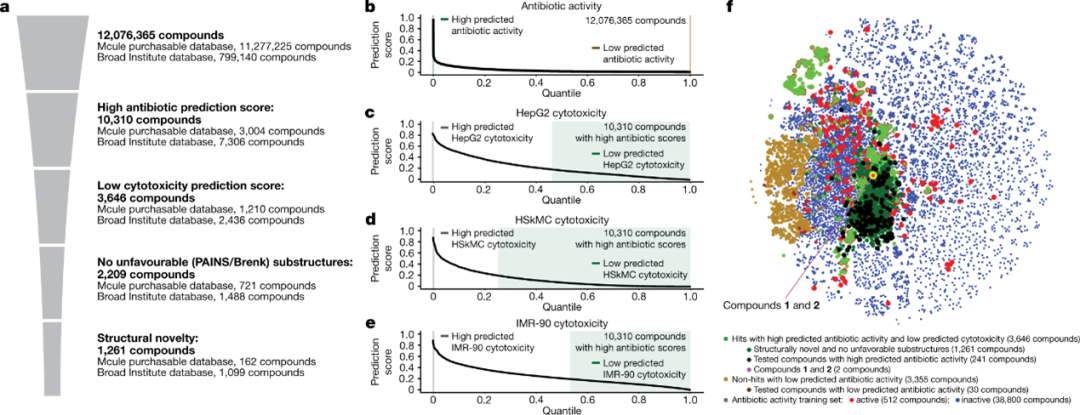
图2 过滤和可视化化学空间
使用t分布随机邻近嵌入25(t-SNE)对Morgan指纹进行分子表示,可视化了化学空间。这表明HITS在结构上与非HITS不同,而训练集包括来自不同类别已知抗生素的化合物,在很大程度上区分了非HITS和HITS(图2F)。值得注意的是,正如t-SNE和我们随后的基于子结构的分析(图3)所表明的那样,多个命中在结构上与训练集中的活性化合物不同,这表明模型外推到了未知的化学空间。
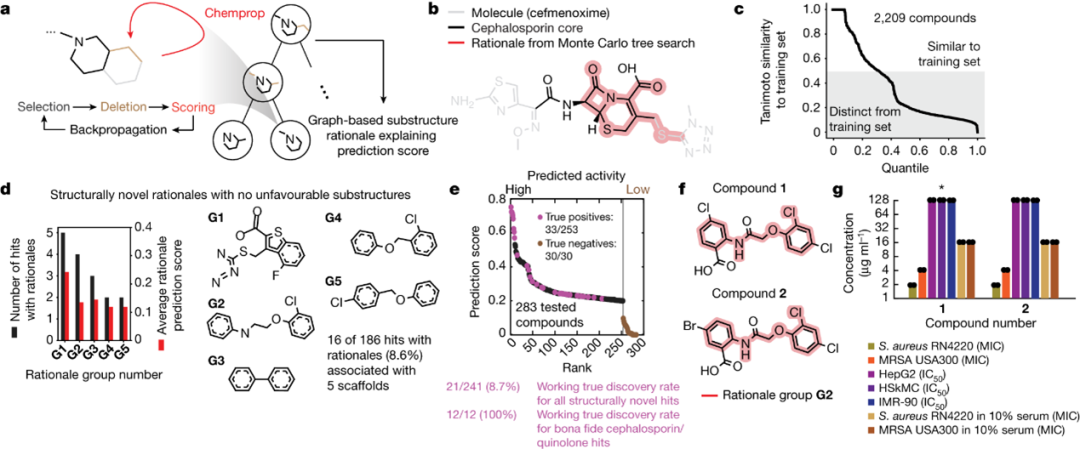
图3 基于分子图原理揭示了未来抗生素类别
解释预测抗生素种类
为了提高模型的可解释性,使用基于图的搜索算法确定这些“解释”或子结构,以更好地理解化合物的抗生素预测分数。通过Monte Carlo tree搜索,确定了具有至少八个原子且显示高于0.1的抗生素预测分数的解释,从而提供了模型的可解释性保证(图3a)。首先通过quionlones和β-内酰胺两个在训练数据中丰富的结构类别的“留一法”分析验证了理性计算的有效性,结果表明作者的方法能够识别出训练中未见的新的抗生素骨架(图3b)。将理性分析应用于整体模型的筛选结果,发现380个(总计3,646个)化合物的解释,其中许多与已知抗生素类的结构片段相符,同时也包括一些与任何已知抗生素类别都不相关的解释。这一方法有望帮助更好地过滤出具有结构新颖性的感兴趣的化合物,并深入研究其相应的解释。
新型过滤的子结构
模型通过图形理性的能力预测具有高抗生素预测分数的亚结构(图3a,b),接下来试图识别模型预测的结构新颖的抗生素类别。去除了所有包含PAINS和Brenk警报的HITs(偶发反应、诱变性或药代动力学不利的亚结构)。这将3,646个预测的HITs缩减到2,209个(图2a)。计算了每个HITs与训练集中任何活性化合物的最大Tanimoto相似性,并将具有最大相似性分数≤0.5的命中物列入初步筛选名单(图3c),以及那些不含β-内酰胺环或喹诺酮双环核心的HITs,产生了最终的1,261个HITs。最终计算显示,186个HITs具有解释性。通过计算确定了至少有12个原子的化学支架,这些原子在原理上是保守的。通过这种方法,作者发现186个具有可解释性的hits中的16个可以使用5个不同的支架–G1-G5(图3D)–分组。
可解释抗生素新结构
实验结果显示,与结构类别G1-G5相关的9个化合物中有4个对S. aureus表现出抑制生长的活性,最小抑制浓度(MIC)≤ 32 μg ml−1。与G1-G5无关的45个化合物中没有一个表现出活性,而187个没有理论基础的化合物中有17个(9.1%)表现出活性。新颖结构的有效命中物的真正发现率较高(7.4%),跨所有测试的新颖结构的发现率为8.7%,这比训练集中活性化合物的比例(1.3%)更高,表明深度学习模型对于广泛的化学空间的泛化具有实用性。对所有结构新颖的命中物,尤其是G1-G5的四个活性化合物,进行了进一步的实验验证,证实它们对MRSA同样具有活性,并在10μM的浓度下对人细胞无细胞毒性。其中,化合物1和2表现出较高的选择性和有利的药代动力学特性,呈现出进一步开发的潜力。这项研究通过实验验证了深度学习模型预测的结构新颖的抗生素类别,为新型抗生素的发现提供了一种创新的方法。
作用机理与耐药性
与氨苄青霉素、环丙沙星和四环素相比,药物对MRSA相对于甲氧西林敏感株的MIC至少增加了16倍,但对化合物1和2的MIC只增加了两倍,这表明这些化合物可能与β-内酰胺、氟喹诺酮和四环素不同机制的作用。这些化合物对革兰氏阴性菌具有特异性,因为它们不抑制大肠杆菌、鲍曼不动杆菌或假单胞菌的生长。在液体培养基中连续用化合物1和2处理的S. aureus RN4220,发现MIC在30天内基本保持不变(图4b)。相反,培养物在30天后对环丙沙星的MIC至少增加了64倍(图4b)。在抑制突变体产生实验中,作者在含有超常MIC水平的化合物1和2的固体培养基上接种了高接种量的S. aureus RN4220,并发现在5天后只有在4×MIC存在下出现菌落(图4c),这表明存在低水平的抗性。单细胞成像显示,用化合物1或2处理的细胞发生溶解(图4d),与这些化合物的杀菌活性一致(图4a),暗示了细胞包裹物的靶向作用机制。进一步地,化合物1和2处理都导致金黄色葡萄球菌和枯草杆菌中DiSC3(5)的荧光猝灭,表明这两种化合物都破坏了ΔpH(图4e)。这证明化合物的杀菌机理是裂解细菌细胞膜。
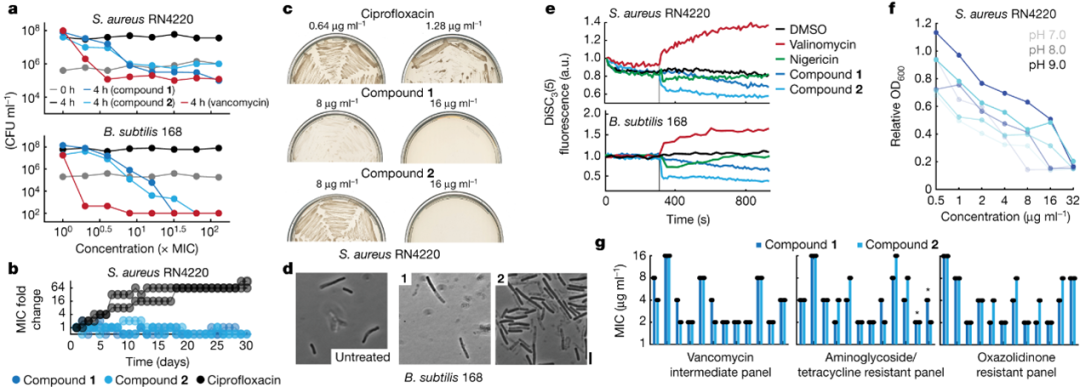
图4 化合物的作用机理与耐药性
毒理学、化学性质及体内功效
研究了化合物1在小鼠局部和全身应用时治疗MRSA的疗效。使用耐氨基糖苷和四环素的MRSA临床分离株,在中性粒细胞减少的小鼠浅表皮肤感染模型上测试了局部给药。与赋形剂(图5a)相比,化合物1的治疗使平均细菌负荷减少了约1.2个数量级。进一步使用耐恶唑烷酮的MRSA临床分离株,在中性粒细胞减少的小鼠大腿感染模型中测试了化合物1的系统给药。与赋形剂处理相比,化合物1以80 mg kg−1处理显著降低了平均细菌负荷约1.2个数量级(图5b)。
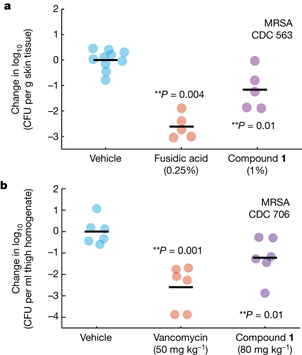
图5 体内治疗效果
5
通讯作者介绍
James J. Collins,Broad研究所教授。研究方向:1.抗生素与AI;2.合成生物学。
6
编者有话说
传统药物发现领域在发现新的抗生素结构方面面临挑战,而本研究利用图形神经网络的解释性模型,成功地在庞大的化学空间中预测了对金黄色葡萄球菌具有抗菌活性的多个化合物。其中,一个结构类别表现出很高的选择性,克服了耐药性,具有良好的毒理学和化学性质,在小鼠感染模型中对耐甲氧西林金黄色葡萄球菌(MRSA)的局部和全身治疗均有效。这项工作展示了一种深度学习方法,该方法在预测单一化合物命中的基础上系统地探索了庞大的化学空间,为发现和设计抗生素提供了新的思路,特别是那些能够克服对抗革兰氏阴性菌的耐药性的抗生素。研究结果表明,深度学习模型在药物发现中可以变得可解释,这为更好地理解和利用这些模型提供了可能性,为下一代药物探索提供了有益的见解。
转载须知
【原创文章】AItellU原创文章,欢迎个人转发分享,未经允许禁止转载,所刊登的所有作品的著作权均为AItellU所有。AItellU保留所有法定权利,违者必究。
原文链接:
https://www.nature.com/articles/s41586-023-06887-8
点击下方蓝字阅读原文



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


