今天为大家介绍的是发表在ICLR
2023上的一篇关于分子生成的论文。由于3D空间中的原子相互作用是显式建模的,在靶向药物设计过程中包含3D结构显示出优于其他无靶标模型的性能。然而,目前的3D靶标感知模型要么依赖于体素化的原子密度,要么依赖于自回归采样过程,这两种方法都不具有旋转等变性,并且容易违反几何约束,导致生成的结构不真实。在这项工作中,本文开发了一个3D等变扩散模型来解决上述挑战。

1 介绍
最近,专门针对靶标感知分子生成任务提出了新一代生成模型。然而,现有方法存在一些缺点。例如,Tan 没有明确地模拟 3D 空间中分子原子和蛋白质之间的相互作用,而只将靶标视为中间条件嵌入。Ragoza 等人将 3D 空间表示为体素化网格,并使用 3D 卷积神经网络(CNN)对蛋白质和分子进行建模。但是,该模型不具备旋转等变性,因此无法完全捕获3D结构信息。最后,目前的自回归模型无法估计生成分子的质量。人们必须依靠其他基于物理化学能量函数的工具,例如AutoDock来选择候选药物。
为了解决这些问题,本文提出了TargetDiff,这是一种3D全原子扩散模型,以非自回归方式生成靶标感知分子。该模型可以基于蛋白质原子提供的上下文信息在连续的 3D 空间中生成分子,并且具有结合复合物的全局平移和旋转的不变性。本文定义了连续原子坐标和离散原子类型的扩散过程,在这个过程中,噪声是逐渐添加的,并使用 SE(3)等变图神经网络学习联合生成过程,该神经网络交替更新分子的原子隐藏嵌入和原子坐标。在特定参数设置下,可以通过前向传递输入分子一次而不进行重新训练来从模型中提取代表性特征。实验发现这些特征提供了强大的信号来估计采样分子和靶蛋白之间的结合亲和力,然后可用于对候选药物进行排名并改进其他用于结合亲和力预测的监督学习框架。实验结果表明,TargetDiff 生成的分子具有更逼真的 3D 结构和更好的与蛋白质结合位点的结合能。
2 实验方法
2.1 概述
本文开发了 TargetDiff,这是一种基于 DDPM 框架的等变非自回归方法,用于生成靶标感知分子。如图1所示,该方法包括正向扩散过程和反向生成过程,两者都定义为马尔可夫链。扩散过程逐渐将噪声注入数据,生成过程学习使用θ参数化的网络从噪声分布中恢复数据分布。
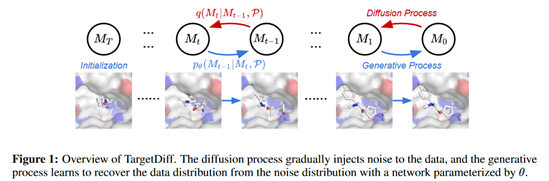
图1
2.2 分子扩散过程
使用高斯分布 N 来模拟连续原子坐标 x,使用分类分布 C 来模拟离散原子类型 v。原子类型被构造为一个one-hot向量,其中包含元素类型和芳香环中的成员身份等信息。将分子分布表述为原子坐标分布和原子类型分布的乘积。在每个时间步长t中,根据具有固定方差调度的马尔可夫链,将小的高斯噪声和所有类别的均匀噪声分别添加到原子坐标和原子类型中。
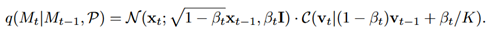
将联合分子分布分解为扩散过程中原子坐标和原子类型的两个独立分布的乘积,

2.3 等变分子生成过程的参数化
生成过程将从初始噪声MT中恢复真实分子 M0 ,使用由 θ 参数化的神经网络近似反向分布:
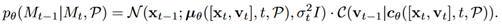
生成过程的一个期望特性是 pθ(M0|P)应该对蛋白质-配体复合物是平移和旋转不变的,这是生成分子等3D对象的关键。
将 SE(3)变换表示为 Tg,可以在蛋白质-配体复合物上实现关于Tg 的不变性。将蛋白质原子的质心(CoM)移至零,并使用 SE(3)等变网络参数化马尔可夫状态转移过程。
受到了近期等变神经网络进展的启发,采用了SE(3)等变图神经网络来模拟配体分子原子与蛋白质原子之间的相互作用。
在第l层,原子隐藏嵌入h和坐标x交替更新如下:
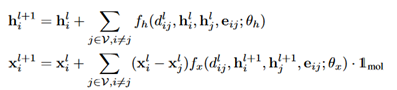
其中 dij=‖xi−xj‖是两个原子i和j之间的欧式距离,eij是一个附加特征,表明连接是蛋白质原子之间、配体原子之间或蛋白质原子和配体原子之间的。1mol是配体分子掩码,用于防止更新蛋白质原子坐标。初始原子隐藏嵌入h0由对原子信息进行编码的嵌入层获得。将最终的原子隐藏嵌入hL馈入多层感知器和softmax函数中,得到v0的预测值。
2.4 训练目标
对于原子坐标损失,由于q(xt−1|xt,x0)和pθ(xt−1|xt)都是高斯分布,因此KL散度可以写成封闭形式:
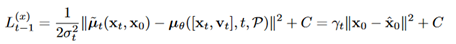
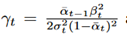
C 是一个常数。在实验中,使用未加权的 MSE 损失(设置 γt =
1)训练模型也可以获得更好的性能。
对于原子类型损失,可以直接计算分类分布的KL散度,如下所示
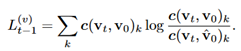
最终损失是原子坐标损失和原子类型损失的加权和:
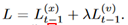
TargetDiff 的整体训练和采样过程如下表所示
表1


2.5 作为无监督学习器的亲和力排名和预测
生成模型是无监督的学习器。然而,在靶标感知分子生成领域,还没有人建立生成模型和结合亲和力之间的联系,这是评估生成分子的重要指标。现有的生成模型无法(准确)估计生成分子的质量。
首先建立了无监督生成模型与结合亲和力排序/预测之间的联系。给定蛋白质-配体复合物,可以将[x0,
v0]输入φθ中,同时冻结x更新分支(即只更新原子隐藏嵌入h),最终得到hL和v0的预测值。
本文的假设是,如果配体分子与蛋白质的结合亲和力较好,则原子类型的灵活性应该较低,这可以从ˆv0的熵中反映出来。因此,它可以作为评分函数来帮助排序,其有效性在实验中得到了验证。
3 实验结果
实验使用CrossDocked2020数据集来训练和评估TargetDiff。从分子结构、靶标结合亲和力和分子特性等角度比较了本文的模型和基线的性能。
首先,在图2中绘制了所有原子间距离和碳碳键距离的经验分布,然后将它们与参考分子的相同经验分布进行比较。对于整体原子距离,TargetDiff 可以很好地捕获整体分布,而 AR 和 Pocket2Mol 对小原子距离具有过度表示。liGAN只能捕获整体形状,而不能指定模态。
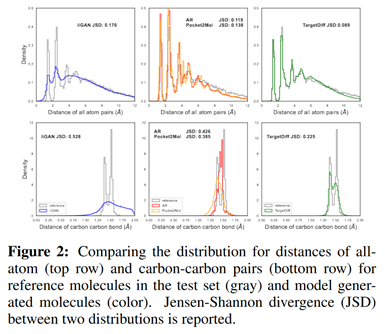
图2
其次,用默克分子力场(MMFF)对生成的结构进行优化,并计算了不包含任何可旋转键的不同刚性片段的MMFF优化前后的坐标之间的均方根偏差RMSD。如图 3 所示,TargetDiff 能够生成更一致的刚性片段。
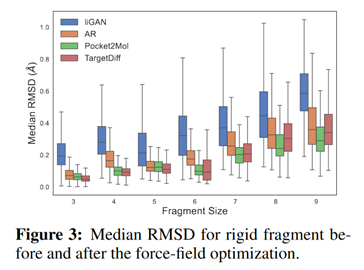
图3
图 4 显示了每个结合口袋的所有生成分子的中值 Vina 能量(由 AutoDock
Vina 计算)。基于Vina能量,TargetDiff生成的分子在57%的靶点中显示出最佳的结合亲和力,而从liGAN、AR和Pocket2Mol生成的分子仅在4%、13%和26%的靶标具有最佳结合亲和力。在高亲和力结合方面,实验发现平均58.1%的TargetDiff分子表现出比参考分子更好的结合亲和力,这明显优于其他基线。
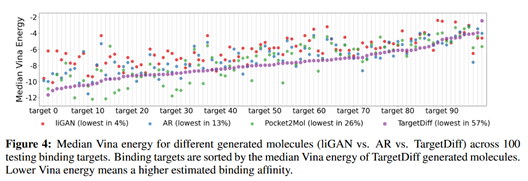
图4
除了结合亲和力外,本文还进一步调查了生成分子的其他分子属性,包括药物相似性QED、可合成性SA以及多样性。如表2所示,与liGAN、AR和GraphBP相比,TargetDiff可以生成更多的高亲和力结合分子,同时保持与其他2D指标相似的其他指标。
表2
如图5所示,实验发现:(1)去噪原子类型ˆv0的熵(即v_ent)与pK具有合理相关性,表明无监督学习可以提供一定程度的结合亲和力排名信息。(2)熵得分向传统的化学/物理得分函数(如Vina)提供了一些补充信息,因为它们的组合(即combined)取得了更好的相关性。(3)当提供带标签的数据时,简单的线性转换后的最终隐藏嵌入hL(即hidden emb)可以大幅提高相关性。实验进一步证明,本文的无监督学习特征可以改善 PDBBind v2020 数据集上的有监督亲和力预测。
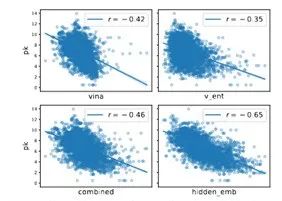
图5
4 总结
本文提出了TargetDiff,一种用于靶标感知分子生成和增强结合亲和力预测的3D等变扩散模型。基于该模型提出了一种有效的方法来评估生成分子的质量,其中模型既可以用作评分函数以帮助排序,也可以用作无监督特征提取器以提高结合亲和力预测。



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


