今天为大家介绍的是来自Jürgen Bajorath团队的一篇论文。图神经网络(GNNs)通过分析蛋白质与配体相互作用的图表示结构来预测配体的亲和力。虽然有一些研究表明GNNs能够详细了解蛋白质与配体的相互作用,但这种预测方法也存在争议。比如,有证据显示GNNs可能并不是真正学习蛋白质与配体的相互作用,而是仅仅记住了训练数据中的配体和蛋白质信息。为此作者对六种不同的GNN架构进行了亲和力预测实验,并使用可解释的人工智能技术对结果进行了分析。

化合物效能预测是药物设计中的核心任务,目前有多种计算方法。在基于配体的设计中,这些方法包括用于定量结构-活性关系建模的多元线性回归模型,支持向量回归,或随机森林回归。此外,深度神经网络(DNNs)的使用也越来越广泛。在基于结构的设计中,配体结合能量大致通过不同类型的基于分子力学的评分函数来近似估算,或者使用量子力学和分子力学以及相对自由能扰动计算来预测。与基于配体的预测相似,深度学习的日益普及也激发了基于结构设计中DNN的应用,比如使用卷积DNN从配体结合位点的体素表示中预测配体亲和力。此外,图神经网络(GNNs),包括信息传递神经网络(MPNNs),越来越多地被用于从蛋白质-配体复合物的解析结构获得的蛋白质-配体相互作用图预测亲和力。目前已经研究人员提出了各种GNN亲和力预测模型。对于这些模型,经常观察到预测和实验配体亲和力之间的强相关性,这导致人们认为GNNs可能能够学习蛋白质-配体相互作用及其相关能量。尽管很难想象,蛋白质-配体相互作用的物理基础和热力学关系可能从相对简单的图表示中学到,但GNNs所取得的明显预测准确性激发了对基于图的亲和力预测在基于结构的药物设计中的高期望。另一方面,这些亲和力预测结果也存在争议。例如,在GNN预测中,不同的训练数据经常产生与实验数据相似的相关性,这对于深度学习来说是违反直觉的,而不同的训练和测试数据的划分导致模型性能有显著差异,也引起了关注。
鉴于对GNN亲和力预测的准确性和相关性存在争议的观点,作者对GNNs在预测蛋白质-配体亲和力时究竟学到了什么感兴趣。因此使用不同类型的GNN系统地预测了亲和力。基于这些结果,作者应用了可解释人工智能(XAI)来分析预测。
研究概念和方法框架
作者用了六种不同的图神经网络(GNNs),系统地预测了蛋白质-配体亲和力,并解释了这些预测,以确定和比较GNN学习特性。这些不同架构的GNN包括图卷积网络(GCN),图注意网络(GAT),图同构网络(GIN)及其边包含变体(GINE),具有泛化激活函数的GNN(GraphSAGE)以及使用图卷积(GraphConv)操作符的另一种GNN,这里称为GC-GNN。这些网络被用来系统地预测基于从PDBbind数据库中获得的X射线结构和亲和力数据生成的蛋白质-配体相互作用图的训练(7,301个蛋白质-配体复合物)和验证(658个复合物)集合的亲和力。测试数据使用了2016年数据集合(186复合物,表1core set)以及2019年数据集合(2,542个复合物,表1hold-out set)。
对于可解释人工智能(XAI)分析,需要考虑相互作用图的信息内容。常用的相互作用图由连接配体伪原子(节点类型1)和蛋白质伪原子(节点类型2)的边组成。配体伪原子代表配体原子或形成分子内非共价相互作用的原子之间的中间位置,蛋白质伪原子代表氨基酸残基。相互作用边解释了不同类型的非共价分子间相互作用(氢键或疏水性和/或范德华相互作用)。评估边重要性是一种通用的方法,用于解释基于相互作用图的预测。节点重要性也可能被考虑。因此,为了合理化不同GNN模型的预测结果,作者首先应用了EdgeSHAPer,这是作者最近专门为GNNs开发的一种公开可用的XAI方法。EdgeSHAPer不依赖于GNN模型,可以量化图中边对GNN学习的重要性,并识别对个别预测最重要的边。这使我们能够直接确定哪些部分(子图)的相互作用图对模型决策负责。为了量化边重要性,EdgeSHAPer使用了Shapley additive explanation(SHAP),它来自合作博弈论的Shapley值概念。Shapley值最初用于量化个别玩家对团队性能的贡献。
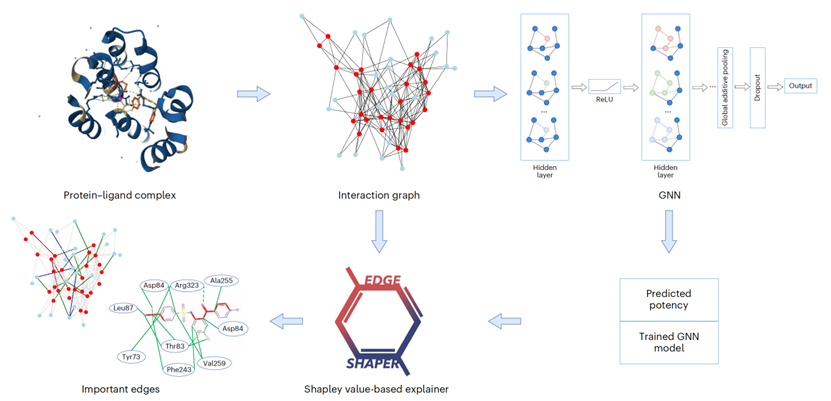
图 1
图1总结了分析的工作流程。相互作用图是从蛋白质-配体复合物的结构构建的。GNNs被使用训练和验证集的相互作用图进行评估,然后用来预测外部核心和保留集的数值亲和力(pKi)值。作为性能度量,计算了预测亲和力值与实验亲和力值之间的常规均方根误差(r.m.s.e.)。然后,使用EdgeSHAPer算法来量化边的重要性,并识别决定个别预测的边(及其对应的子图)。
实验结果
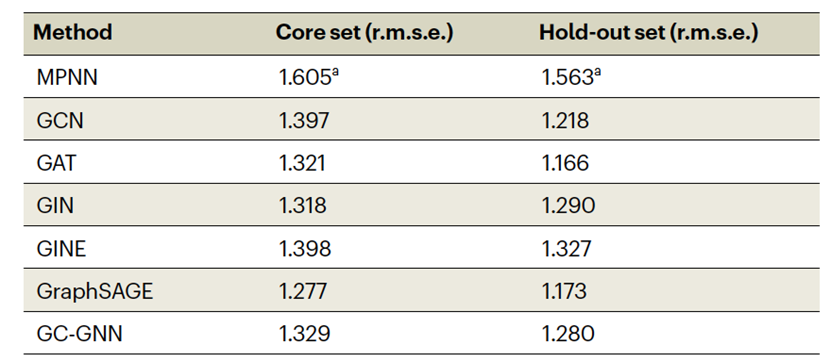
表 1
表1报告了与作为参考的MPNN相比的GNN模型的性能。对于GCN、GAT、GIN、GINE、GraphSAGE和GC-GNN,获得的均方根误差(r.m.s.e.)值低于参考MPNN报告的值。然而,这些模型之间的差异通常很小。对于保留测试集,模型之间最大的r.m.s.e.差异仅为0.397。此外,不同GNN架构之间的r.m.s.e.差异也非常小。对于保留集测试集,任何一对GNN模型之间最大的差异仅为0.161(GINE与GAT)。因此模型在基于相互作用图的亲和力预测中实现了可比的准确性,模型之间的差异非常小。对于我们的XAI分析,这是一个重要的方面,为解释在模型性能可比的情况下不同GNN变体的预测提供了坚实的基础。
模型解释
对于每个预测,作者使用EdgeSHAPer(方法)识别了k个最重要的边,即具有最高绝对SHAP值的前k个边缘。边属于三个不同类别,包括(1)配体伪原子之间形成的分子内边(称为配体边)、(2)蛋白质伪原子之间形成的分子内边(蛋白质边)和(3)配体和蛋白质伪原子之间形成的分子间边(相互作用边)。为了模型解释,保留集被用作测试集。然后单独分析了不同亲和力子范围内的测试实例预测,包括低亲和力(pKi < 6)、中等亲和力(pKi ∈ [6.5, 7.5])和高亲和力(pKi > 8)。分析的低、中和高亲和力子范围分别包含533、698和615个测试实例。
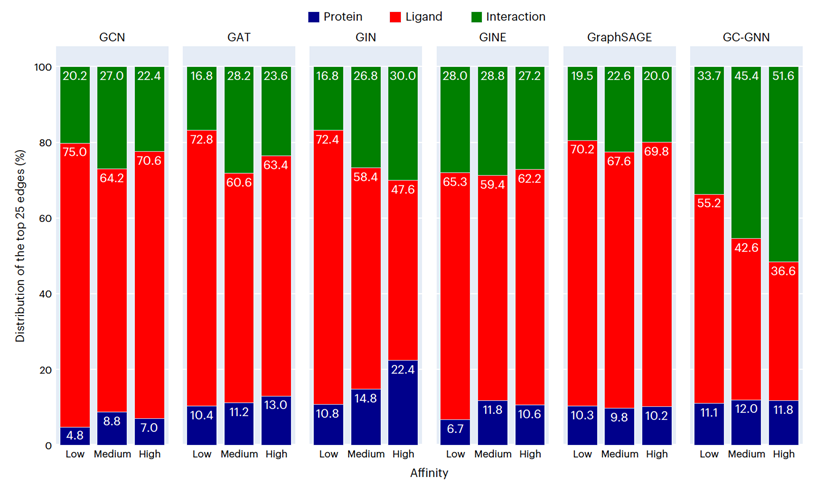
图 2
在所有相互作用图中(包括训练和测试数据以及落入不同效力子范围的测试实例),配体和蛋白质节点的比例大致分别为60%和40%(数据集之间的差异仅为1-2%)。平均而言,参与相互作用的配体和蛋白质节点的相对比例在所有相互作用图中分别为49%和51%。此外,分子间和分子内边的相对比例在所有亲和力子范围内几乎保持不变,大致分别为30%和70%。内边包括大约64%的配体边和6%的蛋白质边。因此,可用的配体边比蛋白质边多得多。比较配体与蛋白质伪原子的比例和配体与蛋白质边缘在相互作用图中的比例表明,配体子图比蛋白质子图连接更密集,这是符合预期的(因为每个蛋白质伪原子最多只能与另外两个连接,除非罕见的额外二硫键情况)。然后使用EdgeSHAPer识别了对所有预测贡献最大的边缘。图2比较了在确定不同GNN模型对三个亲和力子范围预测的前25个边缘,蛋白质、配体和相互作用边的比例。这些值反映了配体、蛋白质和相互作用边对亲和力预测的相对贡献。图2 中的结果显示了预测的清晰且一致的趋势。在不同的亲和力水平上,配体边主导了预测,平均约占前25个最重要的边缘的65%。相比之下,蛋白质边的贡献要小得多,通常只占前25个边缘的大约10%,这可能是因为图中蛋白质边的倾向性远低于配体边。然而,所有GNN模型也学习到了相互作用信息,大约有20%的最重要边是相互作用边,在六种GNN中有四种是这样。两种GNN在不同亲和力水平上显示出不同的边贡献比例。
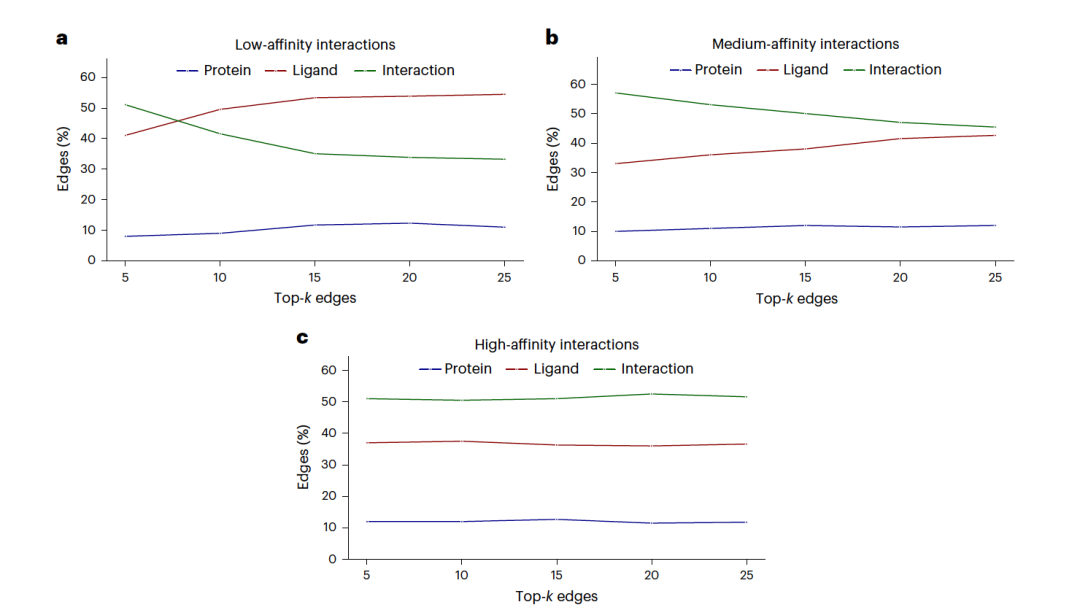
图 3
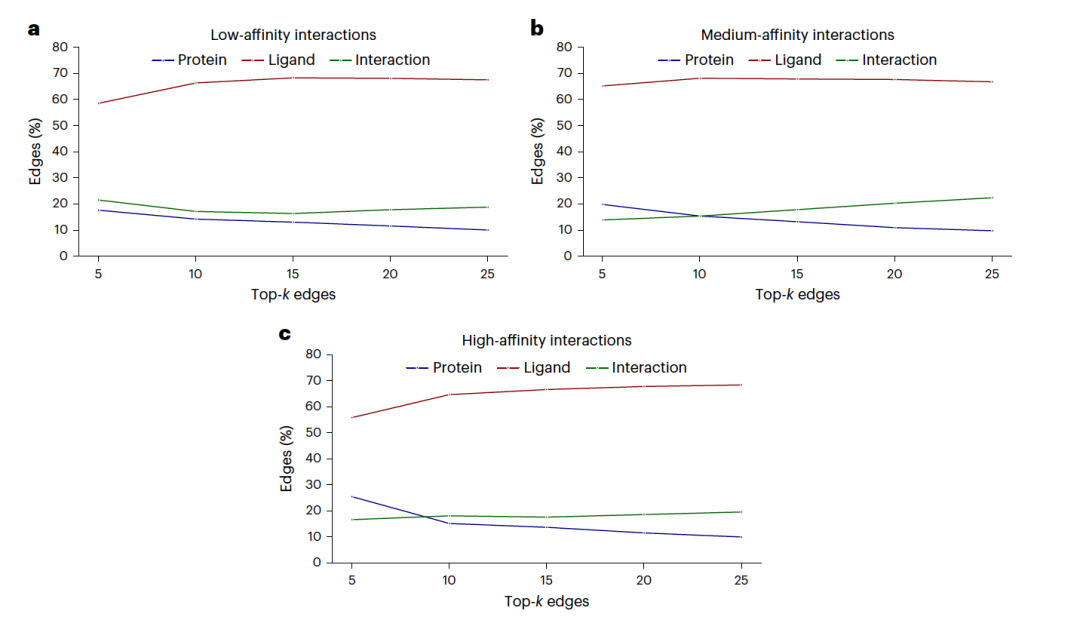
图 4
综合分析结果表明,不同类型的图神经网络(GNN)并没有一致地从图表中学习蛋白质-配体相互作用信息来进行精确的亲和力预测。然而,相互作用边被不同程度地优先考虑。此外,尽管不确定需要多少相互作用边才能正确预测给定的配体亲和力,结果显示配体记忆在预测中占主导地位。因此,GNNs通常可以在不优先考虑相互作用边和通过回忆相似训练化合物的亲和力来学习蛋白质-配体相互作用的情况下,合理准确地预测配体亲和力值。值得注意的是,随着亲和力的增加,不同GNN变体的蛋白质、配体和相互作用边的相对贡献观察到有所不同。最准确预测增加亲和力的贡献是GC-GNN。因此,这种GNN架构对包含相互作用信息以准确预测高亲和力最为敏感。作者进一步分析了GC-GNN与其他GNN的学习特性。图3详细说明了通过监测不同k值的边缘比例来观察GC-GNN预测的趋势。与图2的结果一致,其他网络的预测主要由配体边GNNs主导,如图4中以GraphSAGE为代表示例所示。在这里,配体边在所有亲和力水平上主导了预测,而相互作用和蛋白质边对预测的贡献较小。对于k值大于十的情况,相互作用边变得稍微比蛋白质边更重要。
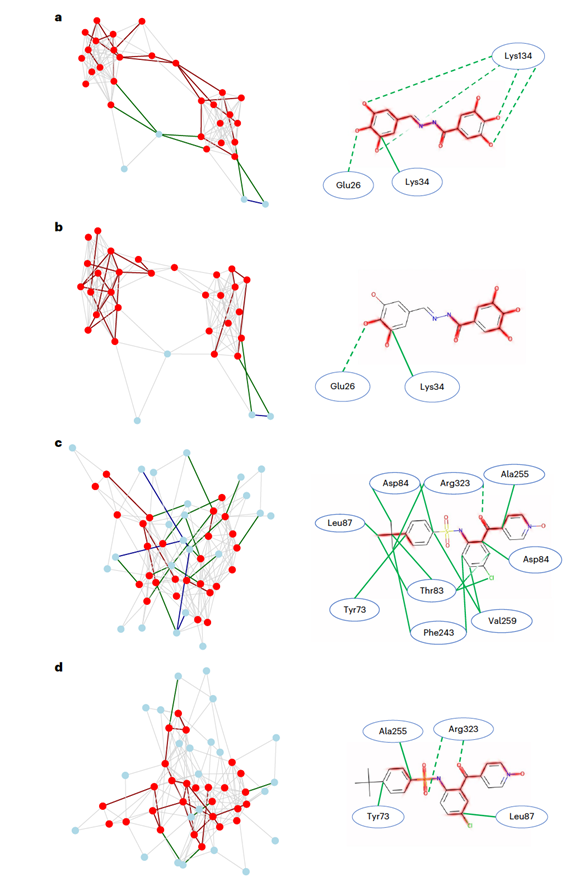
图 5
如图5所示,决定亲和力预测的边也可以从相互作用图映射回实际的蛋白质-配体相互作用。图5a展示了一个GC-GNN对低亲和力复合物(pKi = 5.06)预测的示例解释。在这种情况下,预测主要由配体记忆驱动,因为配体中几乎所有键相对应的边决定了预测。相互作用在这里起到了次要作用,主要集中在与几个配体原子处于氢键距离的Lys134侧链上。通过突出相互作用图中重要的边,并将边信息映射回蛋白质、配体和蛋白质-配体相互作用的结构层面,配体信息的主导地位已经显而易见。图5b展示了由GraphSAGE预测的同一相互作用的解释。
图5c和d分别使用GC-GNN和GraphSAGE解释了一个典型的高亲和力复合物(pKi = 9.96)的预测。可视化进一步说明了不同的学习特性。对于GC-GNN,预测主要由疏水性蛋白质-配体接触决定,而配体贡献只起到了次要作用。相比之下,对于GraphSAGE,配体边再次主导了预测,而相互作用边的贡献非常小,与GC-GNN优先考虑的不同。
编译 | 曾全晨
审稿 | 王建民
参考资料
Mastropietro, A., Pasculli, G. & Bajorath, J. Learning characteristics of graph neural networks predicting protein–ligand affinities. Nat Mach Intell 5, 1427–1436 (2023).
https://doi.org/10.1038/s42256-023-00756-9



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


