
编译 | 裴英豪
审核 | 王永康
今天给大家分享CMU马坚教授发表在Bioinformatics上的一篇论文‘UNADON: transformer-based model to predict genome-wide chromosome spatial position’。染色体相对于细胞核结构的空间定位与基因组功能密切相关。然而,影响染色质在全基因组范围内的空间定位的序列模式和表观基因组特征尚不为人们充分理解。本文作者提出了一个名为UNADON的基于Transformer的深度学习模型,该模型利用序列特征和表观基因组信号,预测了基于TSA-seq测得的染色体到特定类型核小体的全基因组细胞学距离。对UNADON在四个细胞系中的评估显示,在单一细胞系的训练下,该模型在预测染色质相对于核小体的空间定位方面表现出高准确性。UNADON在未见过的细胞类型中也表现良好,展现了模型良好的泛化能力。重要的是,研究人员揭示了影响核小体中大规模染色质分隔的潜在序列和表观基因组因素。总的来说,UNADON为理解序列特征和大规模染色质空间定位之间的原理提供了新的见解,这对于理解核结构和功能具有重要意义。
1 简介
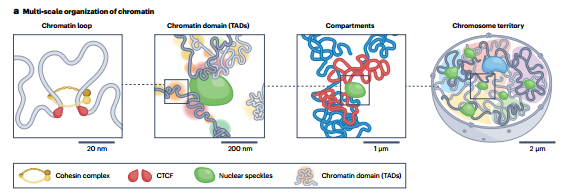
细胞核是高等真核生物的一种异质的细胞器,包含与关键细胞功能相关的独特亚核结构和核体。例如,核小体(nuclear speckle)调节基因的转录和剪接,核纤层将染色质固定到核的外围。染色质在细胞核内折叠包装,它的结构有重要作用,比如基因的表达水平和到核小体的距离负相关,核纤层经常抑制染色质相互作用。
近年来,深度学习模型在预测功能基因组和表观基因组特征方面取得了显著进展。先前的方法中,使用卷积神经网络层扩展感受野,整合了长达1Mb的DNA序列,但可能忽略大于10Mb的空间定位依赖。最新方法Orca通过大型CNN和级联预测机制将最大输入序列长度扩展到整个染色体,但仍然受到感受野大小的限制。此外,很少有模型能有效结合基因组序列和表观基因组特征,实现跨细胞类型的预测。
最近提出的一种名为TSA-seq的测序方法测量了从染色体位点到核体的细胞学距离,从全基因组的角度为探明染色体相对于特定核体的空间定位提供了机会。本文作者引入了一种名为UNADON的新型多模态Transformer深度学习模型。UNADON旨在基于DNA序列和表观基因组信号预测染色质相对于核小体的空间定位。该研究的主要创新在于:(i) UNADON是一个专门设计用于预测染色质相对于核小体空间定位的深度学习模型;(ii) 其独特的神经架构设计使UNADON能够更有效地学习长程依赖关系;(iii) UNADON在跨细胞类型的预测中表现出很好的泛化性,可用于推断新细胞类型中的空间定位;(iv) 对UNADON的解释揭示了瞄准核小体的潜在机制。总体而言,UNADON为染色体空间定位的预测和解释建立了一个新颖的计算框架。
2模型介绍
2.1网络架构
UNADON是一个基于Transformer的深度学习框架,它根据DNA序列和表观基因组信号预测TSA-seq数据。具体而言,为了预测一个大小的基因bins的TSA-seq,模型考虑的上下文信息。来自窗口里经过处理的k-mers序列特征和表观基因组特征被传递到两个独立的特征提取子网络,该网络由具有ReLU激活的密集层组成。然后把序列和表观基因组的特征进行拼接,送到Transformer模块中。之后将Transformer层的输出送入带有正切激活函数的密集层里,对TSA-seq进行预测。
为了进行跨细胞类型的预测,本文作者附加了一个领域自适应模块,以防止模型在训练集上过拟合。这个领域自适应模块是一个额外的分类器,用于识别嵌入中的源细胞类型。前向传播利用嵌入中的细胞类型特定信息来预测源细胞类型。反向传播包含一个梯度反转层,将流回Transformer的梯度反转。因此,模型将最大化细胞类型预测的损失,从而阻止学习任何可用于区分细胞类型的特定于细胞类型的特征。模型总体架构如下图所示:
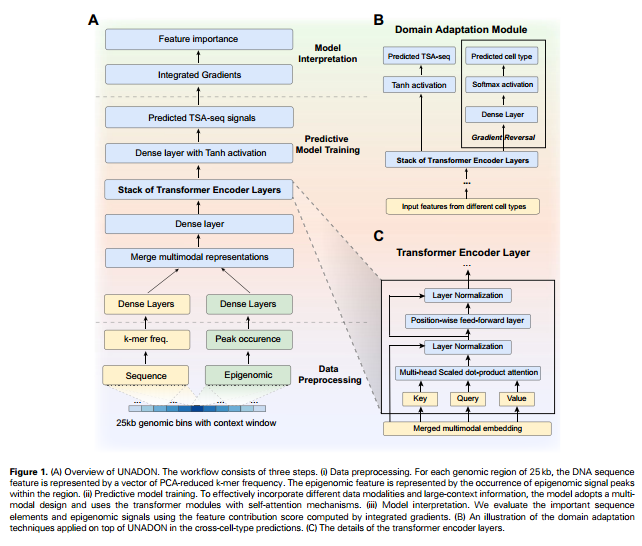
2.2模型训练和评估
在所有实验中,采用了跨染色体评估方法,利用奇数染色体进行训练和验证,偶数染色体则保留用于测试。在寻找最佳超参数时,进行了交叉验证,每次把一个奇数染色体作为验证集。
模型被训练以最小化预测TSA-seq信号的均方误差。进行两种类型的评估来评估模型性能和泛化能力:单细胞类型预测和四种人类细胞系(K562、H1、HCT116、HFFc6)上的跨细胞类型预测。对于单细胞类型预测,模型在特定细胞类型的序列和表观基因组特征上进行训练。对于跨细胞类型预测,一种细胞类型在训练过程中被保留,以评估模型在真实世界中对未见细胞类型的性能。模型在三种细胞类型的奇数染色体上进行训练,并在保留的细胞类型的偶数染色体上进行测试。
除了提出的UNADON模型外,还训练和评估了四个机器学习模型进行性能比较:XGBoost、稠密神经网络、卷积神经网络和扩张卷积神经网络(Dilated CNNs)。所有模型应用相同的输入特征和上下文长度。机器学习算法使用scikit-learn库实现,而基于神经网络的模型使用PyTorch实现。对于跨细胞类型的预测,还评估了额外基线模型,该基线模型利用现有细胞类型的平均信号作为未见细胞类型的估计,考虑到染色体的空间定位在细胞类型间共享相似性。
2.3模型训练和评估
为揭示输入特征对最终预测的贡献,研究团队应用了综合梯度来分别推导序列和表观基因组信号的位点特异性重要性分数。综合梯度是一种基于梯度的特征归因方法,根据输出相对于输入特征的梯度计算重要性分数。直观上,如果从基线偏离一点导致输出发生较大变化,该特征应对预测具有重要性。综合梯度考虑了输入和基线之间的直线路径。重要性分数由该路径上所有点的梯度累积定义。作者将全零向量作为基线。由于综合梯度计算的重要性分数可以根据特征对预测的影响而为正或为负,作者取重要性分数的绝对值作为特征贡献。为了量化DNA序列特征的总贡献,通过对所有维度的重要性分数求和,作者汇总了来自所有维度的贡献。
2.4数据收集与处理
研究团队收集了来自四种人类细胞系(K562、H1、HCT116、HFFc6)的TSA-seq数据的染色体到核小体的全基因组映射。采用了相同的归一化过程,将归一化的读数转换为TSA-seq富集分数。对于不重叠的25 kb基因组区段,作者计算了TSA-seq分数,并将信号缩放在-1到1之间。为了减轻技术噪声的影响,作者使用大小为21的Hanning窗口对TSA-seq信号进行了平滑处理。更高的信号表示距离特定类型核小体更近。作者使用了人类参考基因组GRCh38。删除了低映射性的基因组区域,例如着丝点区域。为了减少DNA序列的维度,作者使用k-mer频率来表示序列,将k-mers与它们的反向互补物合并,以考虑DNA的正向和反向链。我们连接了5和6的k-mer频率,并应用PCA将k-mer频率向量的维度降低到20。
为了捕捉细胞类型特异性的染色质空间定位,我们整合了广泛可用的表观基因组特征。具体而言,作者汇总了来自不同测定和数据库的染色质可及性和八种组蛋白修饰(H2A.Z、H3K4me1、H3K4me2、H3K4me3、H3K9me3、H3K27me3、H3K27ac、H3K36me3)。作者使用由ENCODE峰值调用管道调用的信号峰值来表示25 kb基因组区域内的表观基因组信号。对于每个单独的区域,作者计算位于信号峰值下的碱基数,并通过区域长度进行归一化。为了考虑潜在的与技术相关的偏差,作者通过整体基因组上的平均峰值频率对峰值出现进行进一步的归一化。
3结果
3.1UNADON 准确预测单个细胞类型中染色质相对于核体的空间定位
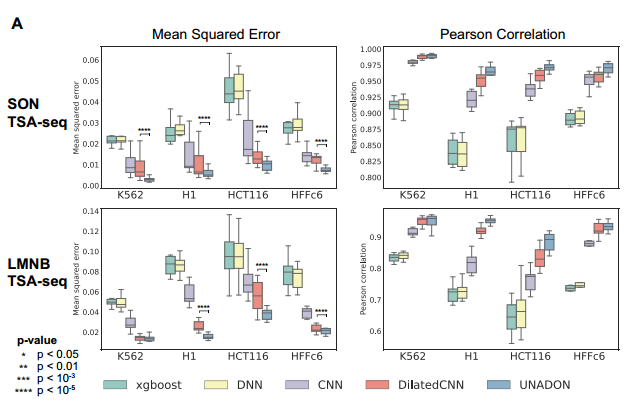
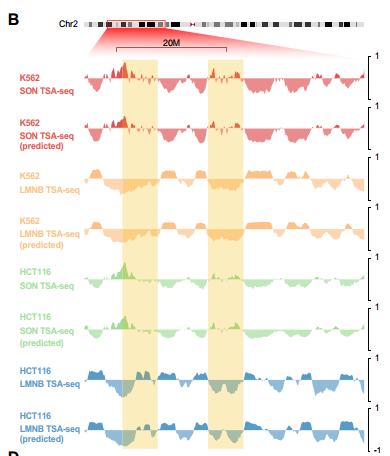
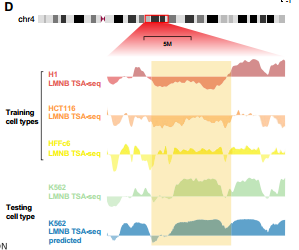
UNADON在个体细胞类型中准确预测相对于核小体的染色质空间定位。研究团队使用UNADON来预测染色体相对于核小体(通过SON TSA-seq测量)和核膜(通过LMNB TSA-seq测量)在四种人类细胞系中的空间定位。上图显示了预测信号与实际TSA-seq轨迹的比较示例。
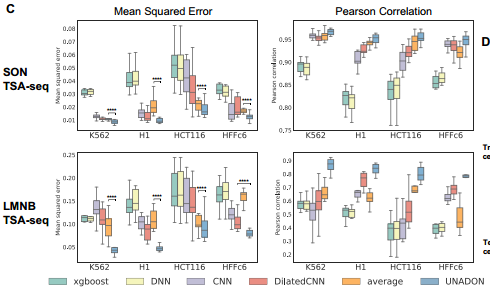
研究团队通过均方误差和皮尔逊相关系数比较了UNADON与四种常用的基线方法(XGBoost、DNNs、CNNs和Dilated CNNs)的性能。发现UNADON在所有细胞类型和核小体中始终优于基线模型的性能。值得注意的是,UNADON比最先进的扩张CNN实现了更好的预测性能,进一步证明了其注意力机制在捕捉基因组特征与空间定位关系方面的优势。需要注意的是,UNADON在细胞类型之间的预测性能有所差异,尤其是在LMNB TSA-seq上。HCT116 LMNB TSA-seq的PCC仅为0.88,而K562 LMNB TSA-seq为0.95。这种差异表明细胞类型可能包含用于大规模染色体定位的不同序列和表观基因组特征,增加了跨细胞类型预测的难度。
3.2UNADON 可以预测未曾见过的细胞类型中染色质的空间定位
在真实世界中,模型可能会面对多种细胞类型的数据。一个具有强大泛化能力的模型可以更广泛地应用于不同细胞类型的染色质空间定位任务。作者将可用的数据集分为不同的细胞类型。在进行交叉细胞类型评估时,每次都选择其中一种细胞类型作为测试集,而其他三种细胞类型作为训练集。为了对比,作者引入了一个基线方法,命名为’average’。该方法简单地使用在训练细胞类型中的TSA-seq平均值作为预测。这个基线方法不仅提供了对比参考,还作为测试细胞类型与训练细胞类型之间空间定位相似性的度量。对模型性能的评估采用了均方误差和皮尔逊相关系数。评估指标用于量化模型在预测中的准确性和与实际观测值的一致性。结果如下图所示,
3.3UNADON 揭示了序列和表观基因组特征的特征重要性
作者采用了整合梯度方法,探究了DNA序列和表观基因组特征在四种不同细胞类型中预测SON TSA-seq和LMNB TSA-seq的基因组范围内影响。结果显示,整体而言,DNA序列是最关键的预测因素,其次是ATAC-seq和H3K4me1。特别是在预测LMNB TSA-seq时,相比于SON TSA-seq,DNA序列的贡献相对较不显著,这表明核边缘朝向染色体的定位机制可能对序列特征的依赖性较低。
在跨细胞类型的设置中,表观基因组特征的重要性变得更为明显,表明相比于通用的序列特征,模型需要细胞特异性的表观基因组信息进行预测。为了更好地理解不同特征的整体贡献,作者采用了k-means聚类,聚焦于来自LMNB TSA-seq的贡献分数。结果显示出六种不同的模式,每一种模式都以不同比例包含序列和表观基因组特征的贡献。特别地,未检测到低序列和高表观基因组贡献的簇,进一步强调了序列特征的重要性。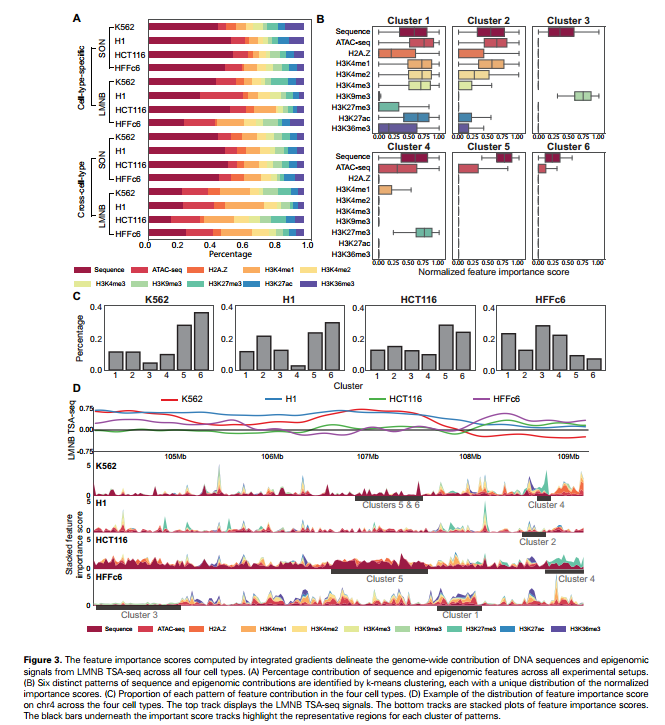
4结论
本文作者作者提出了一种名为UNADON的新型深度学习方法,用于预测染色体相对于核小体的空间定位,并探究了在不同细胞类型中调控这种空间定位的潜在序列和表观基因组决定因素。UNADON采用了Transformer模型和多模态设计,将多组学数据整合到一个较大的上下文中。凭借其独特的神经架构设计,UNADON在个体细胞类型上表现出强大的预测性能,并能准确泛化到未曾见过的细胞类型。。总体而言,UNADON建立了一个新颖的框架,用于预测和解释染色体相对于核小体的定位,可扩展到任何基于大规模序列的预测建模和分析。
关于论文的更多细节可从原文获取
论文链接:https://academic.oup.com/bioinformatics/article/39/Supplement_1/i553/7210492
代码链接:https://github.com/ma-compbio/UNADON



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


