点击下方卡片,关注「AI视界引擎」公众号

自动化分割不同肿瘤区域对于儿科脑肿瘤的准确诊断和治疗计划至关重要。本研究评估了多规划U-Net(MPUNet)方法在分割三个具有挑战性的数据集(儿科肿瘤挑战赛(PED)、脑转移挑战赛(MET)和撒哈拉以南非洲成人胶质瘤(SSA))中的不同肿瘤亚区域的有效性。这些数据集代表了多样化的场景和解剖学变化,使其适合评估MPUNet模型的鲁棒性和泛化能力。通过利用多平面信息,MPUNet架构旨在提高分割准确性。
结果显示,评估的挑战中性能水平各异,其中肿瘤核心(TC)类别的分割准确性相对较高。然而,其他类别的分割,如水肿和增强肿瘤(ET)区域,存在变化。这些发现强调了脑肿瘤分割的复杂性,并突显了进一步优化MPUNet方法和包括MRI更多数据和预处理的潜力。
1 Introduction
自动化肿瘤分割对于手术计划、治疗评估和长期监测至关重要。手动分割耗时且易变。儿科脑肿瘤具有多种特征,如可变 aggressive、预后和异质性组织亚区域,这使得评估具有挑战性。尽管罕见,但儿科中枢神经系统肿瘤是儿童癌症相关死亡的主要原因。与成人脑肿瘤不同,儿科脑肿瘤具有独特的影像和临床表现。例如,儿科弥漫性中间线胶质瘤(DMGs),包括弥漫性内囊髓母细胞瘤(DIPG)亚型,是高级胶质瘤,与成人胶母细胞瘤(GBMs)具有相似的平均总体生存率。然而,GBM的发病率是每10万人中的3例,而DMGs则更为罕见。GBM通常在64岁的平均年龄前发现在额叶或颞叶,而DMGs则常位于脑桥,并在5至10岁之间进行诊断。由于儿科脑肿瘤的独特特征和挑战,需要特定的影像工具进行特征和诊断/预后。
一些肿瘤,如弥漫性内囊髓母细胞瘤(DIPGs),位于无法手术切除的区域,因此需要依赖纵向扫描的大小变化来评估进展。目前的标准使用2D线性测量,但这些测量不准确且增加了操作者之间的变异性。在成人脑肿瘤的研究中,3D体积测量在预测临床结果方面具有优势。虽然体积肿瘤测量在评估儿科脑肿瘤方面正在获得认可,但分割肿瘤亚区域的自动化工具有限。存在少数方法,主要集中在T2 FLAIR异常信号分割,但缺乏对同一数据集的评价和比较,导致儿科脑肿瘤自动化分割工具的基准测试存在缺口。
在过去的11年里,MICCAI脑肿瘤分割(BraTS)挑战赛为成人胶质瘤创建了一个基准数据集和社区。今年的BraTS将焦点扩展到包括一系列挑战,涵盖了不同的肿瘤实体、缺失数据以及技术方面。值得注意的是,BraTS 2022挑战赛首次在测试阶段包括了儿科脑肿瘤,特别是弥漫性内囊髓母细胞瘤(DMGs)。
深度学习技术在计算机视觉的各个领域取得了令人瞩目的成功,包括识别自然图像、目标检测和图像分割。这种主导地位也延伸到医学图像分割任务上。最初用于生物医学图像分割,U-Net架构已经经过多次修改。例如,提出了U-Net的3D版本,然后引入了V-Net,它采用了残差块和软 dice 损失。另一个改进涉及将注意力模块引入到U-Net模型中。
除了U-Net之外,研究行人还探索了其他架构。一些研究将3D体积切成2D部分并使用2D分割网络处理。另一种混合方法出现,其中采用了基于ResNet50的2D编码器并增加了3D解码器。此外,[19]通过3D网络将2D预测融合以增强具有上下文信息的预测。然而,U-Net派生的架构在这个领域仍然保持其优势。最近的发展是MPU-Net的引入[18],该系统在2018年的医学分割Decathlon的第一轮和第二轮比赛中分别获得了第五和第六名。
该系统利用多平面数据增强,使系统能够利用单个类似于知名U-Net设计的2D架构。通过多平面训练,系统结合了2D全卷积神经网络的效率和训练和测试阶段结构的增强策略。这种方法使2D模型能够获得3D图像卷的综合表示,从而提高其有效泛化能力。在本研究中,作者使用了MPU-Net架构进行所有分割任务。
2 Methods
本研究中采用的核心方法围绕多规划U-Net(MPUNet)架构(参见图1),该架构作为作者肿瘤分割方法的基础。MPUNet是U-Net架构的一个修改版本,它是一种专为图像分割任务设计的卷积神经网络。MPUNet引入了多平面训练策略,即输入图像沿各种轴旋转,生成图像的多个视角。这种技术使模型能够发展出对3D图像卷的全面表示,从而增强其泛化和适应不同解剖变异和肿瘤表现的能力。
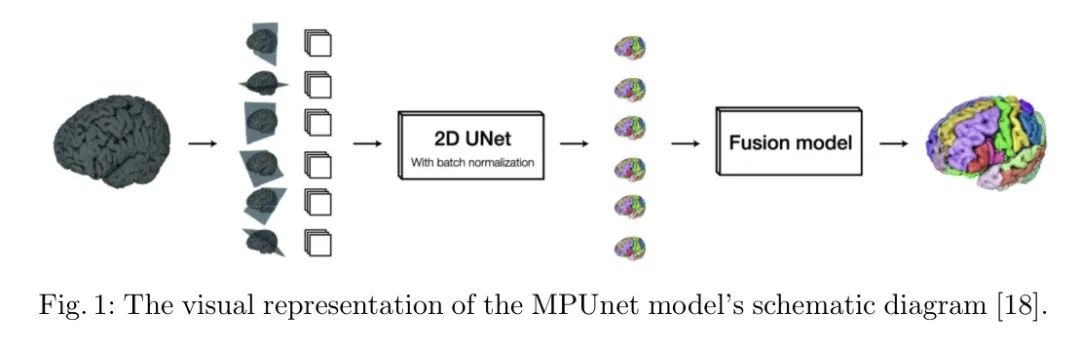
本研究采用了三个不同的挑战:儿科肿瘤挑战赛(PED)、脑转移挑战赛(MET)和撒哈拉以南非洲成人胶质瘤挑战赛(SSA)。这些挑战涵盖了各种肿瘤类型、解剖变异和成像场景,为MPUNet的鲁棒性和泛化能力提供了全面的评估。
为了确保可靠的评估,采用了严格的3倍交叉验证技术。数据集被随机分为三个子集:训练集、测试集和验证集。这个划分重复了三次,有效地覆盖了数据集,同时最小化了偏差的影响。在每个折叠中,两个子集用于训练模型,一个用于评估模型的性能,剩下的子集用于验证目的。这种方法使模型能够在数据集中的不同案例之间进行全面的泛化评估。
Preprocessing
为了应对GPU内存的限制,作者采用了战略性的方法,即只利用所有四种成像模式中的前三种(Pediatrics Tumor Challenge (PED)、Brain Metastasis Challenge (MET)和Sub-Sahara-Africa Adult Glioma Challenge (SSA))进行所有三个挑战。这种模式的选择利用旨在在确保内存约束和保留准确肿瘤分割所需的基本信息之间实现平衡折衷。
Statistical measurement
为了评估作者生成的分割Mask的精度,作者采用了Dice分数矩阵作为所有三个不同类别(ET(增强肿瘤)、TC(肿瘤核心)和WT(整个肿瘤))的主要评估指标。在图像分割领域,Dice分数衡量了作者模型预测的分割Mask与参考真实Mask之间的相似程度。这个确立的度量标准使作者能够有效地衡量作者的分割结果与Pediatrics Tumor Challenge (PED)、Brain Metastasis Challenge (MET)和Sub-Sahara-Africa Adult Glioma (SSA)数据集中的实际肿瘤边界之间的匹配程度。
Results
MPUNet的性能结果显示了在三个不同的挑战(PED、MET和SSA)的验证上的Dice分数的平均值和标准差。Dice分数报告了三个类别:ET(增强肿瘤)、TC(肿瘤核心)和WT(整个肿瘤),分别代表了分割质量的不同方面。
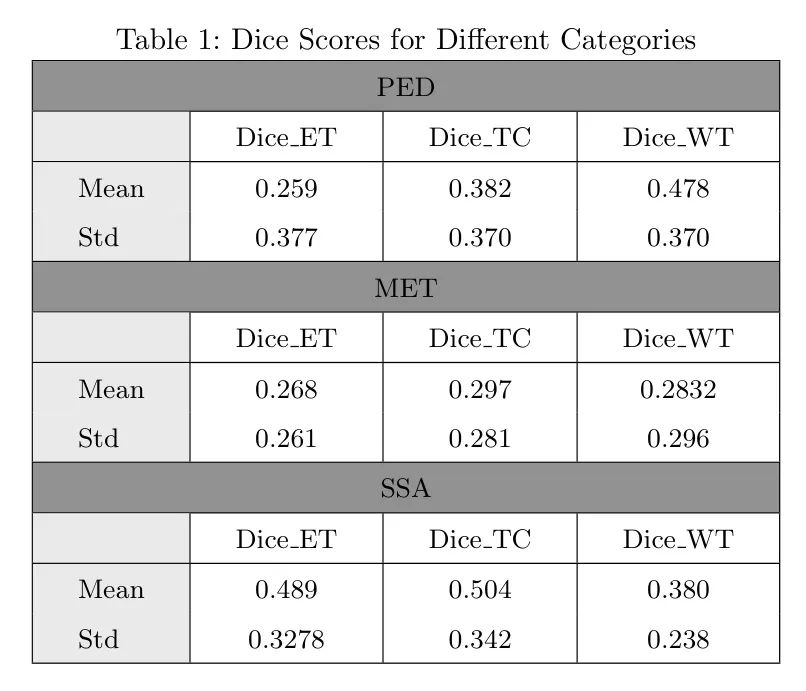
在MET挑战中,MPUNet在ET、TC和WT三个类别上的平均Dice分数分别为0.268、0.297和0.283,标准差分别为0.261、0.287和0.296。在这里,三个类别的性能似乎相对平衡,最低的平均分数是WT。然而,标准差仍然适度,表明性能存在一定的变异性。
在SSA挑战中,MPUNet在ET、TC和WT三个类别上的平均Dice分数分别为0.489、0.504和0.38,标准差分别为0.327、0.342和0.238。这些结果表明,模型在TC类别上表现相对良好,在该挑战中获得了最高的平均分数。WT类别的较低平均分数可能表明在特定类别分割方面存在改进的空间。
总体而言,MPUNet在不同挑战和类别上的性能水平存在差异,有些挑战的表现比其他挑战更稳定。在某些情况下,较高的标准差突显了模型分割预测的鲁棒性的重要性,表明可能需要进一步优化和微调以提高整体分割精度,尤其是在平均Dice分数较低的类别中。
3 Discussion
MPUNet架构在跨越不同数据集对不同肿瘤亚区域进行分割的应用,展示了在自动化肿瘤分割中提高准确性的潜力。作者的方法,结合多平面信息,通过旨在捕获这些肿瘤中存在的异质性和解剖变异,解决了儿科脑肿瘤分割的复杂性。
在Pediatrics Tumor Challenge (PED)、Brain Metastasis Challenge (MET)和Sub-Sahara-Africa Adult Glioma (SSA)数据集上的评估显示了不同的分割性能水平。肿瘤核心(TC)类中观察到的相对较高的准确性表明了MPUNet方法在捕捉肿瘤中心区域的潜力。然而,其他类别的分割,如水肿和增强肿瘤(ET)区域,表现出了更多的变异性。这种变异性可能源于不同肿瘤类型和成像模态所特有的独特特性和复杂性。
儿科脑肿瘤的复杂性在跨越不同数据集进行分割的挑战中进一步凸显。肿瘤外观、大小和位置的固有变异性使得实现一致的分割结果变得艰巨。此外,尽管MPUNet架构利用多平面信息来增强分割,但在捕捉某些肿瘤亚区域的复杂细节方面可能会遇到困难,从而导致性能的变异性。
这些结果强调了需要继续改进和增强分割方法,特别是在处理具有挑战性的病例,如儿科脑肿瘤的情况下。整合更全面的数据,探索先进的预处理技术,并对特定肿瘤类型和亚区域进行微调MPUNet架构,可能潜在地导致分割精度和一致性的提高。
4 Conclusion
总之,本研究对MPUNet方法在儿科脑肿瘤跨数据集分割不同肿瘤亚区域的评估,突显了其在提高准确性方面的潜力,尤其是在捕捉肿瘤核心区域。然而,在分割像水肿和增强肿瘤等类的其他类别的变异性,强调了儿科脑肿瘤分割的复杂性。
该方法的依赖性多平面信息,展示了其对解剖变异和多样化肿瘤外观的适应性。Pediatrics Tumor Challenge (PED)、Brain Metastasis Challenge (MET)和Sub-Sahara-Africa Adult Glioma (SSA)数据集上的挑战,强调了需要持续改进,包括整合全面数据、先进预处理和针对特定肿瘤类型和亚区域的微调架构,以进一步提高分割精度,并推动儿科脑肿瘤的自动化分割领域的发展。
参考
[1]. Fully Automated Tumor Segmentation for Brain MRI data using Multiplanner UNet.

点击上方卡片,关注「AI视界引擎」公众号



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


