今天为大家介绍的是来自Fang Bai团队的一篇论文。随着人工智能技术的不断发展,越来越多用于生成新分子的计算模型被开发出来。然而,我们常常面临一个问题:这些化合物是否容易合成,这涉及到化合物的合成可及性。在这项研究中,提出了一个基于深度学习的计算模型,名为DeepSA,用于预测化合物的合成可及性,为选择分子提供了一个有用的工具。

随着人工智能技术的不断发展,基于各种人工智能算法,越来越多的计算分子生成模型被开发出来,包括基于流的生成模型和扩散模型等。这些模型可以为现有疾病的已知靶标生成新的命中化合物,并优化现有的先导化合物结构。它们帮助药物化学家从庞大的化学空间中找到有用的分子,缩短药物发现和开发的时间。然而,大多数由生成模型创建的新分子在合成可及性方面面临重大挑战。合成可及性预测可以看作是一个需要大量数据的复杂问题,机器学习十分适合处理这类问题。大量的分子及其预定义的“合成可及性得分”数据集是用机器学习研究该问题的先决条件。然后我们就可以设计一个人工智能模型来学习分子结构与其合成可及性之间的关系。
在这项研究中,作者提出了一种新的模型DeepSA,用于评估化合物合成可及性。这个模型能够以更高的准确率区分易于合成和难以合成的化合物。作者将DeepSA与其他现有评估化合物合成可及性的模型(GASA、SYBA、RAscore、SCScore和SAscore)的区分能力进行了比较。结果显示,DeepSA的性能表现特别好,因为它能更准确地评估现有研究报告中真实药物分子的合成难度。
DeepSA的架构
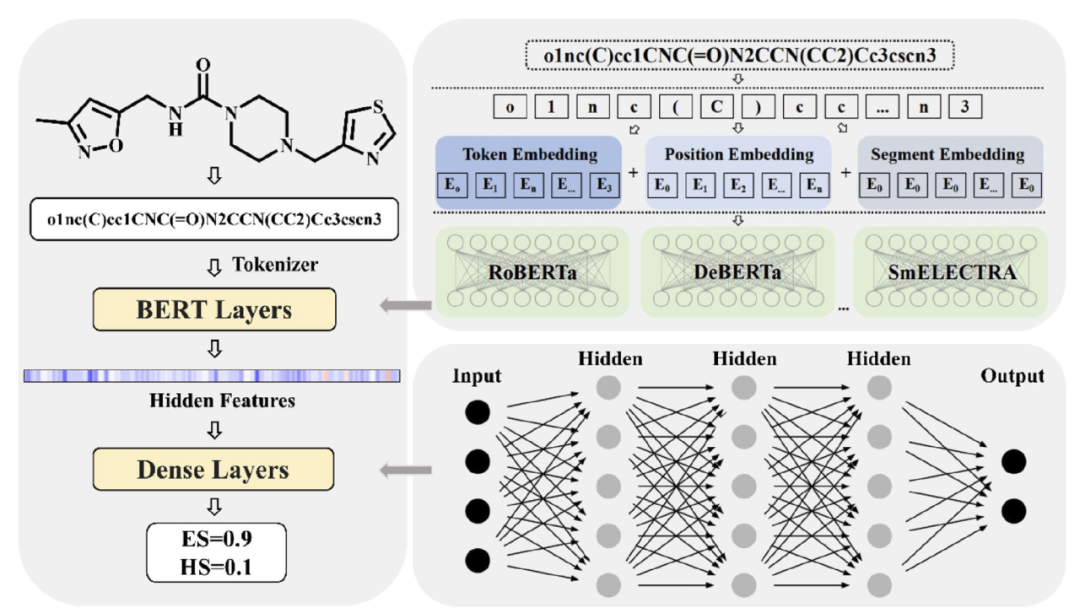
图 1
作者提出的DeepSA模型包含三个模块:数据处理模块、特征嵌入模块和解码器模块。DeepSA的架构如图1所示。作者将数据集中的原始SMILES转换为标准SMILES,然后通过RDKit引入了一些随机生成的非标准SMILES,从而进一步扩展了数据集。数据增强后的训练和测试数据集的最终大小分别为3,593,053和399,216。同时,使用了HuggingFace tokenizer库中的Byte-Pair Encoder(BPE)对化合物的结构进行编码,这可以将输入SMILES中的基本原子和环结构视为“单词”或“句子”,并使用之前研究人员提出的ChemBERTa SMILES-Tokenizer进行有意义的预测。首先,作者收集了来自不同自然语言模型的多种网络架构,包括bert-mini (MinBert)、bert-tini (TinBert)、roberta-base (RoBERTa)、deberta-v3-base (DeBERTa)、Chem_GraphCodeBert (GraphCodeBert) 和 electra-small-discriminator (SmELECTRA),以及两种化学语言模型,包括ChemBERTa-77M-MTR (ChemMTR) 和 ChemBERTa-77M-MLM (ChenMLM)。随后,作者基于这些不同的自然语言模型和两种化学语言模型使用设计的分子合成可及性数据集训练DeepSA模型。这些自然语言模型的架构针对合成可及性预测任务进行了微调。在微调阶段使用了AutoGluon软件包。学习率设置为0.001,并在训练过程中通过余弦衰减调度进行调整。训练过程最多进行20个周期,每0.2个周期进行一次验证,并存储在验证集上ACC最高的最多三个检查点模型。最后,通过融合了前三个表现最佳的模型,生成了最终的DeepSA模型。
DeepSA模型以预测分子的合成可及性
近年来,自然语言处理技术的发展催生了众多自然语言模型,为像自然语言一样处理蛋白质序列数据提供了一系列框架。直观上,用于表示化合物的SMILES序列与自然语言有一定的相似性。两者都由简单字符的多样词汇组成,并通过简单规则生成复杂的句子。这种相似性激发了研究人员将自然语言模型的框架转移到化合物数据上,目的是训练化学语言模型,例如SMILES-BERT,以提高化合物相关任务的性能。因此作者旨在探索类似于自然语言中文本任务的训练策略是否可以应用于评估分子合成可及性的化学语言模型,这将进一步加深我们对化学语言模型和化合物合成可及性的理解。
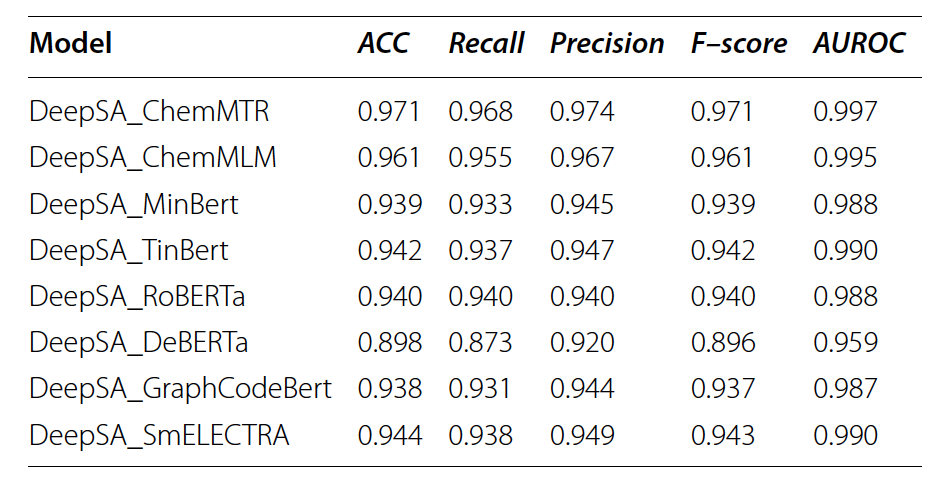
表 1
在这项研究中,作者设计了一个用于预测分子合成可及性的化学语言框架,作者尝试了各种语言模型作为编码层,如bert-mini (MinBert)、bert-tini (TinBert)、roberta-base (RoBERTa)、deberta-v3-base (DeBERTa)、Chem_GraphCodeBert (GraphCodeBert)、electra-small-discriminator (SmELECTRA)、ChemBERTa-77M-MTR (ChemMTR) 和 ChemBERTa-77M-MLM (ChenMLM)。DeepSA在测试集上的性能显示在表1中。DeepSA的测试结果上有高ACC、Recall、Precision和F分数。模型的高精度降低了将易合成化合物(ES)误判为难合成(HS)的风险,提高了决策准确性。令人印象深刻的是,几乎所有模型的AUROC值都高于0.98。
与其他模型的比较
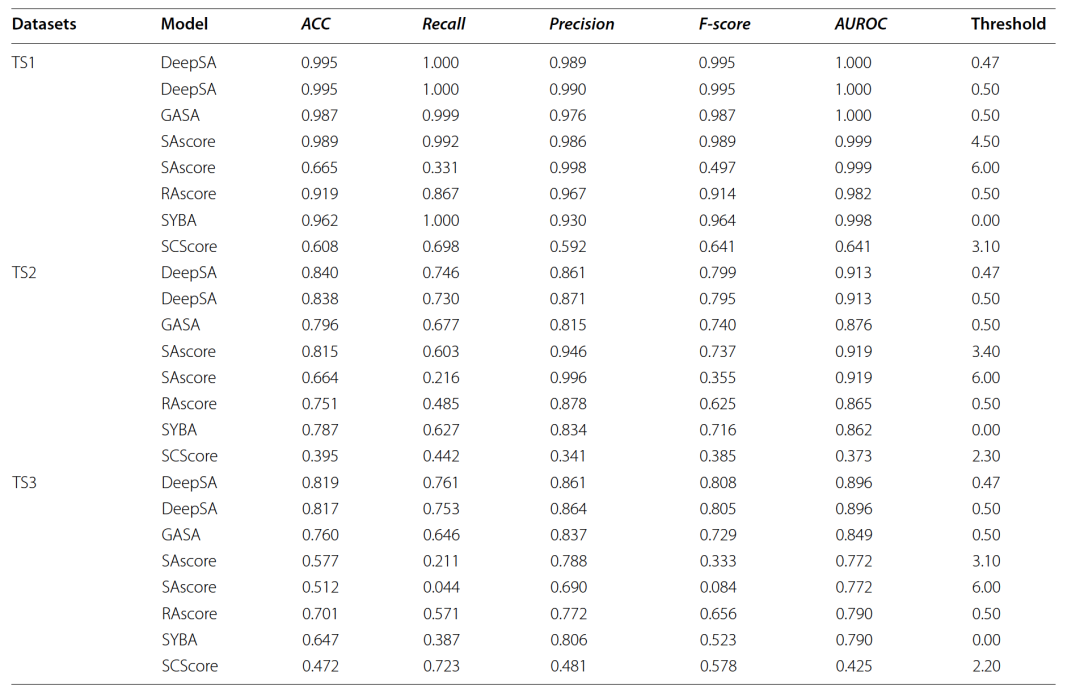
表 2
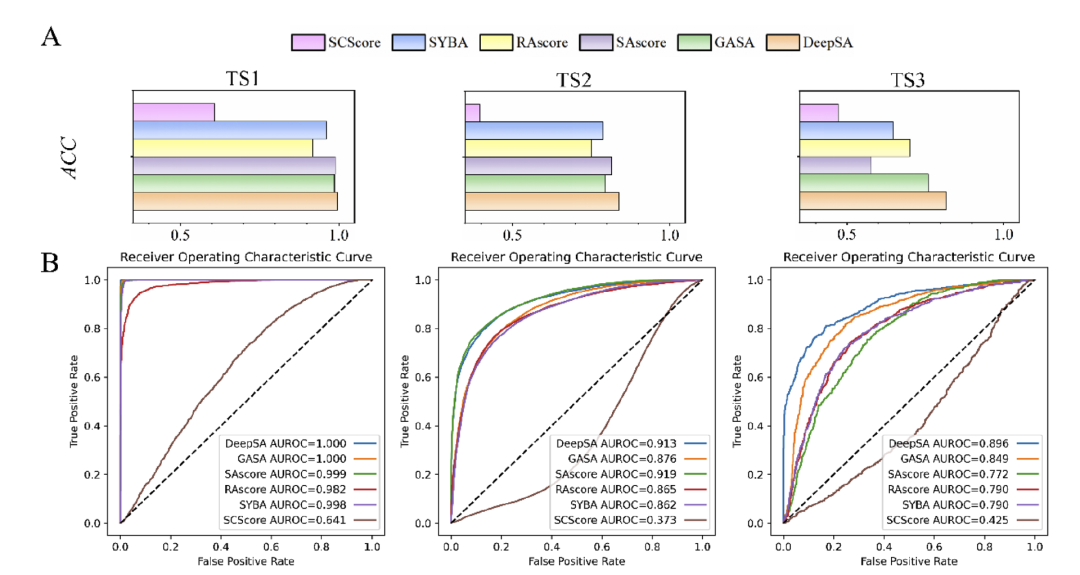
图 2
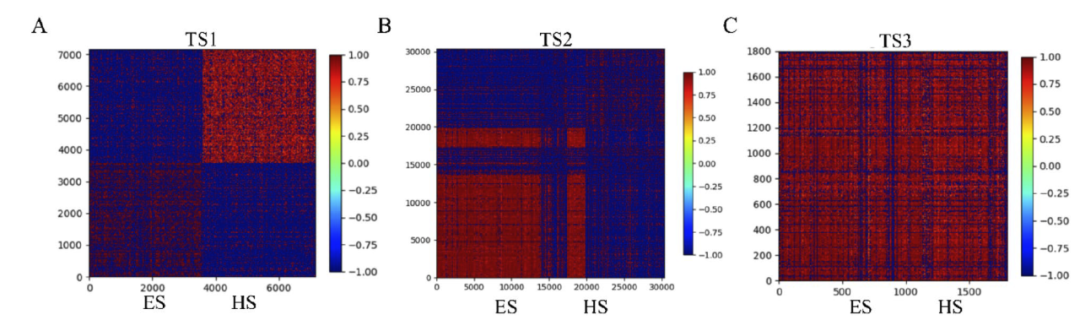
图 3
在此研究中,使用了三个独立测试集TS1、TS2和TS3来比较DeepSA与一些最先进的分子合成可及性评估方法。结果总结在表2中,显示DeepSA能够在TS1上完美区分ES和HS,在TS2上的表现仅略低于SAscore,在最具挑战性的TS3上超越所有现有方法,表明其在识别类似化合物的合成难度方面的出色性能。基于SmELECTRA模型的DeepSA在TS3上表现更佳,反映了真实世界分子合成的难度(图2A),因此作者将DeepSA_SmELECTRA作为后续分析的标准DeepSA模型。如图2B所示,展示了评估化合物合成可及性的现有方法的ROC曲线,包括GASA、SYBA、SCscore、SAscore和RAscore。DeepSA在识别HS方面比GASA和其他模型实现了显著更高的早期富集率。如表2和图2所示,模型的预测准确率在TS1、TS2和TS3上依次降低,这可能是由于这三个独立测试集中的各种分子属性不同造成的。因此,作者使用指纹和尤尔相似性度量来计算这三个数据集的相似性矩阵,并以热图形式展示在图3中。TS1中的ES和HS分子群体在各自群体内显示出高指纹相似性。ES和HS组之间可以看到相当显著的差异。这表明分子特征的模式差异可能对区分HS和ES很有帮助(图3A)。图3B展示了TS2中分子的指纹模式。大多数分子在HS和ES之间显示出明显的差异,但有些分子在HS和ES之间显示出相似的模式,这在预测中呈现一定难度。与TS1和TS2不同,TS3中HS和ES的指纹模式非常相似,表明了该测试集的挑战性(图3C)。
DeepSA的泛化能力
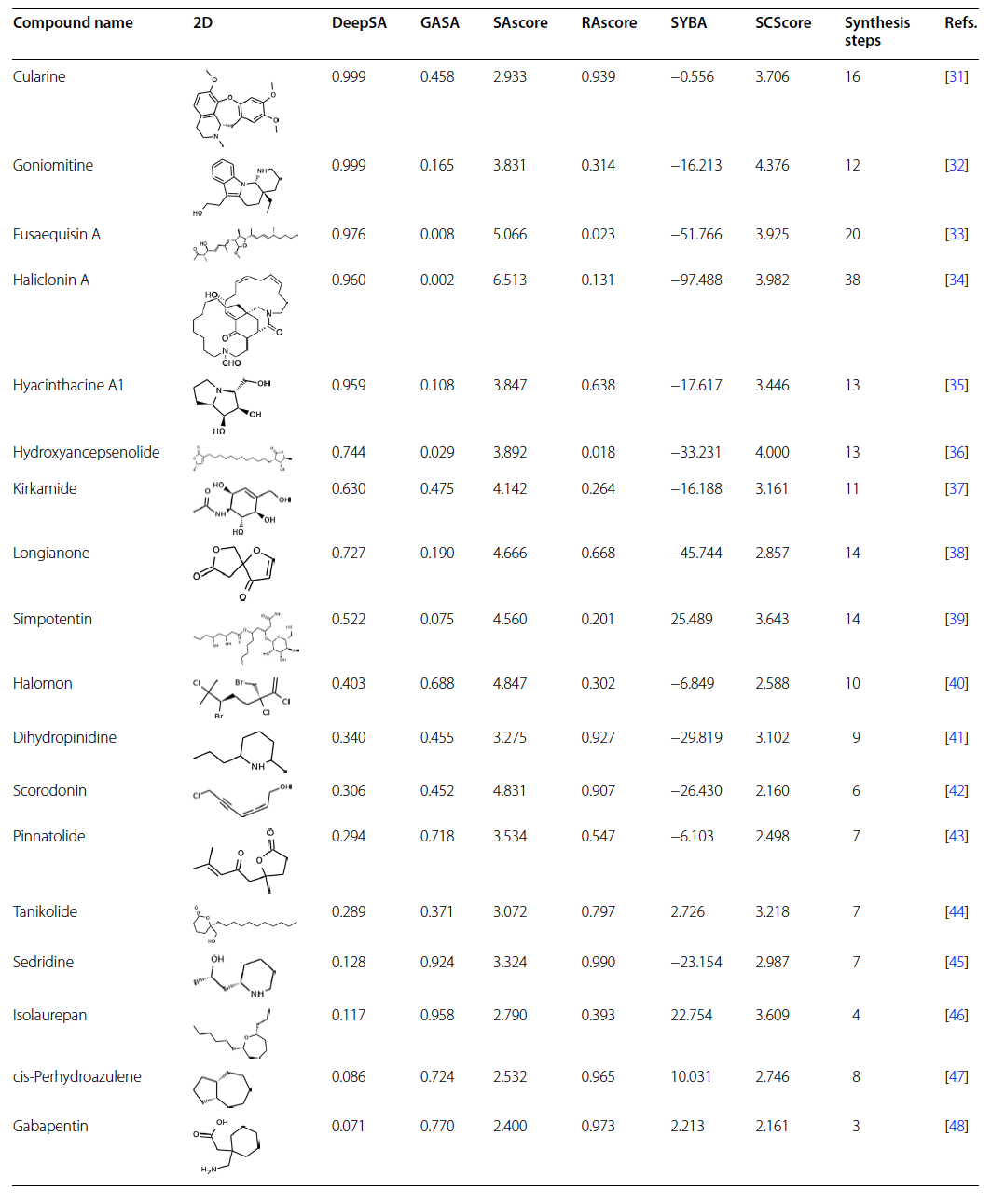
表 3
为了进一步验证DeepSA对具有真实合成路径化合物的预测性能,作者测试了18种已发布合成路径的化合物(这些是真实的合成路径,而不是逆合成分析或化学家创建的路径),这些化合物不属于训练集和独立测试集(表3)。使用DeepSA和其他合成可及性评估方法为这些化合物预测了合成可及性得分。结果显示,当按10个合成步骤划分时,DeepSA成功区分了所有化合物的合成难度标签。同时,作者检查了这些化合物在DeepSA中的特征,其中每个化合物表示为一个256维的向量。由于DeepSA是一个化学语言模型,作者探究了同一化合物的不同随机SMILES是否对嵌入有影响。作者为每种化合物生成了三种不同的随机SMILES表示,并提取了上述18种化合物的共54个嵌入特征。最终对所有嵌入进行了归一化。作者发现,在大多数维度向量区域内,HS和ES的嵌入特征差异显著。ES的前一百维的向量大小明显高于HS,而在最后一百维则相反。HS或ES群体内的模式差异主要在维度向量的中间范围。同时,嵌入的差异与化合物合成可及性的预测得分相对应。如果一个化合物被预测为HS或ES的概率较高,它在嵌入特征中会更加明显地标记。对于由不同随机SMILES表示的同一化合物,嵌入大多显示出高度相似性,这可能表明同一化合物的不同随机SMILES不会影响合成可及性的预测准确性,显示出DeepSA模型具有一定的稳健性。
编译 | 曾全晨
审稿 | 王建民
参考资料
Wang, S., Wang, L., Li, F., & Bai, F. (2023). DeepSA: a deep-learning driven predictor of compound synthesis accessibility. Journal of Cheminformatics, 15(1), 103.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


