今天为大家介绍的是来自Antoine Taly团队的一篇论文。论文介绍了AlphaFold2(AF2)和RoseTTaFold(RF)两种作为高度可靠和有效的蛋白质结构预测方法对结构生物学的革命性影响。文章探讨了这两种技术的影响和局限性,重点关注它们在实验流程中的整合,以及在多种蛋白质类别中的应用。

AlphaFold和RoseTTaFold迅速改变了结构生物学领域,并被广泛认为是可靠且有效的蛋白质结构预测方法。这些方法的成功已经通过在CASP(蛋白质结构预测的关键评估)第14和15轮的严格评估得到证实。然而,当预测性地研究新的蛋白质结构时,研究人员必须能够批判性地评估这些模型的质量。这篇观点论文源自于2022年11月举行的“结构生物学中的机器学习和人工智能:超越AlphaFold2/RosettaFold探索剩余盲点”的研讨会。参与者探讨并讨论了这些工具从建模和实验角度的使用,提出了知识共享的问题。作者提出了一些待验证的想法,即鼓励读者使用以下方法,特别是在未知结构的案例上,以便在日常实践中更广泛地测试这些模型。
将Alphafold2融入实验中
虽然AlphaFold2(AF2)在蛋白质结构预测方面取得了显著成果,但它并没有完全解决结构预测问题。AF2不应该与传统的实验方法对立,而是应该与实验数据结合使用。例如,最近的一些研究聚焦于评估AF2模型在实验环境中的应用。这些模型的优势在于,当之前对某个蛋白质结构一无所知时,现在能够提供接近现实蛋白质结构。在Akdel等人的研究中,用AF2得到的模型与之前通过同源建模得到的模型进行了比较。结果显示,AF2能够为大约25%的蛋白质残基提供新的、高置信度(pLDDT > 70)的预测。此外这些模型还可以用来探索突变及其在蛋白质三维结构中的效应。
质谱(MS)是另一种与结构预测方法互补的实验方法。事实上,已有多项研究展示了如何将MS数据与AF2模型结合。目前已经开发了一些特定工具将MS数据纳入建模过程,即AlphaLink和其对AlphaFold-Multimer的扩展,AlphaLink2。核磁共振(NMR)与AF2模型具有显著的互补性。事实上,实验结果已经显示我们可以自信地使用AF2模型来指导和补充实验NMR数据分析。先前研究人员开发的一种预测蛋白质柔性的新工具使用AF2预测结构中的局部接触和pLDDT来计算N-H键的S2顺序参数,这与NMR实验数据相符。在CASP14之后,人们预期AF2预测可能会对分子置换产生影响。相关实验展示了AF2和RF模型在分子置换方面的潜力,包括对于之前的工具无法应用的目标。这一结果与AF2和RF超越先前模型的观点一致,它们可以使用多序列比对(MSA)的信息预测没有PDB对应物的蛋白质结构。
AlphaFold2(AF2)在预测寡聚体的应用
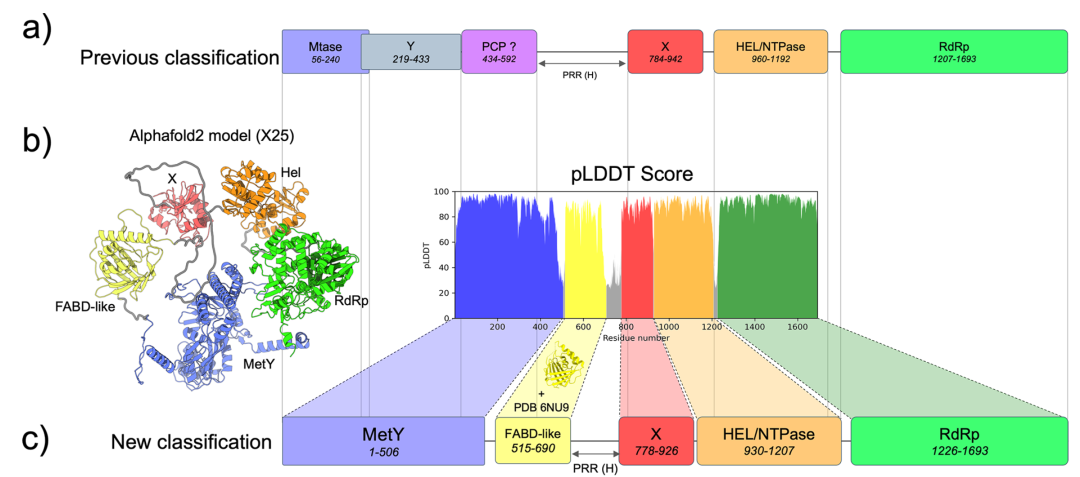
图 1
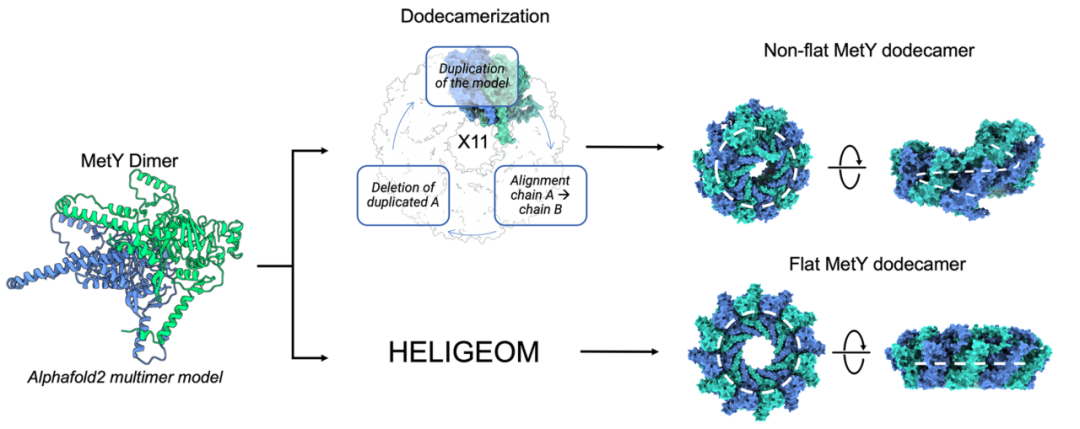
图 2
生物系统通常包含众多蛋白质复合体,AF2能有效建立这些复合体的结构,有助于分析蛋白质之间的相互作用。这在病毒学等蛋白质相互作用至关重要的领域尤为突出。例如,最近有研究使用AF2预测了甲型肝炎病毒(HEV)中一种由开放阅读框架(pORF1)编码的蛋白质的结构和域界限。相关问题已经争论数年,但AF2的pLDDT评分提供了新见解。pLDDT评分与蛋白质的无序程度相关,有助于确定多域蛋白中的域界限。在甲型肝炎病毒的研究中(图1),pLDDT评分提示合并前两个域,并重新定义第三个域的位置,支持pORF1中没有假定的蛋白酶的假说。AF2识别结构中的第一个域与最近公布的脂肪酸结合域(FABD)的结构非常相似。尽管识别方法和考虑的序列数量不同可能导致构象的显著差异,但结构相似性比较方法表明它们有相同的折叠方式。最终结构中甲基转移酶和膜结合域(Met和Y)被合并为一个名为MetY的域。使用结构比较方法发现,MetY的结构与基孔肯亚病毒(CHIKV)的nsP1蛋白非常相似,后者同样具有甲基转移酶和膜结合功能。
然而,预测多链寡聚体结构的准确性会随着链数的增加而降低,AF2难以预测一个500残基域的十二聚体状态。研究显示,AF2预测的C2二聚体构象与通过对两个分子进行多次变换得到的十二聚体状态一致。尽管最终的十二聚体状态不是完美的,但使用HELIGEOM软件优化两个单体之间的接触,得到了一个平整、无碰撞的十二聚体结构(图2)。这表明AF2能预测导致更高寡聚体状态的二聚体接触,虽然其架构的限制使得预测大型结构具有挑战性。其他方法,如Unifoldsymmetry,可以克服这一障碍,但它们需要指定对称状态。尽管Unifoldsymmetry与AF2一样基于多序列,但其对MetY域的单体构象比不上更真实的AF2模型。
在没有高分辨率结构数据的情况下,验证模型是否符合已知的生物学观察成为必要。在HEV pORF1的建模过程中,研究人员使用不同的序列比对方法(如HHBlits、MMSeqs)和多样化的参数,产生了30多个不同的结构,这些结构展示了多种可能的构象。结构根据所采用的方法呈现不同的特征,其中5个域中的3个有不同的构象和折叠状态被观察到。研究者根据现有文献和域功能知识选择了特定的构象,有时使用实验结构模板,有时从AF2模型中提取显示准确构象的域。colabfold实现的AF2有助于使用这些模板进行建模,显著提升了预测结构的置信度和质量。这种方法充分利用了AF2在预测域间接触方面的能力改善了局部折叠质量。
无序蛋白
固有无序蛋白质(IDPs)和含有固有无序区域(IDRs)的蛋白质在许多生物过程中扮演着重要角色,如信号传导和转录,并且在真核生物的蛋白质中非常丰富。虽然AlphaFold2(AF2)最初是为预测折叠蛋白质的结构而开发的,但很快就明确了,pLDDT值(作为每个残基结构预测的置信度评分)也可以作为一种与其他标准方法相比具有竞争力的无序预测器。例如,估计大约30%的人类蛋白质组由IDRs组成;在AF2预测的人类蛋白质组结构中,相同比例的残基显示出非常低的pLDDT值(<50),这两组数据有很强的重叠。然而,人们发现AF2在蛋白质序列中过度估计无序。例如在一项研究的评估中大约一半的残基显示出低置信度(<70)的评分。
最近的研究指出了蛋白质序列中AF2预测的固有限制的可能性,也就是低置信度的结构预测不是与无序相关,而是对应于由于AF2的固有限制(如目标序列缺乏共进化信息)而没有正确预测的可折叠域。在这种情况下,可以将AF2的预测与基于残基物理化学性质的额外工具结合使用,如在疏水团簇分析(HCA)中的疏水性,以揭示对AF2隐藏的有序片段。值得注意的是,蛋白质中的无序具有不同的形式,条件性有序可以由不同的实验条件产生。有时高置信度的pLDDTs与属于单体单元中无序蛋白片段的残基相对应,这些片段在特定条件下会折叠,例如在与另一个蛋白结合时。因此,应谨慎对待AF2模型,多项研究显示它们可能预测的是蛋白质结合结构而非溶液中的非结合结构。
膜蛋白
膜蛋白在多种生物过程中发挥作用,如信号传导、分子运输、细胞增殖等等。它们是重要的蛋白质类别之一,因为它们与癌症和疾病有关,是当前超过60%药物靶标的组成部分。然而,由于它们的高疏水性和对膜环境的依赖性,与可溶性蛋白质相比,只有少量的膜蛋白的结构被解析。尽管蛋白质结构技术的最新进展增加了可用的膜蛋白3D结构的数量,但已知的膜蛋白序列和它们的实验确定结构之间仍然存在显著差距。基于深度学习的蛋白质折叠预测模型,如AF2和RF,有潜力通过提供膜蛋白的宝贵结构信息来弥合这一差距。然而,这些模型存在必须解决的问题。例如,机器学习(ML)折叠模型在倾向于预测能量较低的结构,而蛋白质结构实际上是一系列的构象集合。此外,这些构象集合可以通过它们的环境(例如,配体或脂质结合)和突变来变构调节。在一项最近的研究中,Kiriakidi等人使用AT1受体的动力学模拟显示,结合胆固醇后可以观察到构象变化。在PI3Kalpha的例子中,尽管E545K和H1047R点突变远离活性位点,但它们改变了PI3Kalpha的构象,消除了其C端的自抑制作用,改变蛋白-膜相互作用,并扰乱变构调节途径。ML折叠模型很难预测这些构象变化,因为它们是基于静态结构和进化信息训练的,尽管最近已经进行了尝试。
这种预测构象集合的限制也影响到预测蛋白质-蛋白质多聚体结构,以及当蛋白质接触细胞膜时的结构。例如,对视紫红质二聚体的动力学模拟揭示了多个二聚体界面的采用,这些界面未被AF2预测。在未来,ML折叠模型在预测膜蛋白折叠方面涉及几个关键方面。首先,需要改进构象集合的建模,以捕捉膜蛋白的动态本质和变构调节。其次,对蛋白质结构受突变影响的研究尚不足,限制了我们预测由突变引起的结构变化的能力。最近的研究表明AF2确实可以预测单一突变的效果。此外,将膜特异性特征(如疏水性、跨膜域、蛋白质-膜相互作用、膜组成和膜拓扑)纳入ML折叠模型可以改进膜蛋白的预测,尽管这些数据可能不足以进行训练。最后,预测由高度灵活的单体组成的多聚体结构处于初期阶段,需要显著进展。此外,在研究膜蛋白时,将核酸结构或其他结合分子的描述纳入考虑是至关重要的,因为这些相互作用通常具有重要的生物学意义。最后,揭示膜蛋白的未知折叠过程将有助于深入了解它们的折叠机制,并可能揭示新的功能见解。
模型验证
模型验证可以通过将模型与实验数据(如突变分析、质谱、核磁共振)或来自独立建模工具的数据进行比较来完成。虽然AF2和RF模型与实验数据的互补性已经是许多研究的主题,但可能存在对独立建模工具的使用不足。在AF2的开发过程中,开发者选择不依赖于蛋白质的物理化学知识,尽管它可能已经隐式地学习了一些这方面的知识。虽然不显式使用蛋白质的物理化学知识可能是AF2成功的一个因素,但这不应阻止用户使用这些知识来验证模型。事实上,模型建立过程中没有这些标准使得它成为了一个独立的验证工具。除了像上文提到的,与蛋白质的生化专家分析结构的可能性外,分析结构的基本属性也是值得的。这也为通过分子动力学模拟或直接基于角度的方法改进模型提供了可能。
编译 | 曾全晨
审稿 | 王建民
参考资料
Versini, R., Sritharan, S., Aykac Fas, B., Tubiana, T., Aimeur, S. Z., Henri, J., … & Taly, A. (2023). A Perspective on the Prospective Use of AI in Protein Structure Prediction. Journal of Chemical Information and Modeling.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


