今天为大家介绍的是来自Günter Klambauer团队的一篇论文。生物图像分析领域正经历一场由先进成像技术和人工智能推动的变革。多模态人工智能系统的出现使得我们能够从生物成像数据库中提取并利用基于其他数据模态的知识。此研究利用了多模态对比学习方法,通过生物图像和分子结构编码器,将生物图像和化学结构融入到一个统一的空间中。这种共同的嵌入空间使得我们可以利用化学结构来查询生物成像数据库,找出引起不同表型效应的结构。

生物和化学数据库及其查询机制是分子生物学研究的核心。例如,序列数据库(如RefSeq或UniProt)包含DNA或蛋白质序列,通常通过BLAST或其变体用特定序列进行查询。基因组数据库允许多种查询方式,如基因位置、基因名称等。蛋白质结构数据库(如蛋白质数据PDB)提供从序列相似性到基于3D形状的结构查询。化学数据库ChEMBL和PubChem是巨大的化学结构库,包含数十亿小分子。国际化学标识符(InChI)旨在便于在这些数据库中搜索化学结构。虽然BLAST、PDB中的结构搜索和基于InChI的查询可以被视为关联或基于内容的查询,但生物成像数据库仍依赖于手工注释和基于文本的搜索。然而,用能图像捕捉到的表型效应的化学结构查询大型生物成像数据库,可能大大增强生物医学研究。同样重要的是,利用显微镜图像(捕捉化学结构的表型效应)查询化学数据库,如图1ab所示。
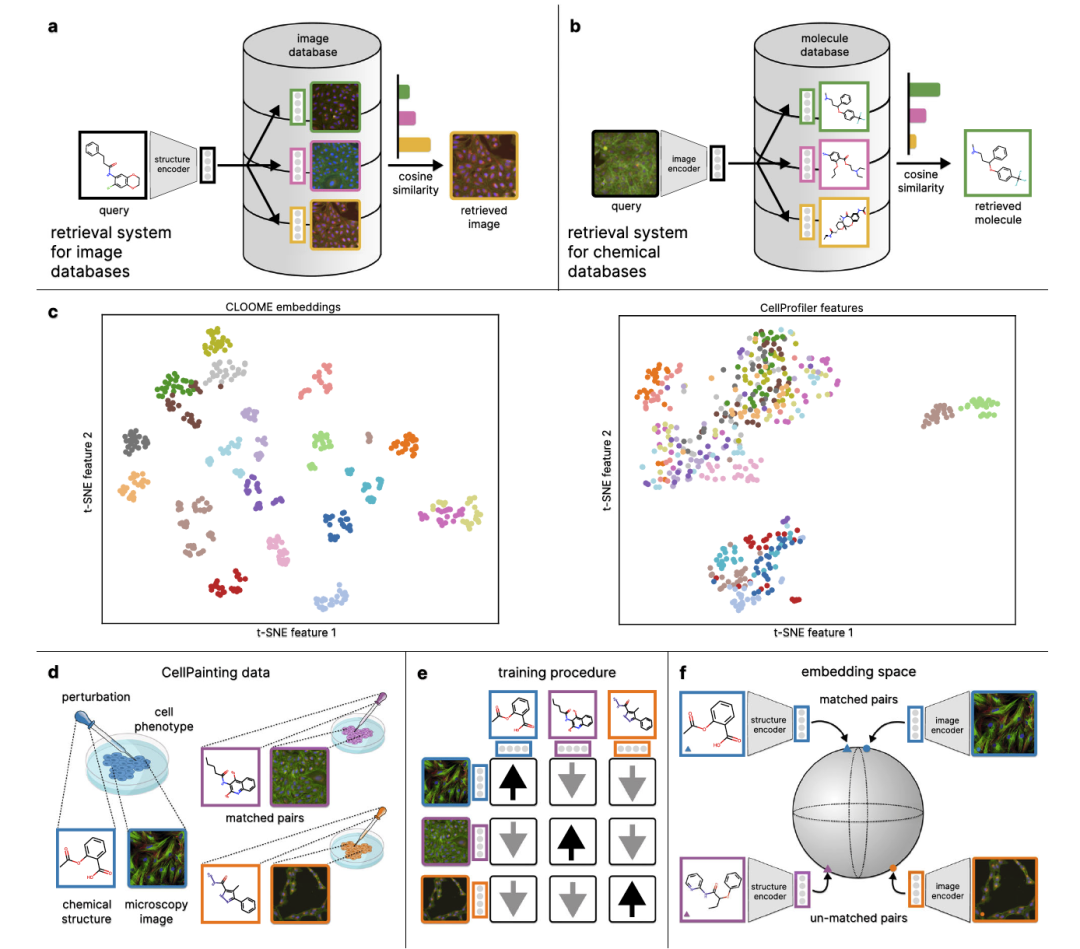
图 1
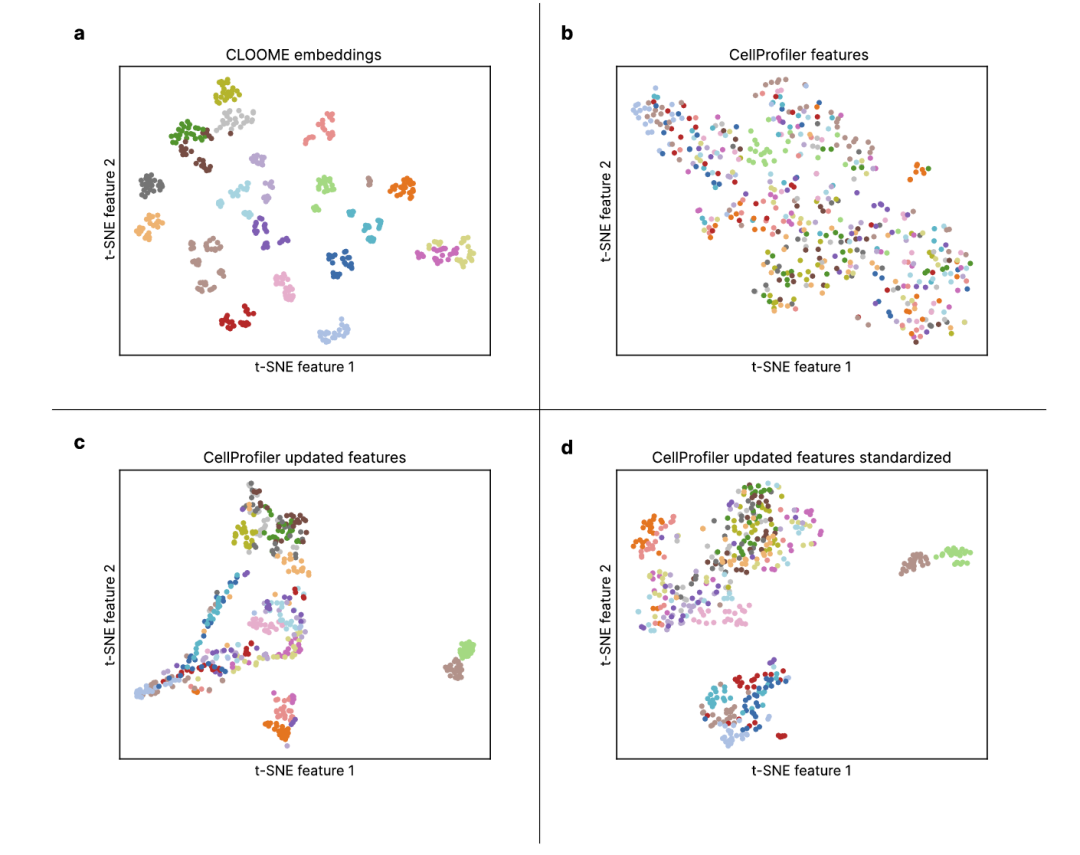
图 2
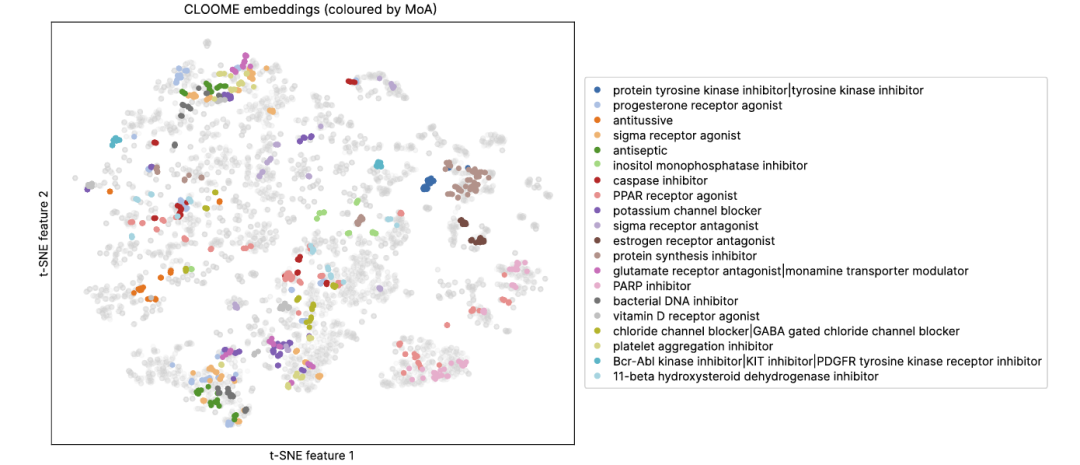
图 3
最近,对比学习作为一种强大的范式出现,用于从数据中学习丰富的表征。对比学习方法CLIP和CLOOB将自然语言和图像嵌入到相同的表示空间。对比学习确保图像及其匹配的文本标题在这个嵌入空间中彼此接近,而不匹配的图像和标题则被分开。因此,文本提示可以通过提取嵌入空间中附近的图像(反之亦然)来查询图像数据库。在这项工作中,作者使用这些强大的对比学习方法(CLOOME),使生物成像数据库的查询或检索系统成为可能。显微镜成像作为一种信息丰富且节约时间和成本的生物技术,用于表征细胞表型、组织或细胞过程。因此,科学界在使用高通量显微成像作为细胞系统和细胞表型在多种干扰下的信息读出和表征方面做出了重大努力。例如,从显微镜图像计算的特征已被证明在高通量筛选实验中找到比从化学结构更高多样性性能的化合物集合。除了对人类专家可理解和有益的信息外,这些显微镜图像还包含大量对人类来说难以理解的生物信息,但可以通过计算方法(如深度学习)成功提取。大量的显微镜图像存储在大型生物成像数据库中,然而,它们的查询机制仍然仅限于文本注释的查询。一个共同的嵌入空间,包括(a)捕捉干扰表型效应的显微镜图像,和(b)引起这些效应的化学结构,将允许对成像和化学数据库进行关联或基于内容的查询。这样的嵌入空间将以诱导它们的化学结构和捕捉这些过程所导致的细胞表型的图像的形式,代表细胞过程。通过这样的嵌入空间,可以通过检测新的细胞表型等新应用成为可能(图2,图3)。
表征实验评估部分
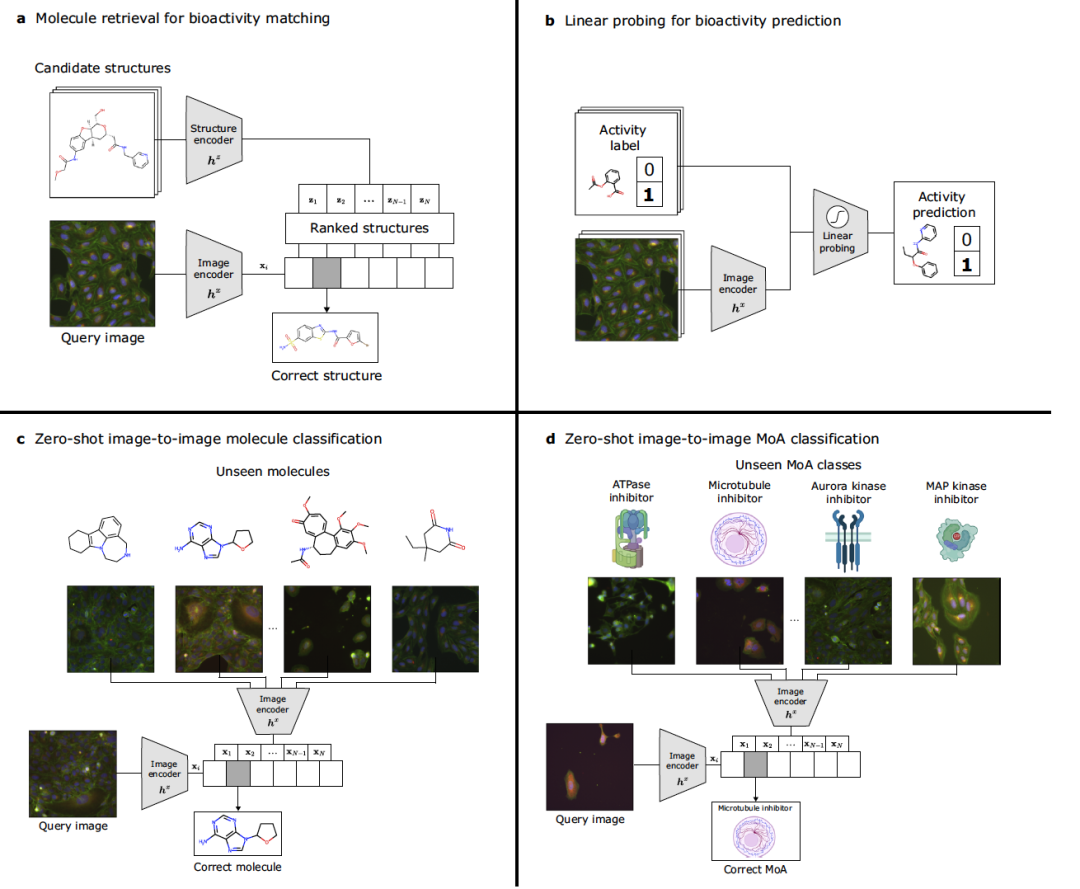
图 4
文章的论述顺序如下:作者首先展示了CLOOME作为生物成像和化学数据库检索系统的能力(见图4a)。接着利用CLOOME的图像嵌入来预测实验生物活性,仅通过拟合逻辑回归模型(见图4b)。最后评估了CLOOME图像嵌入在分子和作用机制分类任务中的表现。图4c和4d展示了这些任务的实验设置。
作者首先评估了CLOOME准确检索与显微镜细胞图像匹配的化学结构的能力。值得注意的是,这对于人类专家来说是一个极具挑战性的任务,甚至被认为几乎不可能实现:给定细胞的显微镜图像,任务是从数千个候选结构中选择与之匹配的化学结构。由于细胞常常不表现出任何或只有微妙的表型变化,这个任务很难。这种基于图像的检索任务也可以被理解为生物等效替代任务:生物等效物是具有大致相同生物属性或活性的分子。在药物发现中,当需要用另一种分子替换某种分子,同时保持其生物活性时,生物等效性非常重要。极端情况的生物等效替代是骨架跳跃,因为需要找到化学结构高度不同但仍具有相同生物活性的分子。通过这个实验作者评估了CLOOME准确排列与相应图像匹配的分子结构的能力。排名靠前的其他结构可能是潜在的生物等效物,这使得该实验成为生物等效替代问题的替代。作为基线,作者训练了一个线性模型,以及一个随机基线。
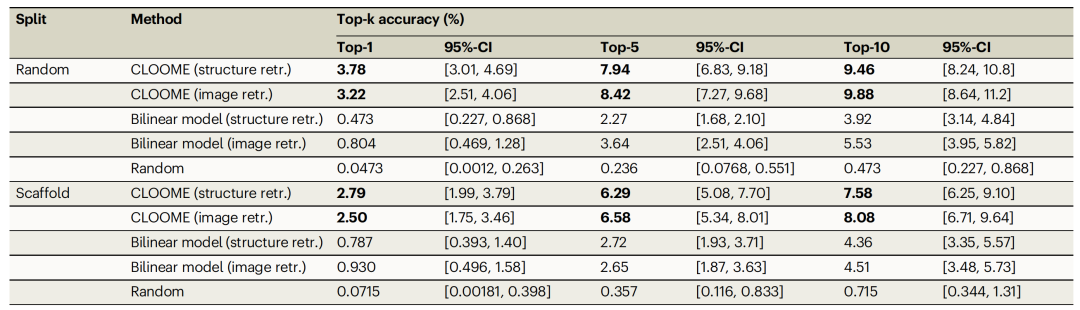
表 1

图 5
在2115个图像和分子对的保留数据上,CLOOME在3%的情况下将匹配的分子排在第一位。随机方法的值约为1/2115 ≈ 0.047%,这表明CLOOME的改进约为70倍。对于从2115个实例的数据库(随机拆分)和1398个实例的数据库(骨架拆分)检索,表1给出了top-1、top-5、top-10的准确率。另外,作者还报告了1%采样率(即1个匹配例子与99个不匹配例子)下的相同指标,这是评估检索系统常用的设置。此外,一些示例展示在图5中。据作者表示,这是第一个基于细胞图像的分子结构检索系统。
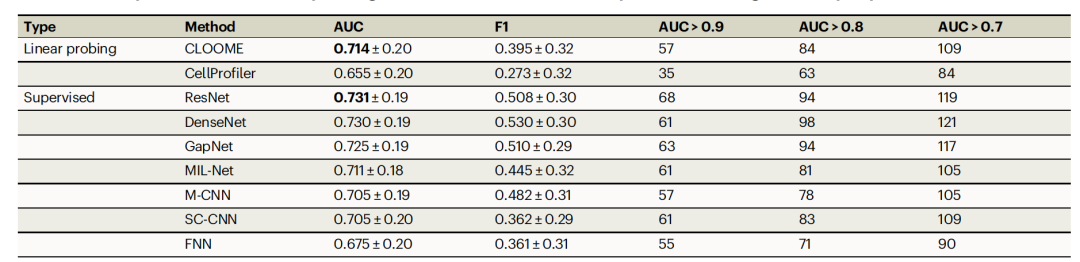
表 2
随后作者测试了CLOOME学习到的表征是否可以通过线性模型在209个下游活性预测任务上进行迁移。线性探测测试常用于对比学习方法,以检验学习到的特征的迁移能力。在这类实验中,使用预训练编码器的表征,并且只拟合一个单层网络(如逻辑回归)到监督任务的给定标签上。如果线性探测测试具有良好的预测质量(通常低于完全监督的方法),则这些表征被认为是可迁移的。结果报告在表2中。CLOOME在预测任务中达到了平均AUC为0.714 ± 0.20,这表明所学习的表征确实是可迁移的。在AUC方面,CLOOME甚至超过了完全监督的方法,如M-CNN和SC-CNN。
零样本评估部分
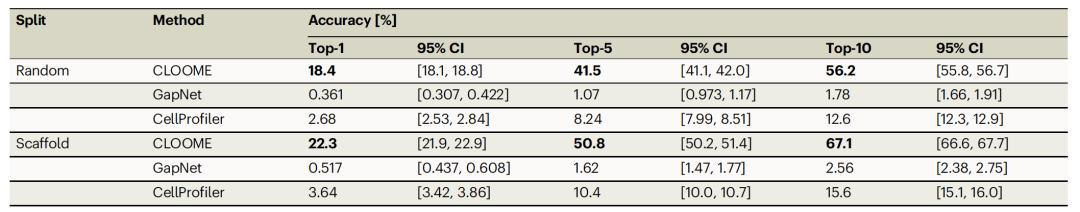
表 3
作者评估了CLOOME的图像嵌入在区分特定分子方面的潜力。这种评估也可以看作是对用同一分子处理的细胞图像嵌入聚集程度的定量评价。需要注意的是,与检索任务中使用分子和图像嵌入不同,此处仅使用了图像嵌入。任务采用了零样本分类设置,意味着测试集包含了新的、即“未见过”的图像类别,这些类别在训练集中未被包括。具体而言,从测试集中的每种分子随机选择了一张图片构成“保留测试集”,这意味着每个未见过的分子类别由单张图片代表。然后,为确保分类不受板效应影响,从完整测试集中移除了该集合中的样本以及对应于相同分子和板的样本。随后将剩余的样本称为“测试集”。测试集在随机拆分中包含了43,778个样本,在骨架拆分中包含了28,248个样本。如表3所示,CLOOME在top-1、top-5和top-10准确率指标上的表现优于GapNet和CellProfiler提取的特征。
注:“保留测试集”指的是2115个样本和1398个样本(一个样本为一对数据:分子+细胞图像)构成的集合,任务目标是从“测试集”图片查询唯一对应的保留测试集中的图片。因为测试集和训练集划分时不包含相同的分子,所以这项任务为zero-shot。
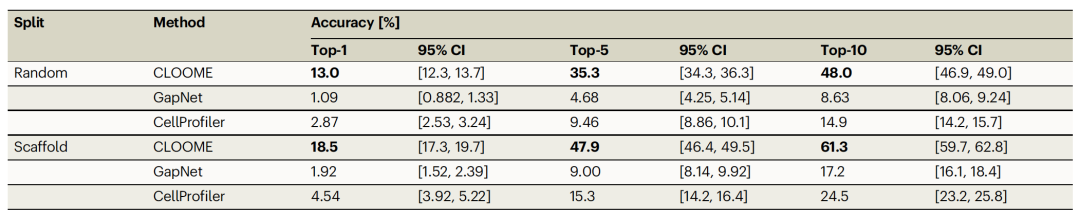
表 4
随后作者应用CLOOME进行了一项挑战性的零样本作用机制(MoA)图像分类任务。这项任务旨在评估这些嵌入特征根据其作用机制的区分度有多高。为了进行这种评估,作者从Drug Repurposing Hub获取了存在于验证集和测试集中的分子作用机制标签。此任务遵循了与零样本分子分类任务相同的程序。在随机分割中,“保留测试集”由126种MoA和202种分子组成。对于骨架拆分,“保留测试集”包含了68种MoA和93种分子。至于测试集,随机拆分中有8826个样本,骨架拆分中有4056个样本。表4中显示的结果表明,CLOOME在作用机制预测方面的性能优于GapNet和CellProfiler特征。
编译 | 曾全晨
审稿 | 王建民
参考资料
Sanchez-Fernandez, A., Rumetshofer, E., Hochreiter, S. et al. CLOOME: contrastive learning unlocks bioimaging databases for queries with chemical structures. Nat Commun 14, 7339 (2023).
https://doi.org/10.1038/s41467-023-42328-w



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


