
编译 | 倪昱静
审核 | 王永康
今天分享的是德国结构生物学研究所发布在bioRxiv上的一篇论文:MISATO – Machine learning dataset of protein-ligand complexes for structure-based drug discovery。该论文提出了第一个结合小分子的量子力学性质和相关的分子动力学模拟的蛋白质配体复合物数据集。
Part1摘要
大型语言模型(LLMs)大大提高了我们理解生物学和化学的能力。然而,基于结构的药物发现的可靠方法相对较少,迫切需要高精度的生物分子-配体相互作用数据集,特别是对于需要大量训练数据的LLMs。本文提出MISATO,这是第一个结合了小分子的量子力学性质QM和相关的分子动力学模拟MD的约20000个实验蛋白质配体复合物的数据集。作者从PDBbind数据集开始,使用半经验量子力学来系统地完善这些结构,并且包含了迄今为止最大的蛋白质-配体复合物的分子动力学痕迹收集,累积到170 μs。本文还提供了ML基线模型应用和简单的Python数据加载器,并旨在围绕MISATO培养一个蓬勃发展的社区。
Part2背景
近年来,人工智能(AI)预测已经彻底改变了许多科学领域。在结构生物学中,AlphaFold 2仅从氨基酸序列就能准确预测蛋白质结构,其精度接近最新的实验数据。它的成功得益于蛋白质数据库(PDB)中存储的近20万种蛋白质结构的丰富数据库。在过去的几十年里,这些结构是通过x射线晶体学、核磁共振(NMR)或冷冻电子显微镜(Cryo-EM)等实验确定的。而在药物发现领域,尽管投资巨大,每年获批的新药仍然很少,开发成本过高,合理的基于结构的药物发现是一个持续的重大挑战。与蛋白质结构预测相比,该任务要困难得多。
虽然计算方法原则上能够解决基于结构的药物发现,包括半经验量子力学(QM)方法、分子动力学(MD)模拟、对接和粗粒度模拟,也可以将这些方法结合起来而更有效,但这些方法要么精度低,要么计算成本太高,同时仍然需要大量的实验验证。将人工智能引入这一过程仍处于早期阶段。原则上,AI方法能够学习描述实验数据的基本状态变量。然而,到目前为止,已经提出的大多数简单的解决方案都没有充分纳入可用的蛋白质-配体数据,例如计算蛋白质-配体吉布斯自由能、ADME性质估计或合成路线预测。大多数这些方法是使用一维SMILES构建的,只有少数尝试正确处理3D生物分子配体数据。目前基于结构的人工智能模型受到几个因素的严重阻碍:
- 忽视了构象的灵活性(动力学和结合时诱导的配合);
- 熵的注意事项;
- 结构数据的不准确性(由于缺少氢原子而导致的错误原子类型、错误的官能团、不一致的几何约束、拟合错误);
- 化学复杂性(例如,不明显的质子态);
- 过于简化的原子性质;
- 目标分子识别中的高度复杂的能量景观。
Part3结果
构建过程
MISATO的基础是来自PDBbind的19443个蛋白配体结构。(这些结构是通过实验确定的,代表了一组不同的蛋白质配体复合物,这些复合物具有实验亲和力。)
这是第一个将小分子的半经验QM特性与整个实验蛋白质-配体复合物的MD模拟动力学相结合的数据集。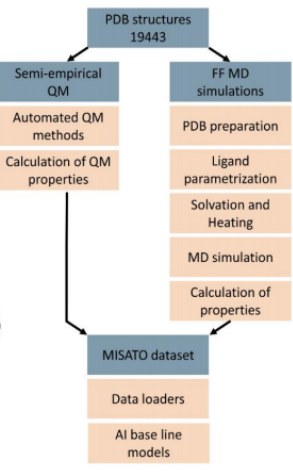
- 量子力学性质QM
在PDB中,不正确的原子分配和不一致的几何图形并不少见。更严重的是,氢原子对其化学和分子环境高度敏感,并且很少通过实验获得。如图所示,展示了一些结构不一致的例子:1WUG包含过度拉长的NO键;4MDN中含有违反VSEPR的氮;5GTR显示了质子化状态的典型问题。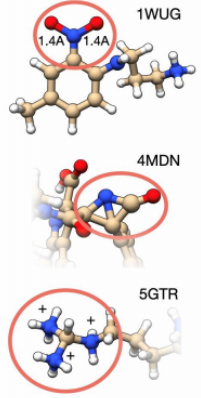 一致的原子赋值是使用一系列半经验测试确定的。半经验量子化学方法在精度和计算效率之间提供了一个很好的折中,适合于精炼各种化学性质和尺寸的近20000个结构的集合。该部分的细化协议从搜索具有强原子重叠的结构开始。接下来,寻找存在波函数收敛问题的结构。最后,研究QM几何优化后配体连通性模式的变化。最终,一共修改了3930个结构,大约相当于原始数据库的20%。如图所示,最常见的调整是从初始的PDBbind几何结构中去除(氢原子)原子,这相当于几乎75%的修改。随后计算了配体的分子和原子性质。
一致的原子赋值是使用一系列半经验测试确定的。半经验量子化学方法在精度和计算效率之间提供了一个很好的折中,适合于精炼各种化学性质和尺寸的近20000个结构的集合。该部分的细化协议从搜索具有强原子重叠的结构开始。接下来,寻找存在波函数收敛问题的结构。最后,研究QM几何优化后配体连通性模式的变化。最终,一共修改了3930个结构,大约相当于原始数据库的20%。如图所示,最常见的调整是从初始的PDBbind几何结构中去除(氢原子)原子,这相当于几乎75%的修改。随后计算了配体的分子和原子性质。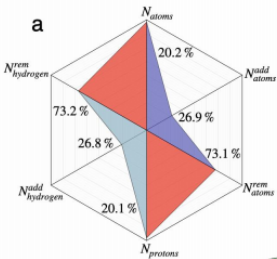
- 分子动力学模拟
实验结构数据是静态快照,代表了晶体中捕获的热力学最稳定状态,但忽略了构象动力学的存在。可以进行分子动力学模拟,从实验结构开始,并使用描述分子势能表面的力场让它们随时间变化。对16972个蛋白质-配体复合物进行了10ns的MD模拟。当遇到不规范的配体原子或蛋白质起始结构不一致时,该结构被忽略。如图,在模拟过程中,可以捕捉到绑定袋的可逆打开和关闭,包括隐藏的绑定位点。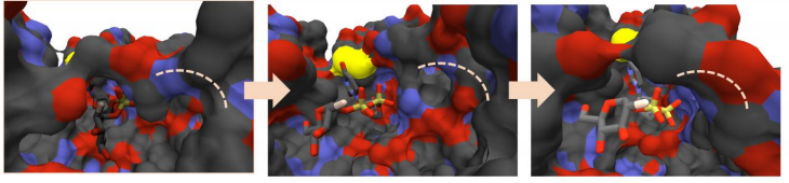
数据集
数据库可以从Zenodo下载。数据以分层数据格式(HDF)存储。作者创建了两个H5文件,一个用于量子化学数据,一个用于蛋白质-配体动力学。使用PDB-id为每个结构划分数据。QM数据可以通过PDB-id访问,性质分为原子性质和分子性质。MD数据也可以通过PDB-id访问,如名称所示,这些属性有的是针对所有原子计算的,有的是针对MD模拟过程的时间步长(帧)或者针对整个轨迹计算的。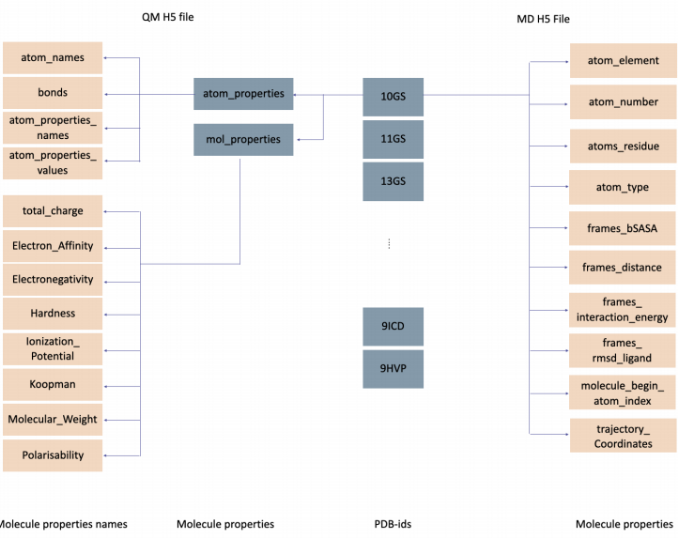 作者还提供了访问数据集文件和相应的数据加载器的示例代码。
作者还提供了访问数据集文件和相应的数据加载器的示例代码。
Part4应用示例
为了举例说明该数据集的可能应用,作者根据atom3d代码库使用数据集训练和评估了该基线AI模型,
这是一套在分子应用背景下的全面的机器学习方法。
-
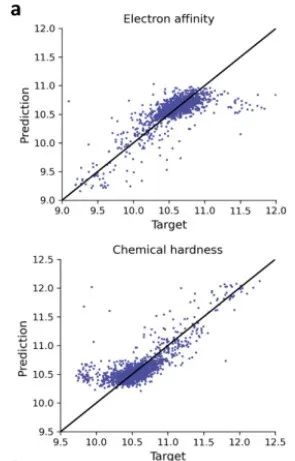
对于QM数据:遵循atom3d42中用于小分子特性预测的GNN架构,预测了配体分子的电子亲和度和化学硬度。使用相关性和平均绝对误差(MAE)评估模型的性能。如图,展示了电子亲和度和化学硬度预测值的散点图。用于预测QM特性的基线模型在电子亲和度和化学硬度方面分别具有0.75和0.77的高度相关性。对于这两个典型的QM特性,实现了较高的精度,为快速推导QM特性开辟了一条道路。
-
对于MD数据:采用的基线模型的架构是5个顺序的GCNConv层和两个线性层,预测了诱导的蛋白质适应性。该模型能够识别生物分子结构中可能适应配体结合的元素。使用皮尔逊相关性和每个复合物中100个最灵活的原子的平均精度来评估训练性能。为了计算每个原子x的适应性,取每个原子在所有时间步骤i到原子的初始位置的平均距离:.
如图所示,该模型取得了0.66的平均皮尔逊相关系数。平均而言,前100个原子中有42个被正确预测(图5)。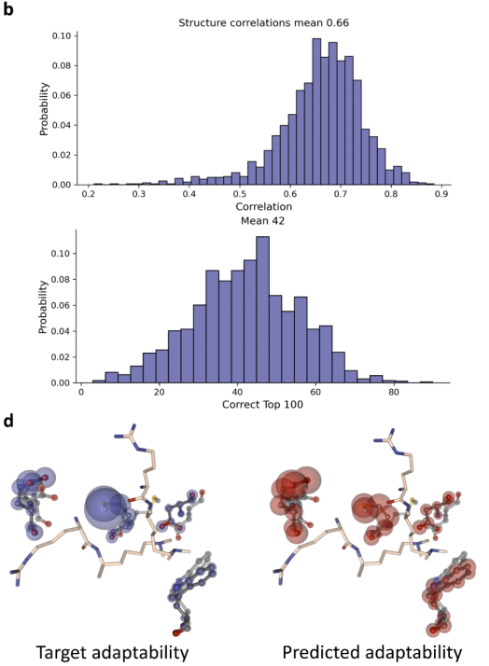 此外,该模型能够预测蛋白质口袋中在MD运行期间最灵活的原子(大球体),也能够检测更具刚性的蛋白质区域(小球体)。这允许对蛋白质口袋进行快速检查,而不需要冗长的MD设置和模拟。
此外,该模型能够预测蛋白质口袋中在MD运行期间最灵活的原子(大球体),也能够检测更具刚性的蛋白质区域(小球体)。这允许对蛋白质口袋进行快速检查,而不需要冗长的MD设置和模拟。
Part5总结
本文提出MISATO,一个将在药物发现中开辟新途径的数据库。MISATO包含第一个量子化学精制配体数据集,含有迄今为止最多的蛋白质配体MD踪迹。总之,MISATO旨在使用人工智能方法为精确的、基于结构的下一代药物发现提供足够的训练能力。
参考资料
文章链接:https://www.biorxiv.org/content/10.1101/2023.05.24.542082v2



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


