今天为大家介绍的是来自Markus J. Buehler团队的一篇论文。自然进化过程中,大自然展现了一系列具有卓越机械性能的蛋白质材料,这些蛋白质在机械生物学中扮演着至关重要的角色。然而,超越自然设计,发现满足特定机械性质要求的蛋白质仍然是一个挑战。在这里,作者报道了一种生成模型,该模型能够预测出为满足复杂的非线性机械性质设计目标的蛋白质设计。作者的模型利用了来自预训练蛋白质语言模型的深层蛋白质序列知识,并将机械展开响应映射出来以创建蛋白质。通过分子模拟进行直接验证,作者展示了所设计的蛋白质是全新的,并且满足了目标机械性质,包括展开能量和机械强度。

近年来,深度学习方法的发展及其在蛋白质研究和设计中的应用为蛋白质研究和设计提供了快速途径。对于以结构识别为中心的前向问题,基于深度学习的工具,如AlphaFold2和RoseTTAFold,在预测基于蛋白质序列的3D折叠结构方面实现了与实验方法竞争的准确性,且成本大大降低。基于这些方法,其他蛋白质折叠工具已经在探索大型语言模型的应用,通过去除对多序列比对(MSAs)作为输入的依赖,展示了在进一步降低计算成本和实现孤立和快速进化蛋白质更好预测方面的改进。基于深度学习的端到端模型已经可以预测给定序列的各种结构特征和属性。同时,满足期望的结构或属性特征的全新蛋白质的逆向设计呈现了一个更具挑战性的任务。面对巨大的序列空间,搭配高效深度学习基础的前向预测器的搜索算法可能仍会受到探索效率低下的困扰,并且发现的序列的设计准确性和多样性不容易控制。另一方面,最近出现的生成模型提供了从期望特性到潜在设计的直接映射,并且正在成为各种材料研究和设计的新兴范式,包括蛋白质。
作者关注蛋白质的纳米机械属性。由于单分子技术的出现,测量在施加机械力下蛋白质展开提供了理解蛋白质变形(弹性/塑性)和断裂的独特分子基础,并且由于内在的结构层次性,可以在影响基于蛋白质材料的一些宏观机械属性中发挥关键作用。因此,生成满足期望机械展开响应的全新蛋白质可以代表蛋白质基材设计中的一个关键分子水平设计步骤。与先前的蛋白质设计案例相比,这个问题提出了一些独特的挑战。首先,这是一个绕过结构级别的属性到序列的端到端设计任务,预计比之前的结构到序列的设计任务更加困难。其次,与蛋白质结构或序列相比,已知蛋白质的机械展开响应的可用或可负担的数据非常稀少。除了机械属性,作者预计这两个挑战也被许多其他蛋白质的属性到序列设计任务所共享。为了解决这个问题,作者结合了一个基于注意力的扩散模型和一个预训练的大型语言模型用于蛋白质,构建了一个生成性深度学习模型,该模型能够根据作为设计目标的机械展开响应来预测氨基酸序列和3D蛋白质结构。
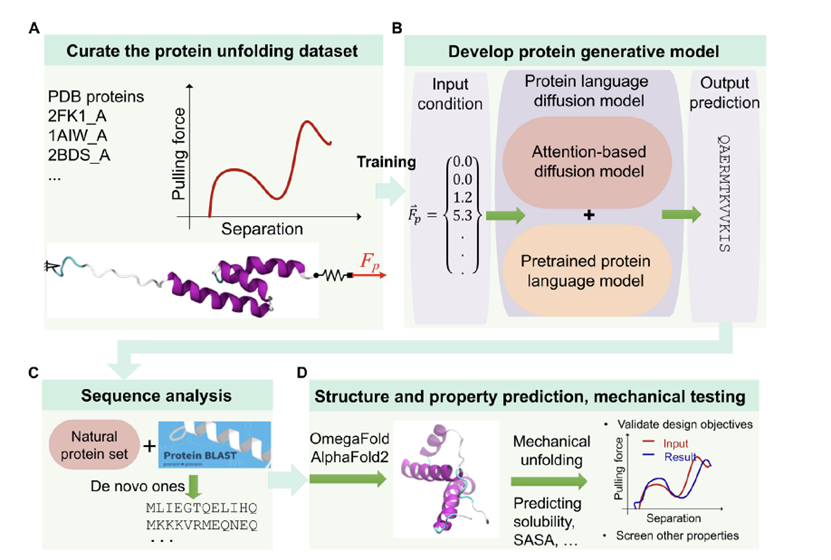
图 1
在一个独特的工作流程中(图1),作者首先进行一系列全原子分子动力学(MD)模拟,以模拟蛋白质数据银行(PDB)蛋白质的机械展开过程并记录力响应(图1A)。然后通过使用预训练的蛋白质语言模型(pLM)将蛋白质序列转换成一个词概率潜在空间,并训练一个扩散模型来学习序列表示和力-分离响应之间的映射,从而构建一个蛋白质语言扩散模型(pLDM)(图1B)。在部署时,训练好的pLDM基于给定的展开力条件预测序列候选,而集成的折叠算法(即OmageFold)确定结果序列的3D结构。为了验证,作者将设计的序列与已知蛋白质进行比较,以分析新颖性(图1C),并使用MD测试设计的蛋白质,以将其机械性质和展开响应与输入条件进行比较。为了准备设计流程以进行进一步的实验验证,可以使用现有的预测工具估计实验合成和测试的其他关键属性,如溶剂可及表面积(SASA)、溶解度或稳定性,以进一步筛选首选蛋白质候选(图1D)。通过严格控制的比较,作者展示了pLDM优于普通扩散模型。
通过力展开蛋白质的全原子建模
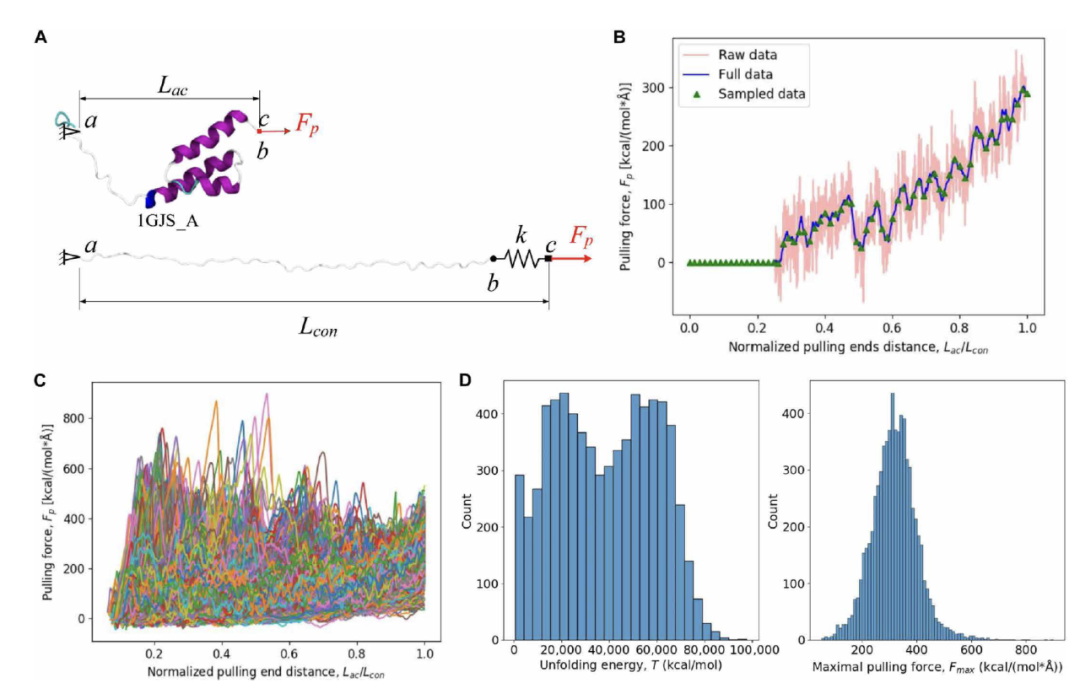
图 2
通过力展开蛋白质的全原子建模受到单分子力学的启发,作者模拟了在机械力作用下蛋白质链的展开过程,以理解其在分子层面的机械性质。如图2A所示,作者从具有实验测量的3D结构的PDB蛋白质开始。使用全原子动力学模拟和CHARMM力场以及一个广义Born隐式溶剂模型,作者首先在体温情况下(即310 K)下模拟蛋白质分子,达到平衡构象。然后通过固定一端并用弹簧(即图2A中b和c之间的部分)以恒定速度v = 0.1 Å/ps拉伸N个氨基酸的蛋白质链,弹簧的力常数k = 0.5 kcal/(mol Å^2)。拉力Fp每0.2 ps记录一次,直到两个拉力端之间的距离Lac达到蛋白质链的等长Lcon。在图2B中,作者平滑原始的力响应(红色曲线)以去除高频波动,得到蛋白质链的展开响应Fp(Lac)(蓝色曲线),从中可以使用展开能量T和最大力值Fmax定义蛋白质分子的韧性和强度。为了基于自然存在的蛋白质创建数据集,作者使用生物分子拉伸数据库(BSDB)作为指导,选择7026个PDB蛋白质,这些蛋白质在实验确定的结构中没有间隙,并且由不超过128个氨基酸组成。展开响应和机械性质的分布概览显示在图2(C和D)中。具体来说,在图2D中,展开能量或韧性显示出双峰分布,而强度呈现出单峰分布;在图2C中,可以观察到蛋白质之间存在各种展开响应。例如,最大力值可能出现在展开过程的中间或接近等长时的峰值,这可能表明非常不同的变形机制。通过使用基于单分子力学的生成结果的大量实验研究,也可以获得更多关于蛋白质展开状态的洞见。
模型部分
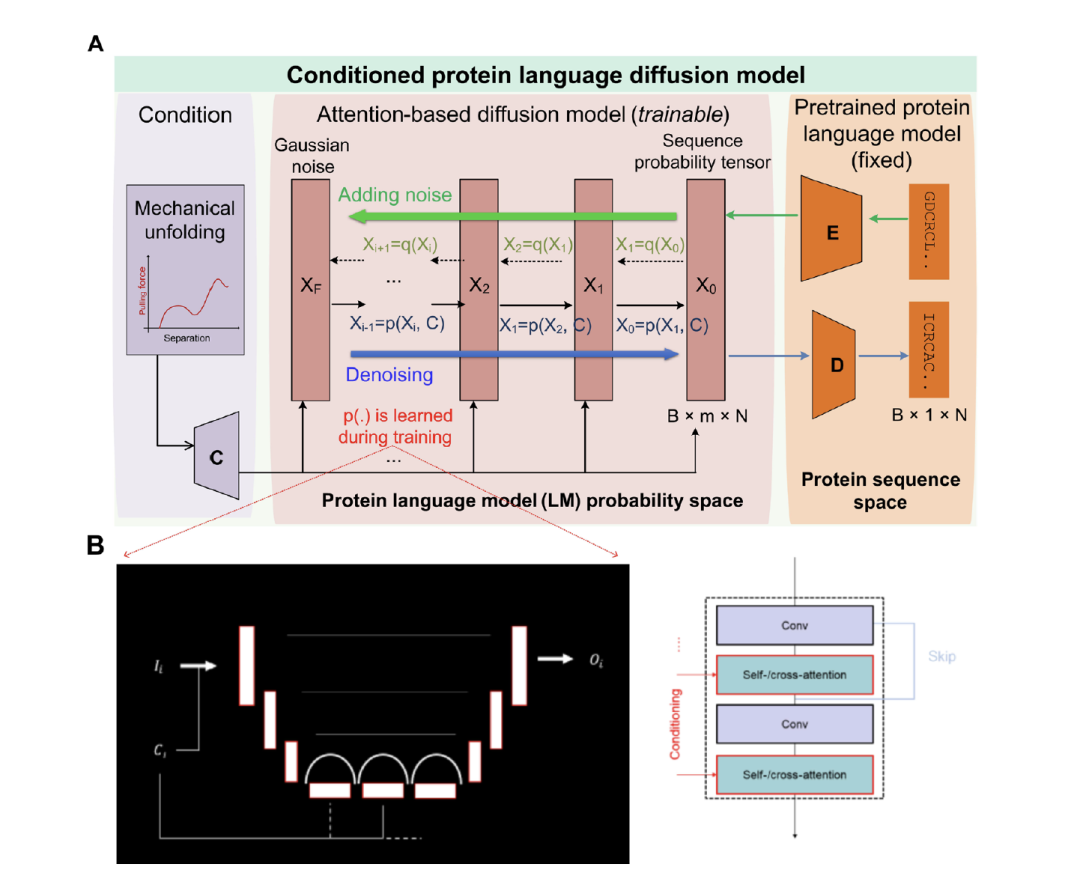
图 3
为了解决有条件的蛋白质设计任务,作者通过结合一个预训练的蛋白质语言模型(pLM)和一个基于注意力的扩散模型开发了一个蛋白质语言扩散模型(pLDM)。图3A展示了工作开发的模型概览。pLM(位于图3A右侧)在大量蛋白质序列数据上进行预训练,以形成内部表示,这些表示不仅能理解序列,还能理解蛋白质的结构和属性。作者利用这一知识,通过应用pLM将蛋白质序列从标记化序列空间转换到词概率潜在空间。然后训练一个在先前工作中开发的扩散模型在这个概率潜在空间中运行。扩散模型建立在一个具有注意力机制的一维U-Net架构之上(图3B)。在部署时,从给定条件(位于图3A左侧)和随机信号种子开始,扩散模型预测并在每一步去除噪声,生成有意义的序列概率张量,然后使用固定的pLM将其转换回蛋白质序列。一旦模型被训练,作者通过用各种机械展开响应测试开发的pLDM来展示其性能,包括来自自然存在的蛋白质和全新的蛋白质。生成的序列使用OmegaFold折叠成3D结构,然后使用全原子MD模拟进行相同的机械展开测试。通过蛋白质BLAST测试和与输入的拉力响应进行比较,作者检查生成序列的新颖性和蛋白质设计的准确性。

图 4
对于与自然存在的蛋白质相对应的机械展开响应的蛋白质设计,作者用测试集中PDB蛋白质的拉力记录测试模型,模型未经过这些数据的训练。图4展示了一些设计蛋白质及其机械展开响应的例子。在设计目标方面,图4(A至F)中有条件的拉力路径(红色曲线)代表了不同的模式,包括简单的示例,显示拉力在展开过程中几乎持续增加(图4D),在整体增加趋势的早期阶段展现局部峰值的示例(图4,A,B和F),达到振荡平台然后增加的(图4E),以及在展开初期阶段达到高峰的(图4C)。尽管蛋白质展开场景的复杂性和拉力的振荡性质,作者的模型生成的蛋白质展示了拉力响应(蓝色曲线),总体上紧密跟随设计目标。
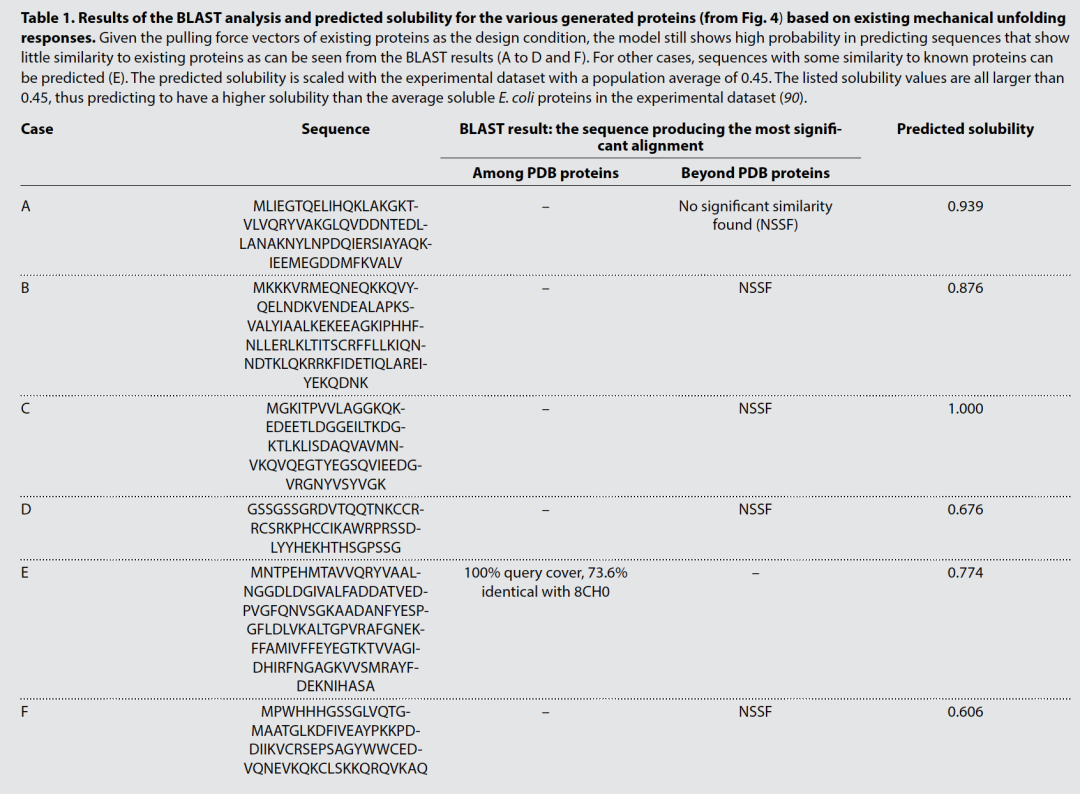
表 1
对于这些生成蛋白质的新颖性,作者对预测的氨基酸序列进行基本局部对齐搜索工具(BLAST)分析,以评估它们在多大程度上代表全新序列或已知蛋白质的密切相关形式。图4列出的各种案例的BLAST分析结果显示在表1中。我们发现,尽管输入设计目标来自现有的PDB蛋白质,许多生成的蛋白质序列(图4中展示的案例,A至D和F)在已知蛋白质的数据库中没有匹配任何序列(即,在蛋白质BLAST测试中返回“未发现显著相似性”),它们是全新的。该模型还可以产生序列(例如,图4中的案例E),显示出与现有蛋白质的一些相似性。然而,发现的最相似示例(即,8CH0)未包含在训练和测试集中。虽然模型只在PDB蛋白质的非常小一部分上进行训练,以现有PDB蛋白质的拉力作为输入,作者预期模型有可能“重新发现”与已知蛋白质显示出一些相似性的序列。
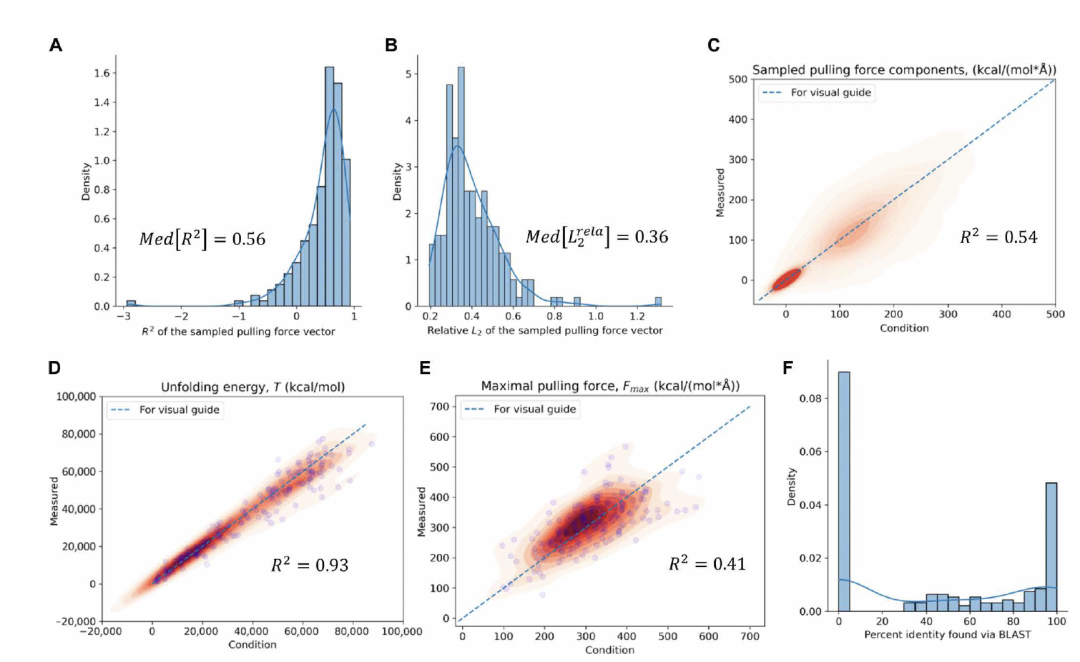
图 5
除了检查个别案例外,作者还展示了更多测试案例中设计准确性和新颖性的分布情况。图5展示了基于独立测试集中各种拉力条件的187个生成蛋白质的结果。在机械展开响应方面,生成蛋白质的测量拉力向量与输入条件之间的R^2值和相对L2误差在案例中显示出单峰分布,中位数分别为0.56和0.36(图5,A和B)。这些分布表明,在许多案例中,设计的蛋白质在整个机械展开过程中的趋势和值方面相当好地遵循输入条件。然而,如图4中的个别案例所示,在每个展开步骤精确遵循输入拉力值仍然是一个挑战。通过比较图5C中所有案例的拉力分量也可以看出这一点。有限的分量级准确性展示了基于详细机械展开响应设计蛋白质的难度和挑战,即使是使用当前模型。同时,作者的模型生成的蛋白质在实现和有条件的机械属性之间仍然表现出合理的一致性,包括韧性(图5D)和强度(图5E)。以拉力的最大值定义的强度显示出R^2值为0.41(图5E),略小于拉力分量的R^2值(如图5C中列出的0.54)。同时,韧性的R^2值为0.93,远高于拉力分量的R^2值(图5C)。R^2值的这种差异表明,当考虑整个展开过程时,分量级误差倾向于相互抵消,设计的蛋白质在韧性方面更敏感地遵循输入条件。在设计蛋白质的新颖性方面,图5F显示了通过蛋白质BLAST分析找到的所有生成序列的最高百分比同一性的双峰分布。最高峰(图5F左侧)对应于生成的蛋白质与现有/已知的蛋白质几乎没有相似性,完全是全新的情况。另一个较弱的峰出现在右侧,对应于生成的蛋白质与已知的蛋白质相似的情况。双峰分布呼应了表1中列出的个别案例的结果,两个峰的相对高度表明模型在生成全新序列设计方面有更强的倾向。
编译 | 曾全晨
审稿 | 王建民
参考资料
Ni, B., Kaplan, D. L., & Buehler, M. J. (2024). ForceGen: End-to-end de novo protein generation based on nonlinear mechanical unfolding responses using a language diffusion model. Science Advances, 10(6), eadl4000.



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


