
稿件来源
aKun
01
引言
自AlphaFold2(AF2)以来,相关的蛋白质结构预测方法如ESMFold、RoseTTAFold(RF)已经可以通过只利用序列信息就完成高精准的蛋白质结构预测。然而在自然界中,蛋白质并不单独存在,在各种生物过程当中它们与不同的小分子物质进行交互作用,比如与DNA和RNA交互实现转录和编译、在代谢过程中与各种化合物结合。然而预测蛋白质与任意一种小分子间结合结构都极具挑战,一种传统的做法是首先针对氨基酸序列利用AF2等方法构建蛋白质结构,然后利用传统docking方法加入小分子基于能量进行迭代优化,得到最终交互构象。尽管也有RoseTTAFold nucleic acid[1]方法将词表由氨基酸拓展到核酸,但面对高度变化的任意种类小分子仍十分有限。长期以来,领域内亟需一种端到端的仅利用小分子组成信息,非3D结构信息就可实现任意种类小分子的交互结构预测的方法。
近期,David Baker团队提出了RoseTTAFold All-Atom(RFAA)[2]方法,依托于RoseTTAFold本身的网络框架,小分子相关信息被合理注入。RFAA在蛋白质结构预测准确性方面与AF2相媲美,在柔性骨架小分子对接方面同样表现出色,可以对于蛋白质共价修饰以及多个核酸链和小分子组装进行合理预测。更进一步依托于RoseTTAFold网络架构上提出的RFDiffusion[3]得到再次提升,在相同处理小分子策略下通过对扩散去噪任务进行微调,新版本RFDiffusion All-Atom(RFdiffusionAA)可以直接在小分子和其他非蛋白质分子周围构建蛋白质结构来生成结合口袋。
02
架构解析
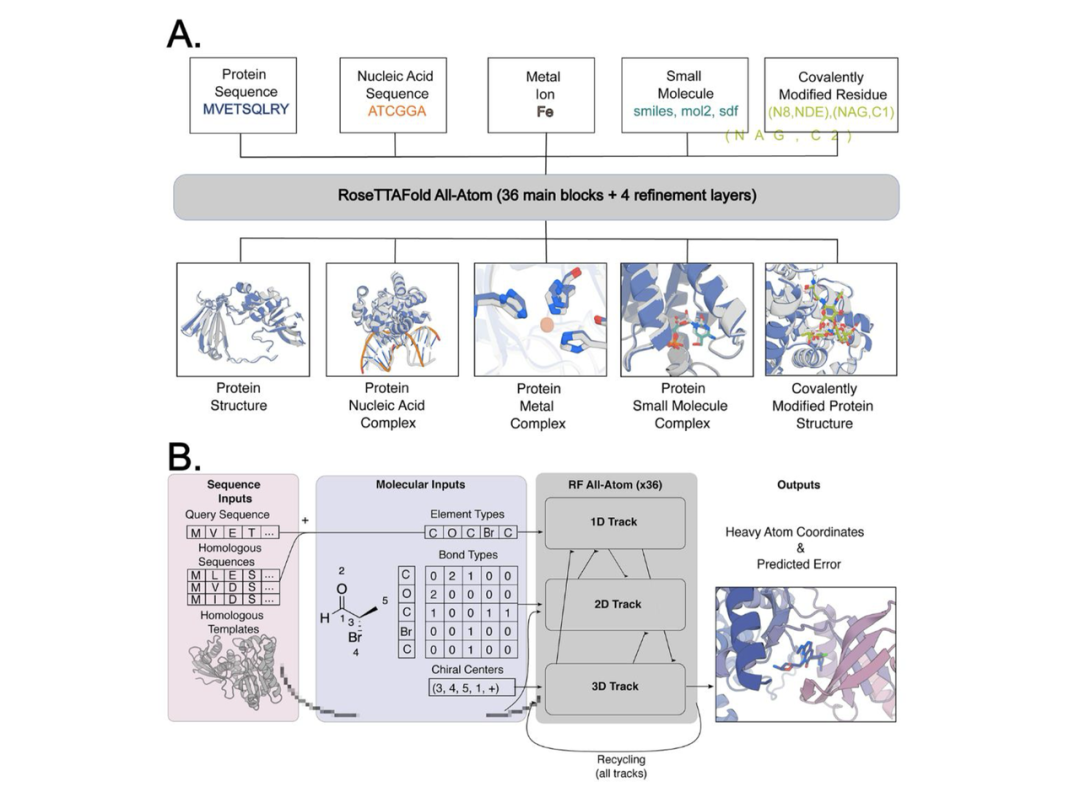
RFAA框架如上图所示。注入小分子信息的首个难题就是缺乏一种合理的表示方法与与之配套的网络框架。小分子难以像蛋白质通过线性序列的形式进行表示,更自然的是以图的形式:节点表示原子,边表示键连接。RFAA从两方面,序列描述与小分子和蛋白共价修饰的原子图描述结合起来,共同表示聚合物。
具体来说,针对小分子图可以采用一种合适的图遍历方式将重原子进行编号,根据标号顺序对应的元素种类可以得到一维的表示,这部分信息将与原本的蛋白序列与MSA融合共同输入编码序列信息的1D轨道;对应的二维信息则表示两个节点之间化学键种类,这种成对信息将输入处于成对距离关系信息的2D轨道。
由于以上一维和二维表示对于反射是不变的,作者团队利用指定手性中心周围原子之间角度在3D轨道中加入立体化学信息。此外对于小分子重原子的坐标只更新偏移量。整个网络主要采用了全原子版本的FAPE损失函数进行优化。
03
应用测试
3.1 小分子蛋白复合物预测性能
为了进行盲性测试,作者团队在CAMEO盲性配体对接评估中注册了RFAA服务器。对新提交到PDB的结构进行预测并评估发现,RFAA对43%预测为高置信(PAE Interaction<10),高置信中77%预测的配体RMSD<2埃。同时对比AutoDock Vina [4]的成功建模率8%(配体RMSD<2埃),RFAA成功建模高达33%。当给定口袋位置与侧链晶体结构时,AutoDock Vina方法又重回第一(52% VS RFAA 42%)。
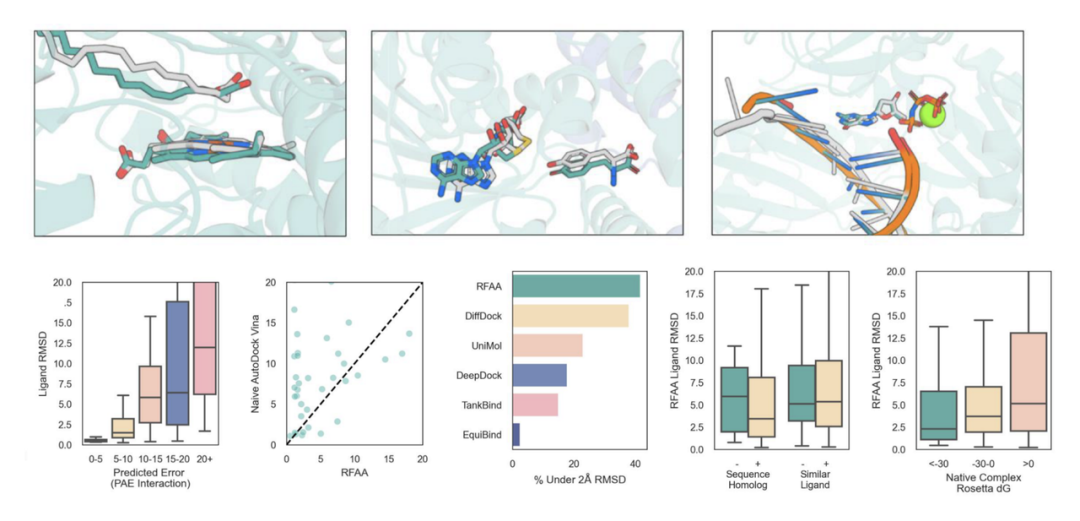
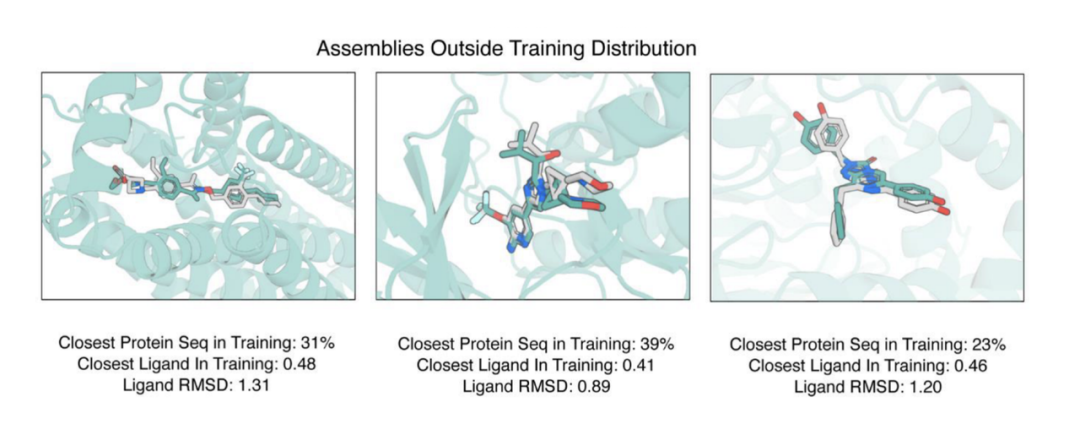
在性能泛化方面,非训练集的蛋白小分子复合物结构经过RFAA的预测rmsd能达到小于1.5埃的级别,使用Rosetta GALigandDock对设计的小分子蛋白质复合物构象进行预测,其物理能量与蛋白质晶体结构类似。这表明模型学习到了小分子与氨基酸互作的基本规律。
3.2 RFdiffusion-AA从头设计
在RFdiffusion的基础上,RFdiffusionAA旨在给定非蛋白分子的条件下实现蛋白质设计。通过训练使用RFAA结构预测权重初始化的扩散模型,使用蛋白质-小分子数据集对以小分子为条件下对加噪的蛋白结构进行去噪。
与RFdiffuison类似,作者团队发现在去噪过程中引入辅助潜在导向影响采样轨迹可以增加小分子与binder之间接触,产生更紧密的界面。
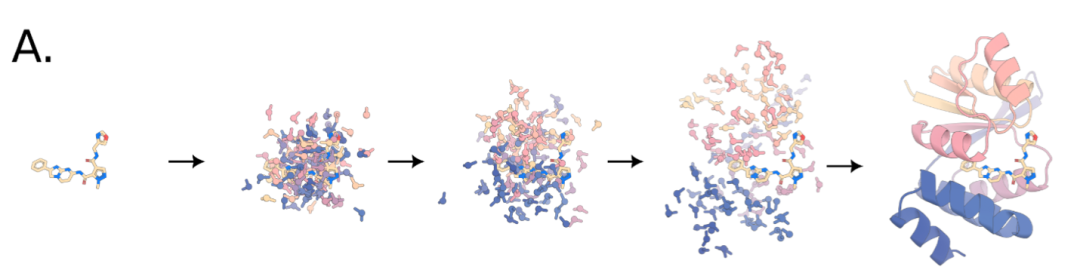
RFdifusion All-Atom扩散过程
由于RFdiffusion-AA只会产生蛋白质的主链,对于设计的蛋白结构,作者团队首先采用LigandMPNN [5]进行序列设计。并使用Rosetta GALigandDock对设计的小分子蛋白质复合物构象进行预测,评估设计的正确性。
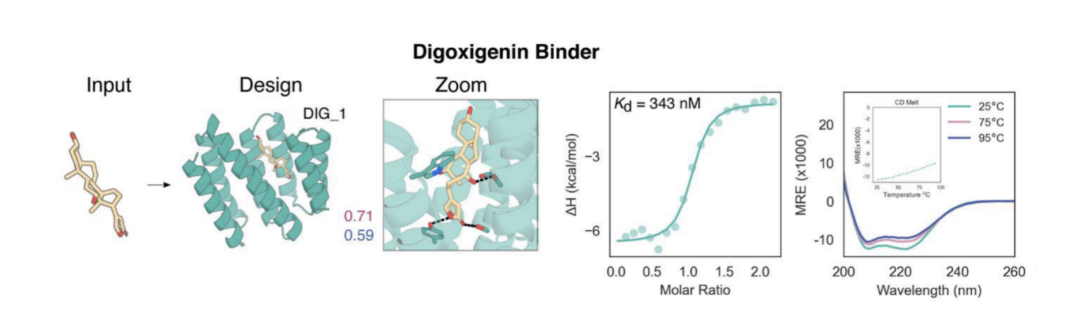
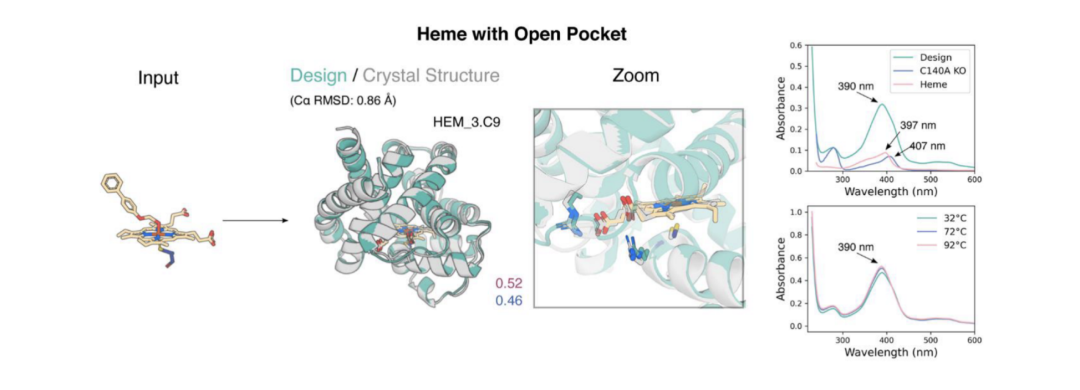
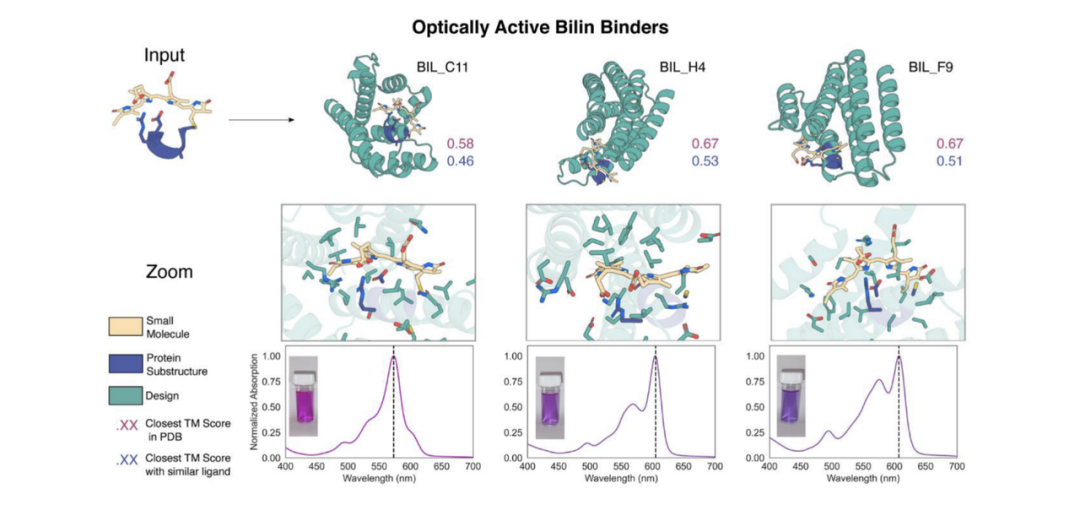
结果表明新版本RFDiffusionAA表现水平大幅度提升。作者团队针对三种不同的小分子结构进行蛋白binder设计,不仅设计的出能高效结合小分子的能力,同时其在蛋白质的稳定性方面极大地超越天然蛋白质,为工业级蛋白质设计提供了新的机会。
04
力评
在深度学习发展过程中,小分子模型与大分子模型往往是并轨发展的,如今蛋白质领域迎来了“Alphafold3”时刻,正式进入了全原子的时代。
在扩散模型的快速发展下的加持下,全原子模型的蛋白质/小分子设计场景得到了巨大的扩展,相信在不久将来,随着模型不断进步和发展,将会有更多的药物和全新品类的功能蛋白质将进入到人们的生活中。
开源代码链接:
https://github.com/baker-laboratory/rf_diffusion_all_atom
https://github.com/baker-laboratory/RoseTTAFold-All-Atom
参考文献
-
M. Baek, R. McHugh, I. Anishchenko, D. Baker, F. DiMaio, Accurate prediction of nucleic acid and protein-nucleic acid complexes using RoseTTAFoldNA. bioRxiv (2022), p. 2022.09.09.507333.
-
Krishna, R. et al. Generalized biomolecular modeling and design with RoseTTAFold All-Atom. Science eadl2528 (2024) doi:10.1126/science.adl2528.
-
Watson, J.L., Juergens, D., Bennett, N.R. et al. De novo design of protein structure and function with RFdiffusion. Nature 620, 1089–1100(2023). https://doi.org/10.1038/s41586-023-06415-8
-
J. Eberhardt, D. Santos-Martins, A. F. Tillack, S. Forli, AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. J. Chem. Inf. Model. 61, 3891–3898 (2021).
-
J. Dauparas, I. Anishchenko, N. Bennett, H. Bai, R. J. Ragotte, L. F. Milles, B. I. M. Wicky, A. Courbet, R. J. de Haas, N. Bethel, P. J. Y. Leung, T. F. Huddy, S. Pellock, D. Tischer, F. Chan, B. Koepnick, H. Nguyen, A. Kang, B. Sankaran, A. K. Bera, N. P. King, D. Baker, Robust deep learning–based protein sequence design using ProteinMPNN. Science. 378, 49–56 (2022).
2024丨力文所
EXPLORE EVOLUTION
DECIPHER LIFE
©️ 力文所原创内容,未经许可转载必究。
欢迎给力文所LEVINTHAL公众号 标星
在文末右下角点击 在看
给本文作者 点赞






 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


