今天为大家介绍的是来自Ava P. Amini团队的一篇论文。文章讲述了一种计算方法,这种方法能够生成新的、在物理上可折叠的蛋白质结构,这对于新的生物学发现以及针对当前还无法治愈的疾病的新疗法具有重要意义。

蛋白质对生命至关重要,几乎参与到生物体内的每一个生物学过程中,从神经元之间的信号传递到识别微观入侵者并随后激活免疫反应,从产生能量到沿着细胞高速公路运输分子。然而,行为异常的蛋白质会导致人类健康领域一些最具挑战性的疾病,包括阿尔茨海默病、帕金森病、亨廷顿病和囊性纤维化等。由于蛋白质能够以高特异性执行复杂功能,它们被广泛研究作为治疗介质,并构成批准疗法的快速增长部分。因此,计算生成新的且在物理上可折叠的蛋白质结构的能力,可能为发现利用细胞途径的新方法打开大门,并最终导致针对尚未治愈的疾病的新疗法。
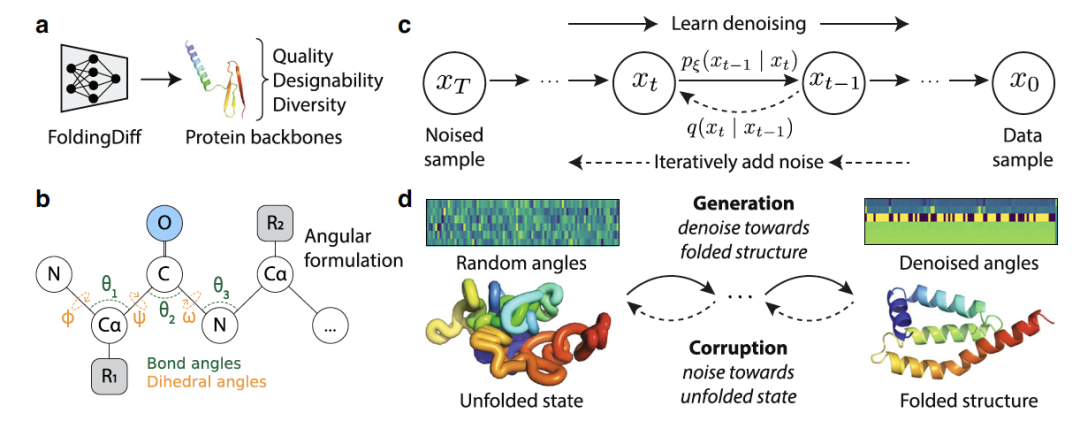
图 1
在这里,作者受到蛋白质折叠过程生物物理学的启发,介绍了一个作用于蛋白质主链中残基间角度而非笛卡尔原子坐标的生成模型(图1)。这种方法将每个残基视为一个独立的参考框架,从而将等变性要求从神经网络转移到坐标系统本身。作者使用一个简单的transformer参数化的去噪声扩散概率模型(简称扩散模型),这些模型在从图像到音频的广泛数据模态上都非常成功,并且与像生成对抗网络(GANs)等方法相比,训练起来更容易,模态覆盖更好。结合这些想法,作者的框架通过从一组随机角度开始,这些角度对应于一个随机的、未折叠状态,并迭代地去噪声化底层角度,到达一个最终的主链结构。尽管这种角度去噪声过程并没有直接捕捉到任何生物物理折叠过程,但它从蛋白质如何扭曲并折叠成最终结构中汲取了灵感;因此,作者将这种方法命名为FoldingDiff。
设计训练FoldingDiff
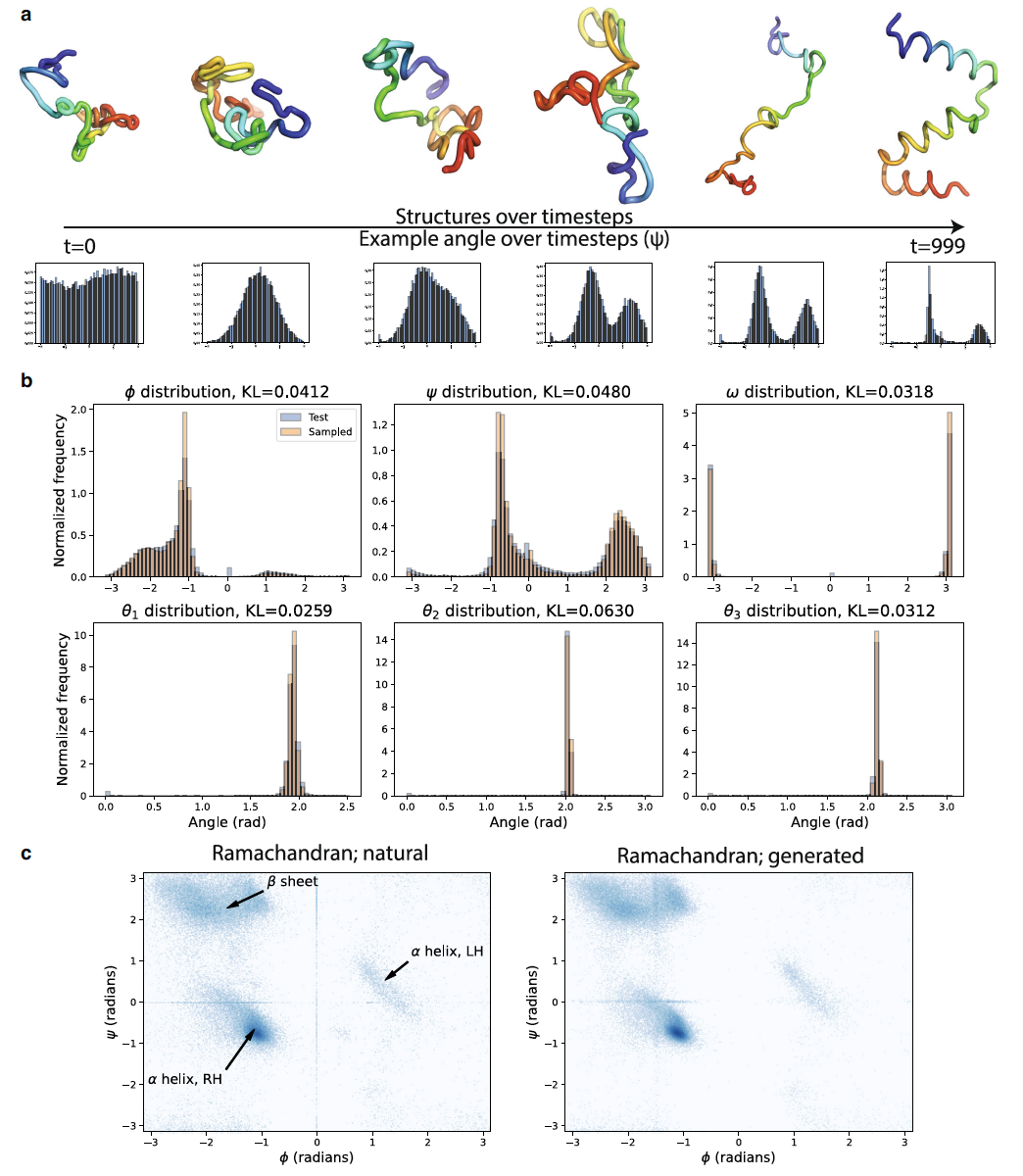
图 2
设计和训练FoldingDiff的过程包括定义了一种简化但完整的基于角度的蛋白质结构表示方法。作者设计了一种去噪声扩散概率模型(简称扩散模型),能够从随机噪声生成蛋白质主链角度。为了学会这样做,扩散模型被训练以迭代去除数据中的噪声。在训练过程中,从一个数据样本x0开始,噪声在T个离散步骤中被迭代添加,直到它与xT处的随机噪声无法区分。在图1c中,这个添加噪声的过程是通过马尔科夫过程实现的。扩散模型被训练以预测每一步添加的噪声,学习一个执行反向去噪声过程的模型。训练完成后,为了生成新的数据点,扩散模型从随机噪声开始,并应用T步的迭代去噪声,其中每一步去噪声的输出用于准备下一个去噪声周期的输入,最终得到一个干净的样本。重要的是,这个添加噪声和去噪声的过程并不模拟蛋白质折叠的任何生物物理过程。作者用一个双向transformer模型来表述去噪声模型,并设置T = 1000个添加噪声步骤。值得注意的是,这种transformer架构并不提供旋转或平移等变性,因为输入表示本身就是固有的旋转和平移不变性。FoldingDiff在CATH蛋白质域的数据集上进行训练,这些蛋白质域的长度在40到128个残基之间;少于40个氨基酸的结构被丢弃,而超过128个残基的结构在每个训练周期中被随机裁剪。总的来说,作者成功地使用30,395个独特的蛋白质域训练了我们的模型,随机分为训练、验证和测试集,比例为80/10/10。
模型基本评估
在训练完FoldingDiff模型之后,作者首先验证了FoldingDiff能够生成蛋白质中二面角和键角的逼真分布。作者无条件地为每个长度区间范围生成了10条主链,总共生成了780条主链。为了确保模型生成的角度在不同蛋白质之间是普遍适用的(而不仅仅是从训练数据集中记忆来的),作者将这780个结构的角度分布与训练期间未见过的实验结构的测试集的角度分布进行了比较。为了匹配生成的主链长度,测试集也只包含长度小于128残基的结构。最后观察到,对于所有角度,生成的分布几乎完全复制了测试分布(见图2b)。与其他蛋白质扩散方法生成的类似图表相比,作者定性观察到模型产生了更紧密的分布,更接近于键角的自然分布。然而,每个角度的单独分布并不足以展现这些角度的相关性。为了测试模型是否正确捕捉到这些相关性,作者制作了Ramachandran图,展示了二面角ϕ和ψ的联合分布。图2c展示了实验确定的测试集的Ramachandran图,与生成的780个结构进行了比较。自然结构的Ramachandran图包含三个主要集中区域,分别对应于右手α螺旋、左手α螺旋和β折叠。作者生成的结构复制了这三个区域(见图2c),表明FoldingDiff能够生成蛋白质主链中的所有三个主要二级结构元素。此外观察到模型正确地学会了右手α螺旋比左手α螺旋更常见,表明FoldingDiff学习并尊重了蛋白质结构的手性。
FoldingDiff生成的结构特征
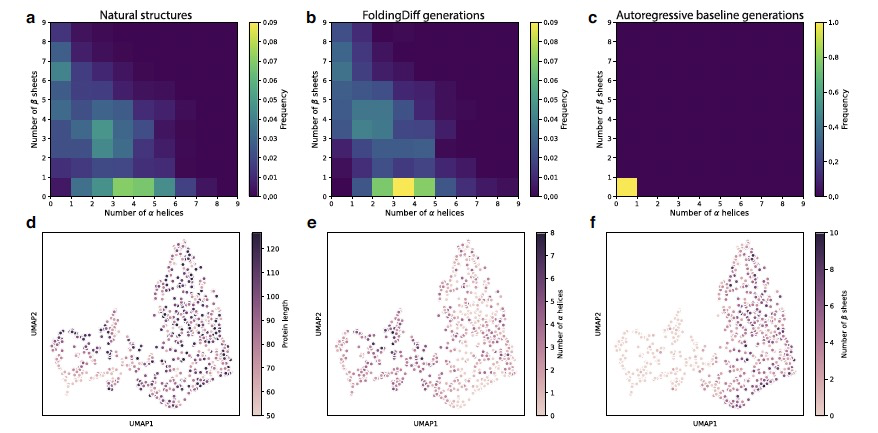
图 3
结果显示FoldingDiff能够生成其个体和联合分布与自然蛋白质结构相匹配的角度。然而,这些之前的评估仅仅是检查了个别残基对是否形成了与二级结构片段一致的角度,并没有评估蛋白质的整体二级结构组成是否在生物学上合理。因此,作者评估生成的结构中二级结构元素的数量和共存是否与自然主链中观察到的相匹配。为此,作者使用了P-SEA,一种计算算法,用于为每个主链注释二级结构元素。将P-SEA应用于自然结构测试集和生成的主链上,计算了检测到的α螺旋和β折叠的数量,并测量了这些二级结构的共存频率(图3a, b)。与自然结构类似,生成的结构经常包含多个二级结构元素,并展现出与自然结构类似的共存模式(例如,平均而言α螺旋比β折叠更常见)。为了更细致地理解FoldingDiff生成的蛋白质主链结构的类型,作者通过它们的嵌入来可视化生成的蛋白质的景观。具体来说,作者使用PHAISTOS软件将生成的蛋白质进行嵌入,然后使用UMAP技术将其投影到二维空间以进行可视化。作者根据几个描述符——长度、螺旋数和折叠数——对这个图进行了注释,并观察到生成的主链的设计空间覆盖了这些描述符的广泛范围(图3d–f)。将这些嵌入与类似长度的CATH测试集结构共同可视化显示,FoldingDiff的生成物与自然结构共享重叠区域,同时也探索了自然结构稀少占据的嵌入空间。这表明FoldingDiff有潜力抽样出与已知结构相似范围的主链。
设计性
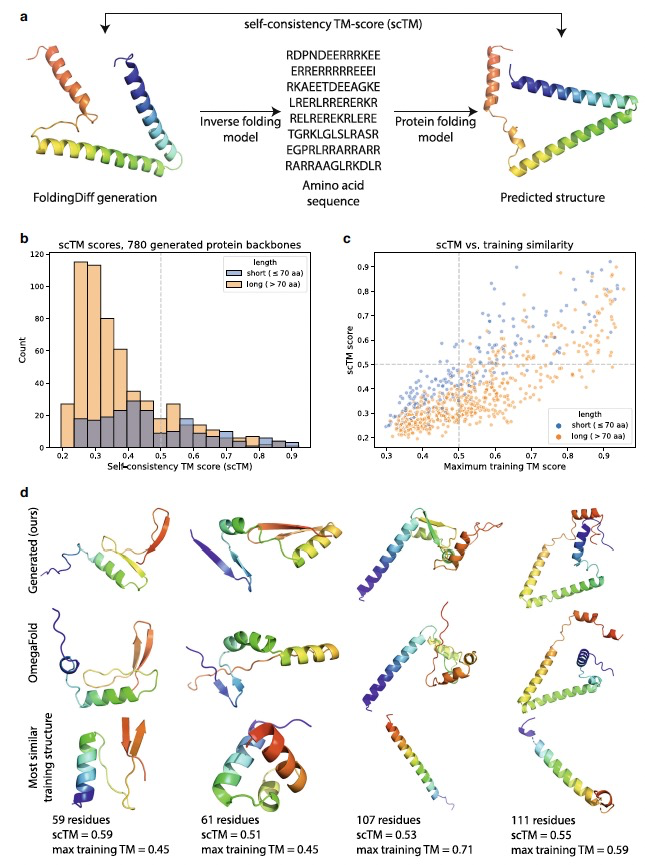
图 4
在评估了生成的结构的生物学可行性后,作者接下来探讨了FoldingDiff生成的结构是否具有设计性。在蛋白质设计中,结构的设计性反映了是否能够使用当前方法确定一个可能折叠成该指定主链结构的氨基酸序列。能够产生高比例设计性结构的生成模型对于下游蛋白质工程应用更为有用。之前的工作通过预测可能折叠成生成主链的氨基酸链,并评估这些序列产生的结构是否与原始主链匹配来在计算上评估设计性。由于实验验证的资源密集性,大多数工作通过机器学习模型预测的序列结构来比较生成的结构(图4)。为了为生成的结构生成候选氨基酸序列,作者使用ProteinMPNN逆折叠模型输出8个不同的候选序列。对每个序列使用OmegaFold结构预测方法预测相应的3D结构,并通过计算它们的TM得分来评分原始生成的主链和预测结构之间的结构相似性,TM得分范围从0到1,数值越大表示相似性越高。TM得分≥0.5通常表明两个主链处于相同的蛋白质折叠中,作者将scTM≥0.5视为自洽且因此可设计的。使用这一程序,作者发现780个结构中有177个,即22.7%,具有scTM得分≥0.5的设计性,无需任何后处理。与此前的工作相比,FoldingDiff在短序列(最多70个残基)和长序列(超过70个残基)的设计性上都有所提高。作者还发现OmegaFold和AlphaFold2产生的置信度得分与scTM得分良好相关,这表明利用FoldingDiff的应用可以直接使用这些得分来识别高质量的生成物。
编译|曾全晨
审稿|王建民
参考资料
Wu, K.E., Yang, K.K., van den Berg, R. et al. Protein structure generation via folding diffusion. Nat Commun 15, 1059 (2024).
https://doi.org/10.1038/s41467-024-45051-2



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


