——简介——
如何高效采样蛋白质尤其是天然无规蛋白质(IDP)的多种结构一直是计算结构生物学领域的一大难点。通常,此类问题是通过分子模拟(包括MC采样和MD采样)来解决,同时配合各种增强采样算法提高效率。近年来,得益于GPU算力的进步,各种基于深度学习的增强采样和构象生成算法被提出,但大都需要针对某一体系预先的MD轨迹数据,没有办法直接从序列生成具有多样性的构象。2024年1月,来自蒙特利尔大学Mila研究院Jian Tang课题组的Jiarui Lu等人开发了基于分数模型蛋白质zero-shot构象采样模型Str2str[1]。该工作目前在ICLR2024上作为Poster发布。该模型通过输入蛋白序列和结构(例如使用AlphaFold预测的结构),在一个分数模型上加噪再去噪,从而采样新的构象。该工具不同于之前的AI辅助增强采样方法和构象生成方法,不需要对结构提前进行MD采样,大大扩展了模型的使用范围。
——方法——
Str2str全称structure to structure,其基本逻辑如下图1所示:先利用蛋白质序列–结构数据集训练一个具有平移旋转等变性的分数模型;之后针对某一输入的蛋白序列通过结构预测模型如AlphaFold2等预测一个初始结构;将结构按一定步数不完全加噪;利用训练好的模型对结构进行去噪;最后得到的结构即为生成后的新结构。
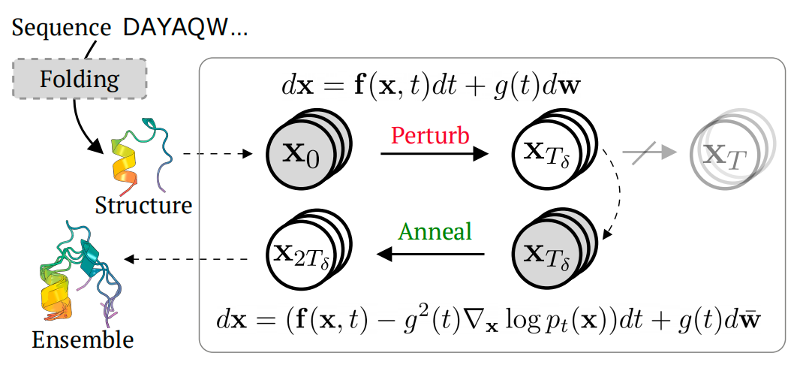
图1. Str2str的运行逻辑
这个先不完全加噪再去噪的过程被作者称为forward-backward过程(FB),具体公式如下:
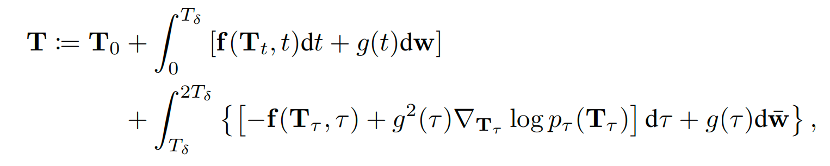 其中,Tδ取值在0到T之间,并且可以通过控制其大小,来达到控制生成构象集合(ensemble)的结构多样性的目的。
其中,Tδ取值在0到T之间,并且可以通过控制其大小,来达到控制生成构象集合(ensemble)的结构多样性的目的。
在训练时,分数模型的随机微分方程(SDE)如下所示:
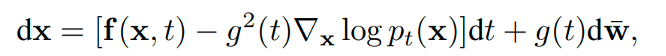 其中dt是时间步,dw是布朗运动项。
其中dt是时间步,dw是布朗运动项。
为了更好的训练效果,作者仿照AlphaFold2的处理方法来表征蛋白结构:将蛋白质按氨基酸为单位划分为主链“frame”以及侧链各种扭转角。其中,对于蛋白主链的表征定义如下:
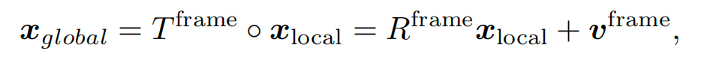
其中,Xglobal代表蛋白的笛卡尔坐标,Xlocal代表在“frame”参考系下的坐标。Tframe可分解为一个旋转项Rframe和一个平移项vframe。而对于侧链,则仿照AF2的侧链对照表处理,本推文暂不赘述。
至于去噪的神经网络架构,作者使用了一个更改后的IPA网络,被作者命名为DenoisingIPA,如下图所示:
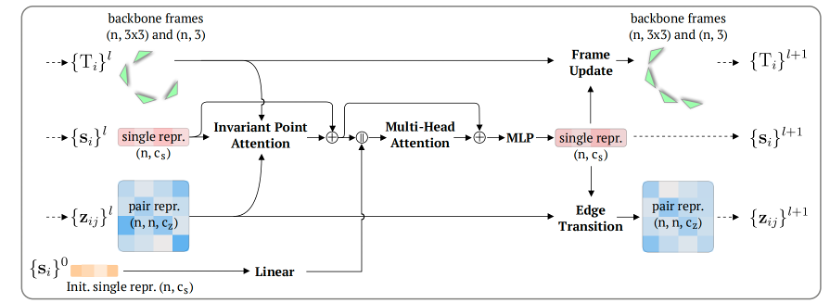 图2. DenoisingIPA的网络架构
图2. DenoisingIPA的网络架构
可见,基本与AF2中的IPA模块相同,其中Si张量来源于positional encoding和分数模型的Time step encoding。此外,作者在其中加入了一个pair representation的transition模块,类似于AF2的Evoformer中所使用的edge transition模块,如下:
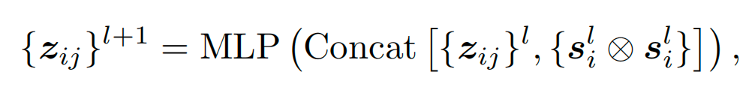
值得注意的是,不同AF2的IPA模块,这个架构中其实并没有嵌入序列信息,所以其实蛋白序列的唯一作用是在一开始通过AF2或ESMFold等工具预测初始结构。在训练过程中,作者是用了两种loss函数来保证训练效果。其一是正常分数模型的loss函数:
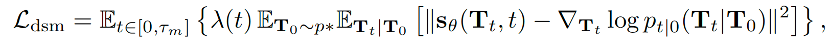 与正常分数模型训练过程不同的是,由于在实际使用时,并不需要用到全部的扩散步数,因此可以只对0到Tm的区间算loss,其中Tm是一个提前设定的超参数,0<Tm<T。
与正常分数模型训练过程不同的是,由于在实际使用时,并不需要用到全部的扩散步数,因此可以只对0到Tm的区间算loss,其中Tm是一个提前设定的超参数,0<Tm<T。
另一种loss被称为Auxiliary Loss,由主链MSEloss:
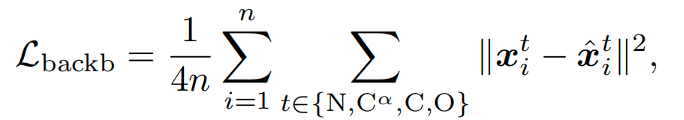
和原子距离图损失distogram loss:
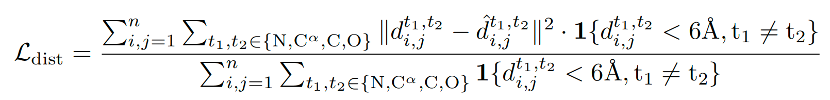
这两种loss在t<T/4时被计算。
最后,总loss如下:
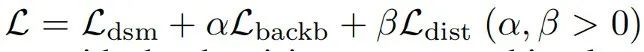 其中,作者将alpha和beta取值为0.25。
其中,作者将alpha和beta取值为0.25。
——结果——
之后,作者对Str2str的效果进行了benchmark。Benchmark的对象主要包括AF2-RVAE、EigenFold、idpGAN等。评价标准主要包括:1.有效性:包括原子clash和化学键键长。2.保真度:两两距离分布的JS散度(JS-PwD)和tICA降维图上的最近组分(JS-TIC)以及回旋半径Rg分布的JS散度(JS-Rg)。3.多样性:包括RMSD和TM-score的MAE。
Benchmark结果如下表1所示,其中PF表示使用概率流进行采样的结果:
表1. Str2str的benchmark结果
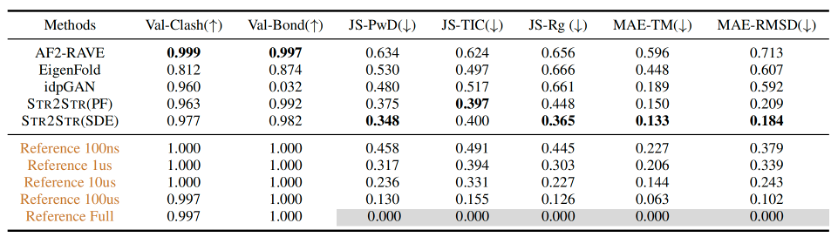 可见,与之前的EigenFold和idpGAN相比,Str2str不仅在有效性和保真度上有优势,在多样性上超过之前的方法很多,同时和MD的结果进行比较也能发现,单从采样效率上讲,Str2str的似乎媲美几十us级的MD采样。
可见,与之前的EigenFold和idpGAN相比,Str2str不仅在有效性和保真度上有优势,在多样性上超过之前的方法很多,同时和MD的结果进行比较也能发现,单从采样效率上讲,Str2str的似乎媲美几十us级的MD采样。
除了量化的benchmark之外,作者对很多MD采样领域比较著名的case进行了具体的分析,包括很多的fast-folding蛋白如:BPTI、Trp-cage等。其中BPTI的tICA结果如下所示:
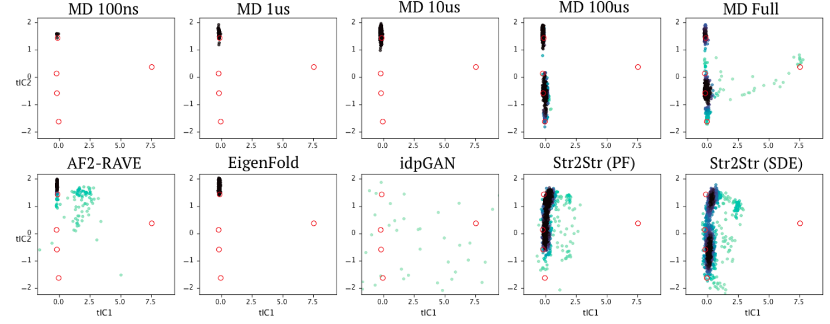
图3. BPTI的benchmark结果
可见,Str2str的生成结果和长时间MD更类似,而其他方法则和短MD的结果更为类似,体现了Str2str更高的采样效率。
Trp-cage的contact-map分布图如下图所示:
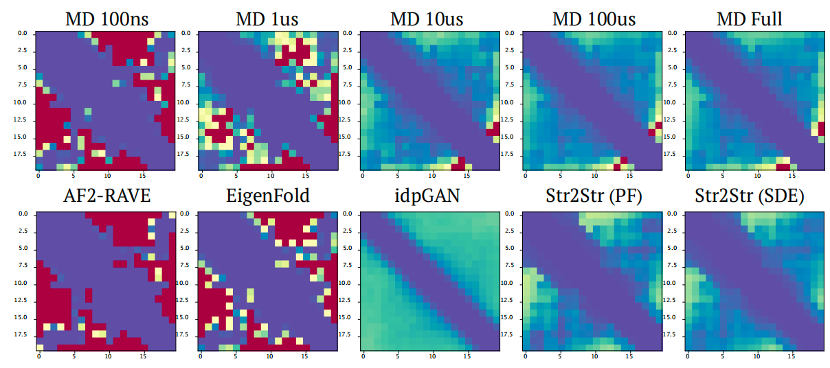 图4. Trp-cage的benchmark结果
图4. Trp-cage的benchmark结果
和BPTI的结果类似,Str2str展现了更类似于长时间MD的采样效率。
其他各种case的contact-map结果如下如所示:
 图5. 各种fast-folding蛋白的benchmark结果
图5. 各种fast-folding蛋白的benchmark结果
此外,作者还统计了在不同去噪步数的选取情况下Str2str的表现,展现了模型可以通过参数控制生成结果多样性的特点,如下图所示: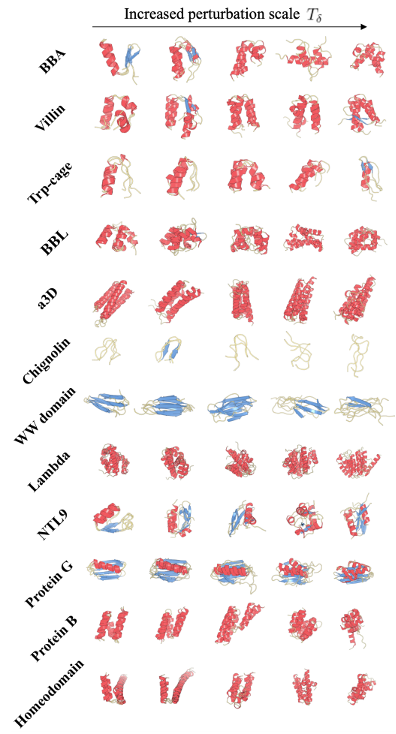
图6. 不同T_delta下的ensemble生成结果
比较有意思的一点是随着T_delta的增大,模型似乎能更容易采样到不同的构象。下图的tICA结果也说明了这一点:
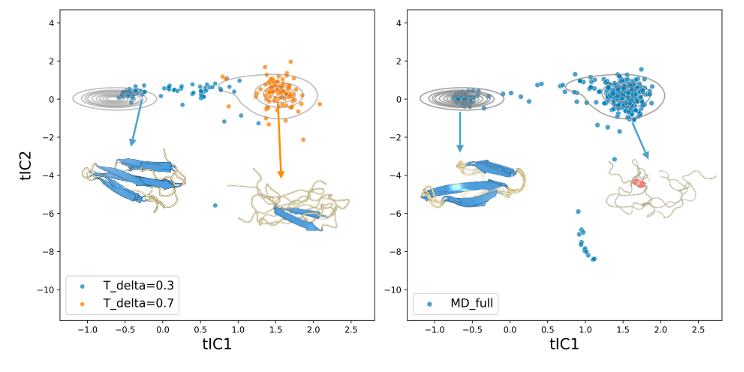 图7. 不同T_delta下的WW domain ensemble生成结果和MD结果比较
图7. 不同T_delta下的WW domain ensemble生成结果和MD结果比较
——小结——Str2str的提出不仅为我们提供了一种高效的AI采样方法,同时这种不完全加噪方式也为将扩散模型迁移到flow上提供了一些启示。此外,据作者言,这种不完全加噪再去噪的过程实际上暗合MC采样或MD采样中跨越势能垒的过程,同样引人深思。
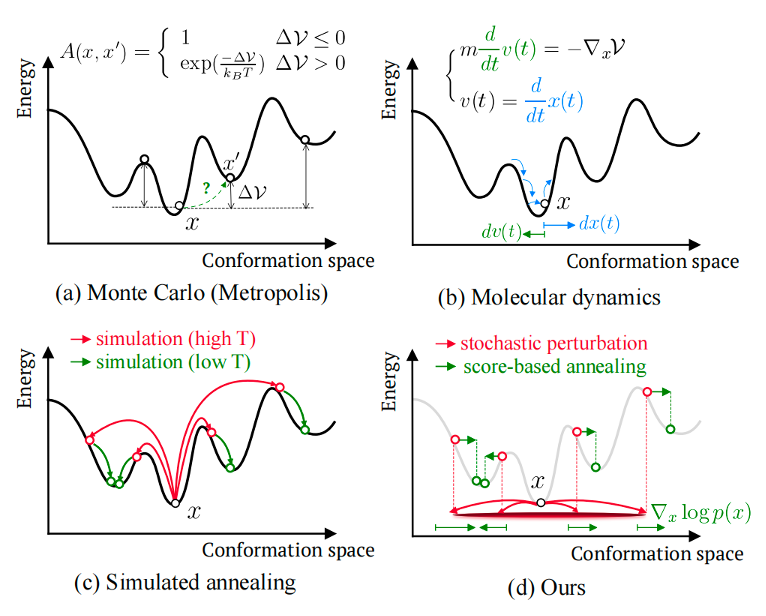
图8. Str2str与常规采样方法的能垒跨越过程类比
当然,就目前而言基于AI的增强采样方法仍然任重道远:比如,Str2str的结果显示,采样结构的比例和MD存在较大差距,这说明模型整体对于玻尔兹曼分布的感知还不足,只能作为初始结构采样器使用。其次,文章展现的都是一些fast-folding的蛋白,对于真正类coil的IDP蛋白缺乏足够的case study。
参考文献:
Lu, Jiarui, et al. “Str2str: A score-based framework for zero-shot protein conformation sampling.” The Twelfth International Conference on Learning Representations. 2024.
作者:穆俊羲审稿:王宇哲编辑:黄志贤
GoDesignID:Molecular_Design_Lab( 扫描下方二维码可以订阅哦!)



 ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง
ufabet
มีเกมให้เลือกเล่นมากมาย: เกมเดิมพันหลากหลาย ครบทุกค่ายดัง


