
1.介绍
药物联合治疗通常用于治疗复杂疾病如癌症。与单一治疗相比,药物联合治疗可以提高癌症治疗的疗效,降低药物的剂量依赖性毒性,防止耐药的发生。然而,药物组合不仅会产生协同作用,还可能产生拮抗作用。
发现新的协同药物组合的最大挑战之一是组合爆炸。虽然药物协同作用的机制已经被探索,但大多数协同药物组合是基于临床经验提出的,早期研究需要进行体内实验。这种基于试验的方法具有耗时、劳动密集和昂贵的缺点,并可能导致患者接受不必要甚至有害的治疗。
人工智能的蓬勃发展,包括机器学习和深度学习,促进了新的计算方法的提出。这些方法的一个优势是可以模拟复杂的非线性过程,其典型模式是首先构建细胞系和药物的特征,然后使用模型进行预测。GCN是专门为生物网络等图结构数据设计的,但目前只有少数实验使用GCN提取药物组合预测领域生物网络的信息特征,而且都没有考虑细胞系特征。
本文提出的PRODeepSyn方法提取PPI网络数据和组学数据的拓扑结构,构建具有GCN的细胞系的低维密集嵌入并预测药物组合的协同得分。本文已经验证了PRODeepSyn优于包括机器学习和深度学习方法在内的其他最先进的方法。总之,PRODeepSyn有望成为一种令人满意的抗癌协同药物联合预测方法。
2.方法
1.药物特征
为了表示药物的结构和理化性质,本文使用分子指纹和描述符来构建每种药物的特征向量。本文采用RDKit根据药物的SMILES表达式计算每种药物的分子指纹和描述符。本文为每种药物生成半径为2的分子指纹,并将其表示为256维二值向量,并且得到每个药物的200个描述符,组成一个实值向量。之后本文将上述两种类型的特征向量拼接起来,过滤掉方差为零的特征。最后保留253维分子指纹和163维描述符作为每种药物的特征,即药物的最终特征向量为416维。本文使用z-score归一化方法对药物特征进行预处理,以消除特征尺度可能产生的影响。
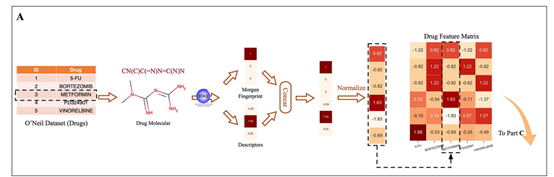
图1 PRODeepSyn构建药物特征
2.细胞系特征
PRODeepSyn整合了三种类型的异质性细胞系特征,包括基因表达数据、基因突变数据和基因表达产物之间的相互作用,以构建细胞系的嵌入。基因表达数据对总共3739个信息基因进行汇总,然后用z-score归一化方法进行处理。细胞系的基因突变数据来自COSMIC细胞系项目,本文删除了突变类型未知的数据,保留了39个细胞系的12333个基因的突变数据。每个细胞系的基因突变数据被表示为一个12333维二值向量。根据细胞系是否在某一基因上发生突变,使载体对应的元素为0或1。基因表达产物之间的相互作用从STRING数据库中包含的PPI网络中收集。本文忽略了STRING中综合得分低于0.7的相互作用,保留了17161个蛋白之间总共839522个相互作用。
3.基因隐藏状态的构建
考虑到基因表达的蛋白质之间的相互作用在药物联合治疗中很重要,PRODeepSyn利用GCN模型基于PPI网络构建基因隐藏状态。PPI网络是一种典型的图结构数据。对于图结构数据,节点的属性和网络的拓扑结构都具有重要意义。GCN是一种图表示学习模型,它可以在保留节点信息和网络拓扑结构的同时生成节点的低维密集嵌入。PRODeepSyn用GCN构造PPI网络中节点的嵌入作为基因隐藏状态。
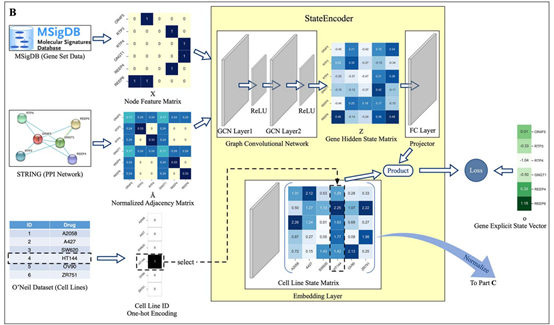
图2PRODeepSyn提取基因的隐藏状态矩阵
4.预测药物组合的协同作用分数
在构建药物特征和细胞系嵌入之后,本文设计了一个名为Predictor的深度神经网络用于预测药物组合在细胞系上的协同作用分数。Predictor接收两种药物的特性和一种细胞系的嵌入作为输入,并预测相应的协同得分。它有三个完全连接层,其中前两个全连接层使用ReLU激活功能,后面是批处理规范化层。作者将第二个全连接层的神经元数量设置为第一层的一半。最后一层全连接层只包含一个神经元,代表模型预测的协同得分。训练Predictor的损失函数是均方误差损失。
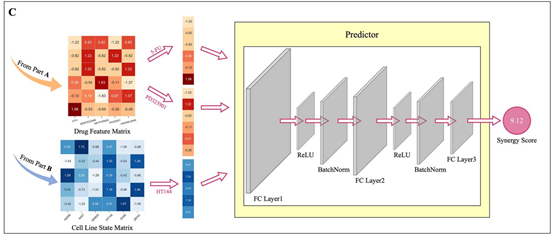
图3PRODeepSyn预测药物组合的协同作用分数
3.结果
1.方法比较
为了展示PRODeepSyn预测新药组合协同得分的能力,本文将此方法与其他两种DL方法与四种ML方法进行了比较。
表1 回归任务的方法比较结果
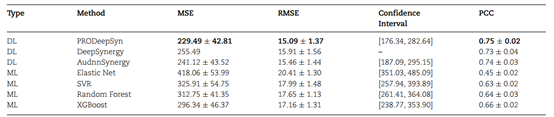
PRODeepSyn与其他方法在回归任务上的对比实验结果如表1所示,其在回归任务中实现了最低的MSE和RMSE以及最高的PCC。其MSE为229.49,比DeepSynergy低10.18%,比AuDNNSynergy低4.82%,比XGBoost低22.56%。这足以证明PRODeepSyn在回归任务上的优势。
表2 分类任务的方法比较结果
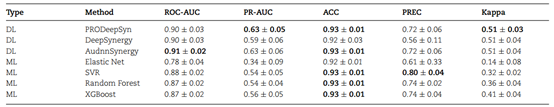
考虑到之前的很多研究将预测任务视为分类任务,本文进一步进行了相关实验,便于对比分析。参照其他文章的做法,本文只将协同得分大于30的样本视为正样本。各方法对分类任务的结果总结如表2所示。
值得注意的是,所有方法都具有几乎相同的ACC,而其他指标的值则有所不同,这是由于测试数据中负样本的比例很高。本文认为ROC-AUC和PR-AUC是相对公平的指标,而在不平衡数据集上,PR-AUC优于ROC-AUC。PRODeepSyn实现了最好的PR-AUC和相对较好的ROC-AUC。以此可知PRODeepSyn在分类任务上比其他方法具有综合优势。
2. 按组织汇总的预测
本文根据细胞系的组织类型将PRODeepSyn的预测结果可视化。在七种不同的组织类型中,大多数真实值和PRODeepSyn给出的预测协同作用分数都集中在[-50, 75]的范围内。对于负样本,PRODeepSyn的预测结果分布与真实值相似。对于正样本,PRODeepSyn倾向于给出更保守的预测结果。
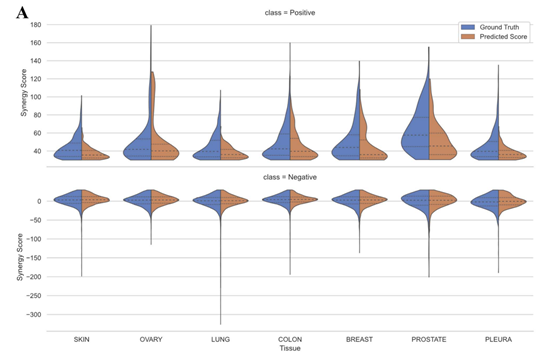
图4 基础真值的分布与组织汇总的预测协同得分之间的比较
大多数组织的预测是比较准确的。对于前列腺组织,PRODeepSyn预测的协同得分与实际情况之间的差异相对明显。作者认为这与数据集中属于该组织的细胞系数量少以及协同得分的分散分布有关。
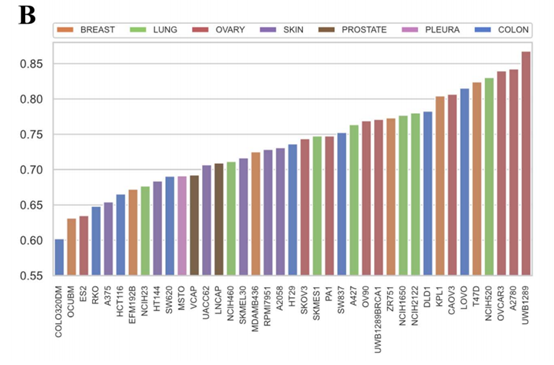
图5 每个细胞系的PCC值
条形图总结了每个细胞系的预测分数与真实值之间的PCC,条形图的颜色显示了细胞系所属的组织。其中,COLOR320DM的PCC最低,为0.60,UWB1289的PCC最高,为0.87。39个细胞系中,只有4个细胞系的PCC低于0.65,而16个细胞系的PCC高于0.75。
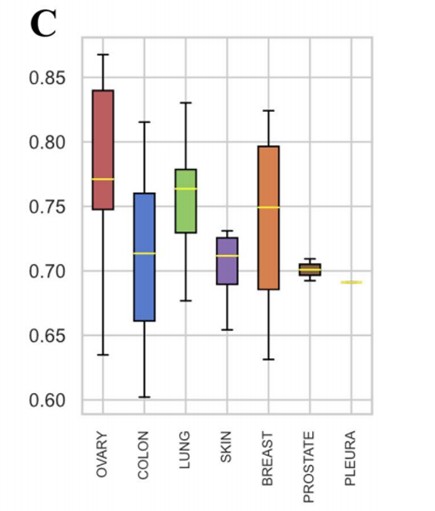
图6 按组织聚集的细胞系PCC值的箱线图
由箱线图可以看出,除胸膜外其余组织PCC中位数均大于0.70。对于属于卵巢的细胞系,PRODeepSyn的预测结果与真实值之间的相关性最强。总的来说,在不同的组织中,PRODeepSyn给出的预测结果与真实值之间存在很强的相关性,PCC与组织类型之间没有明显的相关性。PRODeepSyn在预测各种组织细胞系的抗癌协同药物组合方面具有潜在价值。
4.结论
本文介绍了一种发现抗癌协同药物的深度学习方法PRODeepSyn,该方法能够高效地得出组合药物对于癌症治疗效果的协同得分。在发现抗癌协同药物组合的过程中,相比其他先进方法,无论是回归任务还是分类任务PRODeepSyn都更具有综合优势。并且在不同组织中PRODeepSyn给出的预测结果与真实值之间都存在很强的相关性。PRODeepSyn 有望成为预筛选抗癌协同药物组合的强大工具。





 ดูบอลสด
Very well presented. Every quote was awesome and thanks for sharing the content. Keep sharing and keep motivating others.
ดูบอลสด
Very well presented. Every quote was awesome and thanks for sharing the content. Keep sharing and keep motivating others.



