今天为大家介绍的是来自Jennifer Listgarten团队的一篇概述论文。像ChatGPT和DALL-E2这样的模型可以根据文本提示生成文本和图像。尽管它们处理的数据类型和目标不同,但生成模型在蛋白质工程方面同样具有巨大的潜力。

自然进化通过随机过程(如突变和重组),结合有机体适应性的筛选机制,创造出新的蛋白质。在过去的几十年里,通过有向进化工程改造了蛋白质,这种方法大致模仿了自然进化,但使用了用户指定的筛选器来选择所需的性质。有向进化通常从已知的蛋白质出发,只探索相似的序列,通常通过使用随机方法进行序列修改。随着机器学习的最新进展,以及现代序列合成技术的进步,使用更复杂的生成机制来更有效地探索更广泛的蛋白质空间变得可行。各种机器学习建模策略可以用于涉及蛋白质的不同问题。在此,作者专注于生成模型——那些可以生成蛋白质序列和/或结构的模型——特别是条件生成模型,这些模型生成与指定性质(如蛋白质家族、活性位点结构(称为功能位点支架)或指定的主链结构(称为逆向折叠))一致的序列和/或结构。
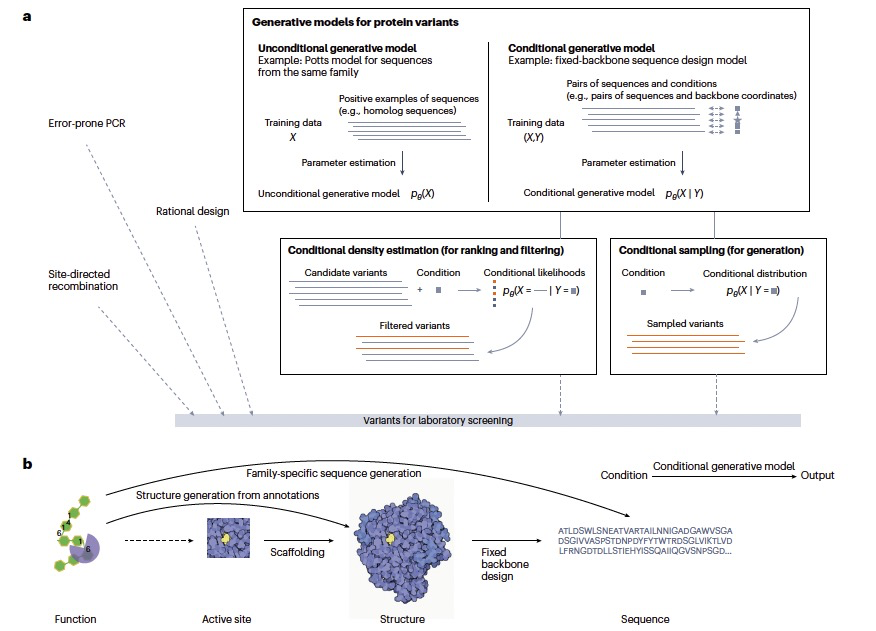
图 1
尽管条件生成模型用于蛋白质序列和结构相对较新,但它们与无条件生成模型紧密相关,如隐马尔可夫模型(HMMs)和Potts模型。无条件生成模型学习概率分布。从原理上讲,模型表示只有一个条件的分布。例如,一个家族的Potts模型学习该家族中蛋白质的氨基酸序列的分布。当有足够多的与感兴趣的蛋白质进化相关的同源体时,这样的无条件生成模型可以是生成多样蛋白质序列的一个简单而有成效的策略。与无条件模型相比,条件生成模型不仅学习一个条件的概率分布,而且学习许多条件的一系列概率分布,每个条件各一个(图1)。原则上,条件生成模型中的条件可以是任何性质,指定为一维标量、向量、矩阵等。这些条件可能包含或不包含几何信息,如3D结构。蛋白质的生成输出可以编码为序列、矩阵或图。为简单起见,作者的讨论基于生成氨基酸序列或以3D坐标形式的结构的模型,但讨论同样适用于其他类型的生成建模问题。
条件生成模型的训练和采样
在条件生成模型的训练和抽样过程中,机器学习模型需要在训练过程中学习参数。为了学习模型的参数,无论是神经网络还是传统的统计模型,我们必须定义一个损失函数,这个函数告诉我们使用特定参数组合会“损失”多少——也就是说,一个参数设置对于训练数据来说有多“不适当”。统计学中的一个经典目标函数是观察数据的似然性,当最大化时,产生参数的最大似然估计(MLE)。MLE的一个吸引人的方面是它的一致性:随着我们获得越来越多的训练数据,估计的参数产生的分布越来越接近真实分布——假设模型类包含了真实数据分布。在深度学习中,MLE有时等同于描述为最小化观察分布和预测分布之间的交叉熵——一种信息论概念,用来衡量两个分布之间的差异。在序列生成上下文中,MLE有时等同于描述为最小化困惑度——一个更具人类可解释性的量,指示每个位置的平均不确定性。为了最小化损失,我们从某些(通常是随机的)初始参数设置开始,然后使用数值优化技术(通常是梯度下降)迭代地精炼它们,直到参数根据预定义的停止标准收敛,例如当验证损失变化低于某个阈值时。一旦学习了模型参数,我们就可以使用模型来条件性地生成蛋白质序列或结构——也就是说,从模型中抽样产生具有高似然性的蛋白质,假设条件已经满足。大多数学习到的条件分布是非确定性的(即,随机的),意味着从一个条件会生成多种样本。这种随机性源于满足给定条件的输出可能有很多,以及从条件到输出的映射中的不确定性。根据模型,更有可能满足条件的序列(或结构)将有更高的条件似然性,并且更有可能被抽样。然而我们可能抽样到根本不满足条件的序列。
序列模型
针对蛋白质序列的条件生成模型可能基于蛋白质功能、蛋白质家族或主链结构来进行条件化。这些模型在首先使用生成模型生成蛋白质主链之后,十分依赖后续处理。在序列生成中,无论是条件还是无条件生成模型面临的一个技术挑战是理论上可能的序列数量巨大:对于长度为N的蛋白质,可以生成20^N种可能的氨基酸序列。因此,序列的生成模型基于将建模和抽样问题分解成更小、更易管理的部分的策略构建。一个常见的策略是分解条件概率分布——即,将可能性分解成较小单独的因素,其中每个因素本身是可管理的,并且所有因素可以通过将它们相乘来轻松组合。无条件模型的一个经典例子是用于序列对齐、分类和生成的隐藏马尔可夫模型(HMMs)。这些模型假设一个位置的氨基酸或核苷酸只依赖于前一个位置(或插入/删除),从而使其成为一个自回归模型。作为简单的自回归模型,HMMs产生的训练和抽样算法在序列长度上线性扩展。然而,许多问题需要在不同位置的氨基酸之间更复杂的依赖关系以进行准确建模。例如,通过将多个建模层组合在一起,如基于Transformer的自回归模型,可以捕捉这些更复杂的依赖关系。这些模型可以更好地捕捉序列中的长距离依赖关系。
自回归模型的一个限制是它们只能用于按一个特定选定的顺序生成序列。为了解决这个限制,可以明确地训练具有不同顺序的模型,所谓的“顺序不可知”模型,并学习更灵活的自回归模型,这些模型可以以任意顺序生成序列。条件分布的分解不必也通常不反映任何因果机制。然而,变量的不同分解或排序通常会导致不同的模型,每个模型可能或多或少有用。除了HMMs和更一般的自回归模型之外,Potts模型——也称为马尔可夫随机场——旨在显式捕捉序列中所有位置对的相互作用。然而,Potts模型的限制在于只能显式建模成对的相互作用,而不是更高阶的相互作用。变分自编码器,另一类无条件生成模型,原则上可以建模任意高阶的相互作用,也已被应用于蛋白质序列和结构的建模。虽然任何无条件序列生成模型原则上都可以适应作为条件生成模型,但序列生成一般倾向有自回归输出层的模型。例如,在GPT系列中的“下一个标记”语言模型也是自回归条件生成模型,其中条件是初始提示。在条件生成中,自回归模型在生成过程的每一步都基于输入进行条件化,根据条件和到目前为止已生成的序列部分调整预测。现代自回归条件生成模型通常采用神经网络架构来参数化概率分布。因此,其他一般机器学习设置中出现的超参数考虑因素在这里也同样出现。也就是说,用户必须考虑架构选择,如是否使用卷积和注意力层,使用多少层,以及每层的宽度。由于这些超参数因素取决于特定问题设置,没有通用解决方案。话虽如此,实践中,编码器-解码器架构经常用于自回归条件生成模型。编码器网络将输入条件y编码成“隐藏的”或潜在的表示,解码器网络将隐藏表示解码成条件序列分布。除了编码器-解码器架构之外,也使用了仅解码器架构进行条件生成,序列直接基于蛋白质家族的标签进行条件化。
虽然被称为“生成”模型,许多条件生成模型也可以用于评分和排名序列。为此,计算一组候选序列的条件似然性,为每个候选序列生成概率分数。这些条件似然性可以用来排名一个序列x满足给定条件y的可能性。这种评分和排名经常出现在零次蛋白质适应性预测和变异效应预测中。
结构模型
虽然蛋白质序列由20种氨基酸的有限“词汇”组成,蛋白质结构的三维坐标则是包含几何信息的实数值,因此它们引入了一组不同的建模挑战和解决方案。与自回归策略相比,结构生成的一个常见策略是将抽样分解成对所有坐标同时进行的迭代过程。采用这种迭代方法的扩散模型最近已经成为一类强大的模型,用于无条件和条件结构生成。扩散模型不是直接估计结构上的(条件)概率分布,而是估计这样一个分布在结构空间的局部邻域中的概率密度是如何变化的——也就是说,当结构的三维坐标被扰动时,似然性是如何增加或减少的。扩散模型学习概率分布的梯度而不是分布本身。概率分布可以从梯度信息中隐式恢复,但以这种方式学习分布似乎具有实际优势。在训练时,首先我们通过逐渐向训练数据中的蛋白质结构添加随机噪声来迭代地“扩散”蛋白质结构,直到它无法识别——也就是说,直到它由随机的三维坐标或无结构聚合物的三维坐标组成。然后我们训练一个神经网络学习如何逐步逆转这个扩散过程,以便我们可以从无法识别的结构或其任何中间状态恢复到原始的、结构完好的蛋白质结构。在某些情况下,逆向扩散模型是从头开始学习的,而在其他情况下,蛋白质折叠模型的进展被重新利用和微调以执行逆向扩散。在模型训练完成后,我们可以通过从一组随机的三维坐标开始,并迭代地应用学习到的逆向扩散模型,每次根据模型的隐式似然性逐步调整三维坐标,朝着更可能的坐标移动,来生成结构。对于一个训练良好的模型,逆向扩散应该会起始于随机坐标产生一个结构良好的蛋白质。虽然这个过程可能看起来类似于在分子动力学模拟中跟随一个力场,但扩散模型中的扩散过程并不一定具有物理意义。
使扩散模型能够成为条件扩散模型有两种主要策略。在“无分类器引导”的方法中,由神经网络描述的逆转扩散过程在开始时接受一个“条件标签”,使其能够相应地条件扩散过程。这样的模型必须直接用所需的用例条件类别进行训练,因为条件化被内置在神经网络中。相比之下,在“分类器引导”的条件化中,条件化不发生在神经网络中;相反,神经网络与一个外部分类器配对,该分类器已被单独训练用于分类,例如,一个三维结构是否可能满足感兴趣的条件;因此,扩散过程和条件化被解耦。分类器引导的条件化因此允许相同的无条件扩散模型轻松地被重新用于广泛的条件,无需重新训练。因此,分类器引导的方法有潜力适应广泛不同的输入条件模态。当然,条件生成的质量关键地取决于分类器的质量。无分类器和分类器引导的条件化方法都已应用于蛋白质结构生成任务,包括基于二级结构、粗略接触图、部分结构基序、对称性和生化属性的条件化。
编译|曾全晨
审稿|王建民
参考资料
Hsu, C., Fannjiang, C., & Listgarten, J. (2024). Generative models for protein structures and sequences. Nature Biotechnology, 42(2), 196-199.






 ดูบอลสด
Very well presented. Every quote was awesome and thanks for sharing the content. Keep sharing and keep motivating others.
ดูบอลสด
Very well presented. Every quote was awesome and thanks for sharing the content. Keep sharing and keep motivating others.



